+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | CryoEM Structure of Phenylalanine Ammonia Lyase from Planctomyces brasiliencis | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Phenylalanine catabolism Lyase / LYASE | |||||||||
| 機能・相同性 | histidine ammonia-lyase / histidine ammonia-lyase activity / Aromatic amino acid lyase / Aromatic amino acid lyase / Fumarase/histidase, N-terminal / L-Aspartase-like / Histidine ammonia-lyase 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  Rubinisphaera brasiliensis DSM 5305 (バクテリア) / Rubinisphaera brasiliensis DSM 5305 (バクテリア) /  Rubinisphaera brasiliensis (バクテリア) Rubinisphaera brasiliensis (バクテリア) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.17 Å | |||||||||
 データ登録者 データ登録者 | Duhoo Y / Buslov I / Desmons S | |||||||||
| 資金援助 |  スイス, 1件 スイス, 1件
| |||||||||
 引用 引用 |  ジャーナル: Angew Chem Int Ed Engl / 年: 2024 ジャーナル: Angew Chem Int Ed Engl / 年: 2024タイトル: Engineered Phenylalanine Ammonia-Lyases for the Enantioselective Synthesis of Aspartic Acid Derivatives. 著者: Ivan Buslov / Sarah Desmons / Yoan Duhoo / Xile Hu /  要旨: Biocatalytic hydroamination of alkenes is an efficient and selective method to synthesize natural and unnatural amino acids. Phenylalanine ammonia-lyases (PALs) have been previously engineered to ...Biocatalytic hydroamination of alkenes is an efficient and selective method to synthesize natural and unnatural amino acids. Phenylalanine ammonia-lyases (PALs) have been previously engineered to access a range of substituted phenylalanines and heteroarylalanines, but their substrate scope remains limited, typically including only arylacrylic acids. Moreover, the enantioselectivity in the hydroamination of electron-deficient substrates is often poor. Here, we report the structure-based engineering of PAL from Planctomyces brasiliensis (PbPAL), enabling preparative-scale enantioselective hydroaminations of previously inaccessible yet synthetically useful substrates, such as amide- and ester-containing fumaric acid derivatives. Through the elucidation of cryo-electron microscopy (cryo-EM) PbPAL structure and screening of the structure-based mutagenesis library, we identified the key active site residue L205 as pivotal for dramatically enhancing the enantioselectivity of hydroamination reactions involving electron-deficient substrates. Our engineered PALs demonstrated exclusive α-regioselectivity, high enantioselectivity, and broad substrate scope. The potential utility of the developed biocatalysts was further demonstrated by a preparative-scale hydroamination yielding tert-butyl protected l-aspartic acid, widely used as intermediate in peptide solid-phase synthesis. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_19897.map.gz emd_19897.map.gz | 86 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-19897-v30.xml emd-19897-v30.xml emd-19897.xml emd-19897.xml | 17 KB 17 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_19897_fsc.xml emd_19897_fsc.xml | 9.5 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_19897.png emd_19897.png | 65.2 KB | ||
| マスクデータ |  emd_19897_msk_1.map emd_19897_msk_1.map | 91.1 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-19897.cif.gz emd-19897.cif.gz | 6.5 KB | ||
| その他 |  emd_19897_half_map_1.map.gz emd_19897_half_map_1.map.gz emd_19897_half_map_2.map.gz emd_19897_half_map_2.map.gz | 84.5 MB 84.5 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-19897 http://ftp.pdbj.org/pub/emdb/structures/EMD-19897 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-19897 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-19897 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  9eq5MC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_19897.map.gz / 形式: CCP4 / 大きさ: 91.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_19897.map.gz / 形式: CCP4 / 大きさ: 91.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.8067 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_19897_msk_1.map emd_19897_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_19897_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_19897_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Phenylalanine ammonia-lyase protein in complex with (1R)-1-amino-...
| 全体 | 名称: Phenylalanine ammonia-lyase protein in complex with (1R)-1-amino-2-phenylethyl]phosphonic acid |
|---|---|
| 要素 |
|
-超分子 #1: Phenylalanine ammonia-lyase protein in complex with (1R)-1-amino-...
| 超分子 | 名称: Phenylalanine ammonia-lyase protein in complex with (1R)-1-amino-2-phenylethyl]phosphonic acid タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  Rubinisphaera brasiliensis DSM 5305 (バクテリア) Rubinisphaera brasiliensis DSM 5305 (バクテリア) |
-分子 #1: Histidine ammonia-lyase
| 分子 | 名称: Histidine ammonia-lyase / タイプ: protein_or_peptide / ID: 1 / 詳細: MDO: non standard Amino acid ALA-SER-GLY / コピー数: 4 / 光学異性体: LEVO / EC番号: histidine ammonia-lyase |
|---|---|
| 由来(天然) | 生物種:  Rubinisphaera brasiliensis (バクテリア) Rubinisphaera brasiliensis (バクテリア) |
| 分子量 | 理論値: 62.710332 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MLASSPSGHT NPVLSGAPLS INVVADIGRQ RLIPSLTDDE QVLNRVHACR DVVQKAVRNN ERIYGITTGF GGMSDIPIPP QHVAQTQDN LLAFLSTSTG ASLDPRHVRA AMALRANVLL QGRSGVRLEL IERLVEFLRQ DAIPVVCDLG SIG(MDO)DLV PL ...文字列: MLASSPSGHT NPVLSGAPLS INVVADIGRQ RLIPSLTDDE QVLNRVHACR DVVQKAVRNN ERIYGITTGF GGMSDIPIPP QHVAQTQDN LLAFLSTSTG ASLDPRHVRA AMALRANVLL QGRSGVRLEL IERLVEFLRQ DAIPVVCDLG SIG(MDO)DLV PL GVIARSIIGH PSTTQVKYQG EQADSHDVLQ QLNYSALQLE AKEGLALVNG TSFSSAIAAN CVFESQRLLS LSLVLQSI M VRALGGHPEA FHPFVDENKP HPGQGWSAQM MRDLLSYSPN DSKRNGDLAQ DRYSLRCLAQ YFAPIVEGIA QISQSISTE MNAVSDNPLI DVDTGRFHQS GNFLGQYVAM SMDQLRRHLG LLAKHLDVQI AQLVAPAFNN GLPASLRGNS SRPFNMGLKG LQITGNSIM PLLTYLGNPL TEHFPTHAEE FNQNINGLSW GSANLAWRSV QLFQHYLSVA SIFAVQAIDL RAGLEADHCD G RELLGETA TELYETVYDL LERNCGQESP FLFNDDEQSL EVDLQMLNGD LAGAGRMHEA VSSVTDSFLA EFCESGGGLE VL FQGPGGS SGSGHHHHHH HHH UniProtKB: Histidine ammonia-lyase |
-分子 #2: [(1R)-1-amino-2-phenylethyl]phosphonic acid
| 分子 | 名称: [(1R)-1-amino-2-phenylethyl]phosphonic acid / タイプ: ligand / ID: 2 / コピー数: 4 / 式: PPH |
|---|---|
| 分子量 | 理論値: 201.16 Da |
| Chemical component information | 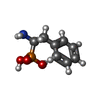 ChemComp-PPH: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1 mg/mL | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 8.8 構成要素:
| |||||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 90 sec. / 前処理 - 雰囲気: OTHER | |||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 95 % / チャンバー内温度: 283 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON IV (4k x 4k) 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 1.8 µm / 最小 デフォーカス(公称値): 0.8 µm / 倍率(公称値): 165000 |
| 試料ステージ | ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー






 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)