+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | AL amyloid fibril from the FOR103 light chain | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Amyloid fibril / AL amyloid / PROTEIN FIBRIL | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
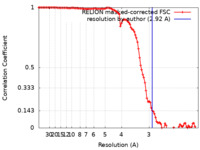
| 手法 | らせん対称体再構成法 / クライオ電子顕微鏡法 / 解像度: 2.92 Å | |||||||||
 データ登録者 データ登録者 | Pfeiffer PB / Karimi-Farsijani S / Kupfer N / Schmidt M / Faendrich M | |||||||||
| 資金援助 |  ドイツ, 1件 ドイツ, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2024 ジャーナル: Nat Commun / 年: 2024タイトル: Light chain mutations contribute to defining the fibril morphology in systemic AL amyloidosis. 著者: Sara Karimi-Farsijani / Peter Benedikt Pfeiffer / Sambhasan Banerjee / Julian Baur / Lukas Kuhn / Niklas Kupfer / Ute Hegenbart / Stefan O Schönland / Sebastian Wiese / Christian Haupt / ...著者: Sara Karimi-Farsijani / Peter Benedikt Pfeiffer / Sambhasan Banerjee / Julian Baur / Lukas Kuhn / Niklas Kupfer / Ute Hegenbart / Stefan O Schönland / Sebastian Wiese / Christian Haupt / Matthias Schmidt / Marcus Fändrich /  要旨: Systemic AL amyloidosis is one of the most frequently diagnosed forms of systemic amyloidosis. It arises from mutational changes in immunoglobulin light chains. To explore whether these mutations may ...Systemic AL amyloidosis is one of the most frequently diagnosed forms of systemic amyloidosis. It arises from mutational changes in immunoglobulin light chains. To explore whether these mutations may affect the structure of the formed fibrils, we determine and compare the fibril structures from several patients with cardiac AL amyloidosis. All patients are affected by light chains that contain an IGLV3-19 gene segment, and the deposited fibrils differ by the mutations within this common germ line background. Using cryo-electron microscopy, we here find different fibril structures in each patient. These data establish that the mutations of amyloidogenic light chains contribute to defining the fibril architecture and hence the structure of the pathogenic agent. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_19818.map.gz emd_19818.map.gz | 7.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-19818-v30.xml emd-19818-v30.xml emd-19818.xml emd-19818.xml | 15.7 KB 15.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_19818_fsc.xml emd_19818_fsc.xml | 10.6 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_19818.png emd_19818.png | 96 KB | ||
| マスクデータ |  emd_19818_msk_1.map emd_19818_msk_1.map | 103 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-19818.cif.gz emd-19818.cif.gz | 5.3 KB | ||
| その他 |  emd_19818_half_map_1.map.gz emd_19818_half_map_1.map.gz emd_19818_half_map_2.map.gz emd_19818_half_map_2.map.gz | 80.3 MB 80.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-19818 http://ftp.pdbj.org/pub/emdb/structures/EMD-19818 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-19818 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-19818 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_19818_validation.pdf.gz emd_19818_validation.pdf.gz | 712.4 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_19818_full_validation.pdf.gz emd_19818_full_validation.pdf.gz | 711.9 KB | 表示 | |
| XML形式データ |  emd_19818_validation.xml.gz emd_19818_validation.xml.gz | 17.9 KB | 表示 | |
| CIF形式データ |  emd_19818_validation.cif.gz emd_19818_validation.cif.gz | 23.6 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-19818 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-19818 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-19818 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-19818 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_19818.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_19818.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.04 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_19818_msk_1.map emd_19818_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_19818_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_19818_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : lambda 3 immunoglobulin light chain fragment
| 全体 | 名称: lambda 3 immunoglobulin light chain fragment |
|---|---|
| 要素 |
|
-超分子 #1: lambda 3 immunoglobulin light chain fragment
| 超分子 | 名称: lambda 3 immunoglobulin light chain fragment / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all / 詳細: residues 2-116 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: lambda 3 immunoglobulin light chain fragment, residues 2-116
| 分子 | 名称: lambda 3 immunoglobulin light chain fragment, residues 2-116 タイプ: protein_or_peptide / ID: 1 / コピー数: 6 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 12.202262 KDa |
| 配列 | 文字列: SELTQDPAVS VALGQTVRIT CQGDSLRSYY ASWYQQKSGQ APVLVIYSYN NRPSGIPDRF SGSNSGNTAS LTITGAQAED EADYYCNSR DSSGHHLVFG GGTKLTVLGQ PKAAPS |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | らせん対称体再構成法 |
| 試料の集合状態 | helical array |
- 試料調製
試料調製
| 緩衝液 | pH: 7 / 詳細: water |
|---|---|
| グリッド | モデル: C-flat-1.2/1.3 / 材質: COPPER / メッシュ: 400 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 40 sec. |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 95 % / 装置: LEICA PLUNGER |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 QUANTUM (4k x 4k) 検出モード: COUNTING / 撮影したグリッド数: 1 / 実像数: 2788 / 平均露光時間: 10.0 sec. / 平均電子線量: 53.7 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.2 µm / 倍率(公称値): 130000 |
| 試料ステージ | ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: BACKBONE TRACE / 当てはまり具合の基準: correlation coefficient |
|---|---|
| 得られたモデル |  PDB-9eme: |
 ムービー
ムービー コントローラー
コントローラー






 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)