+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Retron-Eco1 -1 turn mutant filament with ADP-ribosylated Effector (Consensus refinement) | |||||||||
 マップデータ マップデータ | Retron-Eco1 "-1 turn" mutant with ADPr. Consensus refinement. | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | retron / N-glycosidase / DNA-RNA-Protein complex / IMMUNE SYSTEM | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報RNA-directed DNA polymerase / RNA-directed DNA polymerase activity / defense response to virus / RNA binding / metal ion binding 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
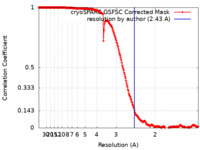
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.43 Å | |||||||||
 データ登録者 データ登録者 | Carabias del Rey A / Montoya G / Pape T | |||||||||
| 資金援助 |  デンマーク, 2件 デンマーク, 2件
| |||||||||
 引用 引用 |  ジャーナル: Mol Cell / 年: 2024 ジャーナル: Mol Cell / 年: 2024タイトル: Retron-Eco1 assembles NAD-hydrolyzing filaments that provide immunity against bacteriophages. 著者: Arturo Carabias / Sarah Camara-Wilpert / Mario Rodríguez Mestre / Blanca Lopéz-Méndez / Ivo A Hendriks / Ruiliang Zhao / Tillmann Pape / Anders Fuglsang / Sean Hoi-Ching Luk / Michael L ...著者: Arturo Carabias / Sarah Camara-Wilpert / Mario Rodríguez Mestre / Blanca Lopéz-Méndez / Ivo A Hendriks / Ruiliang Zhao / Tillmann Pape / Anders Fuglsang / Sean Hoi-Ching Luk / Michael L Nielsen / Rafael Pinilla-Redondo / Guillermo Montoya /  要旨: Retrons are toxin-antitoxin systems protecting bacteria against bacteriophages via abortive infection. The Retron-Eco1 antitoxin is formed by a reverse transcriptase (RT) and a non-coding RNA (ncRNA) ...Retrons are toxin-antitoxin systems protecting bacteria against bacteriophages via abortive infection. The Retron-Eco1 antitoxin is formed by a reverse transcriptase (RT) and a non-coding RNA (ncRNA)/multi-copy single-stranded DNA (msDNA) hybrid that neutralizes an uncharacterized toxic effector. Yet, the molecular mechanisms underlying phage defense remain unknown. Here, we show that the N-glycosidase effector, which belongs to the STIR superfamily, hydrolyzes NAD during infection. Cryoelectron microscopy (cryo-EM) analysis shows that the msDNA stabilizes a filament that cages the effector in a low-activity state in which ADPr, a NAD hydrolysis product, is covalently linked to the catalytic E106 residue. Mutations shortening the msDNA induce filament disassembly and the effector's toxicity, underscoring the msDNA role in immunity. Furthermore, we discovered a phage-encoded Retron-Eco1 inhibitor (U56) that binds ADPr, highlighting the intricate interplay between retron systems and phage evolution. Our work outlines the structural basis of Retron-Eco1 defense, uncovering ADPr's pivotal role in immunity. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_19792.map.gz emd_19792.map.gz | 1 GB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-19792-v30.xml emd-19792-v30.xml emd-19792.xml emd-19792.xml | 24 KB 24 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_19792_fsc.xml emd_19792_fsc.xml | 22.6 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_19792.png emd_19792.png | 82.9 KB | ||
| Filedesc metadata |  emd-19792.cif.gz emd-19792.cif.gz | 6.5 KB | ||
| その他 |  emd_19792_half_map_1.map.gz emd_19792_half_map_1.map.gz emd_19792_half_map_2.map.gz emd_19792_half_map_2.map.gz | 1.1 GB 1.1 GB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-19792 http://ftp.pdbj.org/pub/emdb/structures/EMD-19792 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-19792 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-19792 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_19792_validation.pdf.gz emd_19792_validation.pdf.gz | 1.1 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_19792_full_validation.pdf.gz emd_19792_full_validation.pdf.gz | 1.1 MB | 表示 | |
| XML形式データ |  emd_19792_validation.xml.gz emd_19792_validation.xml.gz | 31.1 KB | 表示 | |
| CIF形式データ |  emd_19792_validation.cif.gz emd_19792_validation.cif.gz | 40.5 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-19792 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-19792 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-19792 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-19792 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_19792.map.gz / 形式: CCP4 / 大きさ: 1.2 GB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_19792.map.gz / 形式: CCP4 / 大きさ: 1.2 GB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Retron-Eco1 "-1 turn" mutant with ADPr. Consensus refinement. | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.725 Å | ||||||||||||||||||||||||||||||||||||
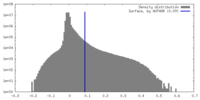
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: Retron-Eco1 "-1 turn" mutant with ADPr. Consensus refinement....
| ファイル | emd_19792_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Retron-Eco1 "-1 turn" mutant with ADPr. Consensus refinement. Half map B. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
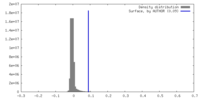
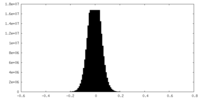
| 密度ヒストグラム |
-ハーフマップ: Retron-Eco1 "-1 turn" mutant with ADPr. Consensus refinement....
| ファイル | emd_19792_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Retron-Eco1 "-1 turn" mutant with ADPr. Consensus refinement. Half map A. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
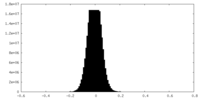
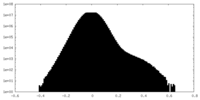
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Filamentous assembly of Retron-Eco1 "-1 turn" mutant in complex w...
| 全体 | 名称: Filamentous assembly of Retron-Eco1 "-1 turn" mutant in complex with ADPr, containing RT-msr-msDNA-Effector complex (Consensus refinement) |
|---|---|
| 要素 |
|
-超分子 #1: Filamentous assembly of Retron-Eco1 "-1 turn" mutant in complex w...
| 超分子 | 名称: Filamentous assembly of Retron-Eco1 "-1 turn" mutant in complex with ADPr, containing RT-msr-msDNA-Effector complex (Consensus refinement) タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#5 / 詳細: Filament formed by addition of NAD+ |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 21 kDa/nm |
-分子 #1: Retron-Eco1 Reverse Transcriptase
| 分子 | 名称: Retron-Eco1 Reverse Transcriptase / タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MKSAEYLNTF RLRNLGLPVM NNLHDMSKAT RISVETLRLL IYTADFRYRI YTVEKKGPEK RMRTIYQPSR ELKALQGWVL RNILDKLSSS PFSIGFEKHQ SILNNATPHI GANFILNIDL EDFFPSLTAN KVFGVFHSLG YNRLISSVLT KICCYKNLLP QGAPSSPKLA ...文字列: MKSAEYLNTF RLRNLGLPVM NNLHDMSKAT RISVETLRLL IYTADFRYRI YTVEKKGPEK RMRTIYQPSR ELKALQGWVL RNILDKLSSS PFSIGFEKHQ SILNNATPHI GANFILNIDL EDFFPSLTAN KVFGVFHSLG YNRLISSVLT KICCYKNLLP QGAPSSPKLA NLICSKLDYR IQGYAGSRGL IYTRYADDLT LSAQSMKKVV KARDFLFSII PSEGLVINSK KTCISGPRSQ RKVTGLVISQ EKVGIGREKY KEIRAKIHHI FCGKSSEIEH VRGWLSFILS VDSKSHRRLI TYISKLEKKY GKNPLNKAKT GSEFELENLY FQGELRRQAS ALEHHHHHH UniProtKB: Retron Ec86 reverse transcriptase |
-分子 #4: Retron-Eco1-Effector
| 分子 | 名称: Retron-Eco1-Effector / タイプ: protein_or_peptide / ID: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MNKKFTDEQQ QQLIGHLTKK GFYRGANIKI TIFLCGGDVA NHQSWRHQLS QFLAKFSDVD IFYPEDLFDD LLAGQGQHSL LSLENILAEA VDVIILFPES PGSFTELGAF SNNENLRRKL ICIQDAKFKS KRSFINYGPV RLLRKFNSKS VLRCSSNELK EMCDSSIDVA ...文字列: MNKKFTDEQQ QQLIGHLTKK GFYRGANIKI TIFLCGGDVA NHQSWRHQLS QFLAKFSDVD IFYPEDLFDD LLAGQGQHSL LSLENILAEA VDVIILFPES PGSFTELGAF SNNENLRRKL ICIQDAKFKS KRSFINYGPV RLLRKFNSKS VLRCSSNELK EMCDSSIDVA RKLRLYKKLM ASIKKVRKEN KVSKDIGNIL YAERFLLPCI YLLDSVNYRT LCELAFKAIK QDDVLSKIIV RSVVSRLINE RKILQMTDGY QVTALGASYV RSVFDRKTLD RLRLEIMNFE NRRKSTFNYD KIPYAHP UniProtKB: Retron Ec86 putative ribosyltransferase/DNA-binding protein |
-分子 #2: Retron-Eco1-msr
| 分子 | 名称: Retron-Eco1-msr / タイプ: rna / ID: 2 |
|---|---|
| 由来(天然) | 生物種:  |
| 配列 | 文字列: AUGCGCACCC UUAGCGAGAG GUUUAUCAUU AAGGUCAACC UCUGGAUGUU GUUUCGGCAU CCUGCAUUGA AUCUGAGUUA CU |
-分子 #3: Retron-Eco1-A2
| 分子 | 名称: Retron-Eco1-A2 / タイプ: rna / ID: 3 |
|---|---|
| 由来(天然) | 生物種:  |
| 配列 | 文字列: CGUAAGGGUG CGCA |
-分子 #5: Retron-Eco1-msDNA "-1 turn"
| 分子 | 名称: Retron-Eco1-msDNA "-1 turn" / タイプ: dna / ID: 5 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種:  |
| 配列 | 文字列: gtcagaaaaa acgggtttcc tggttggctc AGGCgttcca acaaggaaaa cagacagtaa ctcag |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | filament |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 構成要素:
詳細: 20 mM HEPES (pH 7.5),200 mM KCl, 5 mM MgCl2, 1mM TCEP | ||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 60 sec. / 前処理 - 雰囲気: AIR / 前処理 - 気圧: 0.03 kPa / 詳細: 10 mAmps | ||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV / 詳細: Blotting time 3 seconds. | ||||||||||
| 詳細 | The complex was prepared at a concentration corresponding to A260nm = ~12 |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 温度 | 最低: 100.0 K / 最高: 100.0 K |
| 特殊光学系 | エネルギーフィルター - 名称: TFS Selectris X / エネルギーフィルター - スリット幅: 5 eV 詳細: The slit was recentered every 6 hours during data collection |
| 撮影 | フィルム・検出器のモデル: TFS FALCON 4i (4k x 4k) 検出モード: COUNTING / デジタル化 - サイズ - 横: 4096 pixel / デジタル化 - サイズ - 縦: 4096 pixel / 撮影したグリッド数: 1 / 実像数: 26559 / 平均露光時間: 2.6 sec. / 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 0.4 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | PDB ID: Chain - Source name: PDB / Chain - Initial model type: experimental model |
|---|---|
| 詳細 | in ChimeraX |
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT |
 ムービー
ムービー コントローラー
コントローラー












 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)