+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of a 1033 Scaffold Base DNA Origami Nanostructure V4 with HPLC Purified Staples | ||||||||||||
 マップデータ マップデータ | |||||||||||||
 試料 試料 |
| ||||||||||||
 キーワード キーワード | DNA Origami / Multilayer / Square lattice / 1033 scaffold / DNA | ||||||||||||
| 生物種 | synthetic construct (人工物) | ||||||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 7.2 Å | ||||||||||||
 データ登録者 データ登録者 | Ali K / Georg K / Volodymyr M / Johanna G / Maximilian NH / Lukas K / Simone C / Hendrik D | ||||||||||||
| 資金援助 | European Union,  ドイツ, 3件 ドイツ, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Nano Lett / 年: 2024 ジャーナル: Nano Lett / 年: 2024タイトル: Designing Rigid DNA Origami Templates for Molecular Visualization Using Cryo-EM. 著者: Ali Khoshouei / Georg Kempf / Volodymyr Mykhailiuk / Johanna Mariko Griessing / Maximilian Nicolas Honemann / Lukas Kater / Simone Cavadini / Hendrik Dietz /   要旨: DNA origami, a method for constructing nanostructures from DNA, offers potential for diverse scientific and technological applications due to its ability to integrate various molecular ...DNA origami, a method for constructing nanostructures from DNA, offers potential for diverse scientific and technological applications due to its ability to integrate various molecular functionalities in a programmable manner. In this study, we examined the impact of internal crossover distribution and the compositional uniformity of staple strands on the structure of multilayer DNA origami using cryogenic electron microscopy (cryo-EM) single-particle analysis. A refined DNA object was utilized as an alignment framework in a host-guest model, where we successfully resolved an 8 kDa thrombin binding aptamer (TBA) linked to the host object. Our results broaden the spectrum of DNA in structural applications. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_19776.map.gz emd_19776.map.gz | 10.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-19776-v30.xml emd-19776-v30.xml emd-19776.xml emd-19776.xml | 13.7 KB 13.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_19776_fsc.xml emd_19776_fsc.xml | 14.3 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_19776.png emd_19776.png | 66.7 KB | ||
| Filedesc metadata |  emd-19776.cif.gz emd-19776.cif.gz | 4 KB | ||
| その他 |  emd_19776_half_map_1.map.gz emd_19776_half_map_1.map.gz emd_19776_half_map_2.map.gz emd_19776_half_map_2.map.gz | 193.8 MB 193.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-19776 http://ftp.pdbj.org/pub/emdb/structures/EMD-19776 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-19776 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-19776 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_19776_validation.pdf.gz emd_19776_validation.pdf.gz | 835.5 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_19776_full_validation.pdf.gz emd_19776_full_validation.pdf.gz | 835.1 KB | 表示 | |
| XML形式データ |  emd_19776_validation.xml.gz emd_19776_validation.xml.gz | 22.1 KB | 表示 | |
| CIF形式データ |  emd_19776_validation.cif.gz emd_19776_validation.cif.gz | 28.8 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-19776 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-19776 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-19776 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-19776 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_19776.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_19776.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.86 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #2
| ファイル | emd_19776_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
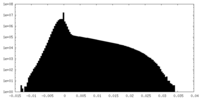
| 投影像・断面図 |
| ||||||||||||
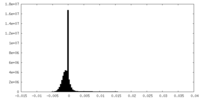
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_19776_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
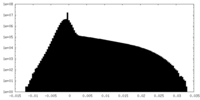
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : High-Resolution Structure of a 1033 Scaffold Base DNA Origami Nan...
| 全体 | 名称: High-Resolution Structure of a 1033 Scaffold Base DNA Origami Nanostructure V4 with HPLC Purified Staples |
|---|---|
| 要素 |
|
-超分子 #1: High-Resolution Structure of a 1033 Scaffold Base DNA Origami Nan...
| 超分子 | 名称: High-Resolution Structure of a 1033 Scaffold Base DNA Origami Nanostructure V4 with HPLC Purified Staples タイプ: complex / ID: 1 / 親要素: 0 |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 |
|---|---|
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 検出モード: COUNTING / 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 0.01 mm / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 0.5 µm / 倍率(公称値): 75000 |
| 試料ステージ | ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー













 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)