+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | GroEL with bound GroTAC peptide | |||||||||
 マップデータ マップデータ | GroEL with bound GroTAC peptide EM map | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | GroEL / GroTAC / PROTAC / degrader / complex / CHAPERONE | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報GroEL-GroES complex / chaperonin ATPase / virion assembly / : / isomerase activity / ATP-dependent protein folding chaperone / response to radiation / unfolded protein binding / protein folding / response to heat ...GroEL-GroES complex / chaperonin ATPase / virion assembly / : / isomerase activity / ATP-dependent protein folding chaperone / response to radiation / unfolded protein binding / protein folding / response to heat / protein refolding / magnesium ion binding / ATP hydrolysis activity / ATP binding / identical protein binding / membrane / cytosol 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
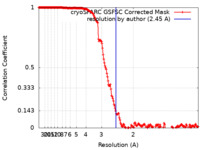
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.45 Å | |||||||||
 データ登録者 データ登録者 | Wroblewski K / Izert-Nowakowska MA / Goral TK / Klimecka MM / Kmiecik S / Gorna MW | |||||||||
| 資金援助 |  ポーランド, 2件 ポーランド, 2件
| |||||||||
 引用 引用 |  ジャーナル: EMBO Rep / 年: 2025 ジャーナル: EMBO Rep / 年: 2025タイトル: Targeted protein degradation in Escherichia coli using CLIPPERs. 著者: Matylda Anna Izert-Nowakowska / Maria Magdalena Klimecka / Anna Antosiewicz / Karol Wróblewski / Jakub Józef Kowalski / Katarzyna Justyna Bandyra / Tomasz Góral / Sebastian Kmiecik / ...著者: Matylda Anna Izert-Nowakowska / Maria Magdalena Klimecka / Anna Antosiewicz / Karol Wróblewski / Jakub Józef Kowalski / Katarzyna Justyna Bandyra / Tomasz Góral / Sebastian Kmiecik / Remigiusz Adam Serwa / Maria Wiktoria Górna /  要旨: New, universal tools for targeted protein degradation in bacteria can help to accelerate protein function studies and antimicrobial research. We describe a new method for degrading bacterial proteins ...New, universal tools for targeted protein degradation in bacteria can help to accelerate protein function studies and antimicrobial research. We describe a new method for degrading bacterial proteins using plasmid-encoded degrader peptides which deliver target proteins for degradation by a highly conserved ClpXP protease. We demonstrate the mode of action of the degraders on a challenging essential target, GroEL. The studies in bacteria are complemented by in vitro binding and structural studies. Expression of degrader peptides results in a temperature-dependent growth inhibition and depletion of GroEL levels over time. The reduction of GroEL levels is accompanied by dramatic proteome alterations. The presented method offers a new alternative approach for regulating protein levels in bacteria without genomic modifications or tag fusions. Our studies demonstrate that ClpXP is an attractive protease for the future use in bacterial-targeted protein degradation. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_19687.map.gz emd_19687.map.gz | 566.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-19687-v30.xml emd-19687-v30.xml emd-19687.xml emd-19687.xml | 29.5 KB 29.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_19687_fsc.xml emd_19687_fsc.xml | 17.8 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_19687.png emd_19687.png | 139.7 KB | ||
| マスクデータ |  emd_19687_msk_1.map emd_19687_msk_1.map | 600.7 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-19687.cif.gz emd-19687.cif.gz | 7.8 KB | ||
| その他 |  emd_19687_additional_1.map.gz emd_19687_additional_1.map.gz emd_19687_half_map_1.map.gz emd_19687_half_map_1.map.gz emd_19687_half_map_2.map.gz emd_19687_half_map_2.map.gz | 290.6 MB 556.6 MB 556.6 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-19687 http://ftp.pdbj.org/pub/emdb/structures/EMD-19687 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-19687 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-19687 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_19687_validation.pdf.gz emd_19687_validation.pdf.gz | 1.1 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_19687_full_validation.pdf.gz emd_19687_full_validation.pdf.gz | 1.1 MB | 表示 | |
| XML形式データ |  emd_19687_validation.xml.gz emd_19687_validation.xml.gz | 27.6 KB | 表示 | |
| CIF形式データ |  emd_19687_validation.cif.gz emd_19687_validation.cif.gz | 36.3 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-19687 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-19687 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-19687 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-19687 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8s32MC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_19687.map.gz / 形式: CCP4 / 大きさ: 600.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_19687.map.gz / 形式: CCP4 / 大きさ: 600.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | GroEL with bound GroTAC peptide EM map | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.5878 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_19687_msk_1.map emd_19687_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: Unsharpened map
| ファイル | emd_19687_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Unsharpened map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: GroEL with bound GroTAC peptide EM half map B
| ファイル | emd_19687_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | GroEL with bound GroTAC peptide EM half map B | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: GroEL with bound GroTAC peptide EM half map A
| ファイル | emd_19687_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | GroEL with bound GroTAC peptide EM half map A | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : A complex of GroEL with GroTAC peptide
| 全体 | 名称: A complex of GroEL with GroTAC peptide |
|---|---|
| 要素 |
|
-超分子 #1: A complex of GroEL with GroTAC peptide
| 超分子 | 名称: A complex of GroEL with GroTAC peptide / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 4 kDa/nm |
-超分子 #2: GroEL
| 超分子 | 名称: GroEL / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  |
-超分子 #3: GroTAC
| 超分子 | 名称: GroTAC / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #2 |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
-分子 #1: Chaperonin GroEL
| 分子 | 名称: Chaperonin GroEL / タイプ: protein_or_peptide / ID: 1 / コピー数: 14 / 光学異性体: LEVO / EC番号: chaperonin ATPase |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 57.391711 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MAAKDVKFGN DARVKMLRGV NVLADAVKVT LGPKGRNVVL DKSFGAPTIT KDGVSVAREI ELEDKFENMG AQMVKEVASK ANDAAGDGT TTATVLAQAI ITEGLKAVAA GMNPMDLKRG IDKAVTAAVE ELKALSVPCS DSKAIAQVGT ISANSDETVG K LIAEAMDK ...文字列: MAAKDVKFGN DARVKMLRGV NVLADAVKVT LGPKGRNVVL DKSFGAPTIT KDGVSVAREI ELEDKFENMG AQMVKEVASK ANDAAGDGT TTATVLAQAI ITEGLKAVAA GMNPMDLKRG IDKAVTAAVE ELKALSVPCS DSKAIAQVGT ISANSDETVG K LIAEAMDK VGKEGVITVE DGTGLQDELD VVEGMQFDRG YLSPYFINKP ETGAVELESP FILLADKKIS NIREMLPVLE AV AKAGKPL LIIAEDVEGE ALATLVVNTM RGIVKVAAVK APGFGDRRKA MLQDIATLTG GTVISEEIGM ELEKATLEDL GQA KRVVIN KDTTTIIDGV GEEAAIQGRV AQIRQQIEEA TSDYDREKLQ ERVAKLAGGV AVIKVGAATE VEMKEKKARV EDAL HATRA AVEEGVVAGG GVALIRVASK LADLRGQNED QNVGIKVALR AMEAPLRQIV LNCGEEPSVV ANTVKGGDGN YGYNA ATEE YGNMIDMGIL DPTKVTRSAL QYAASVAGLM ITTECMVTDL PKNDAADLGA AGGMGGMGGM GGMM UniProtKB: Chaperonin GroEL |
-分子 #2: GroTAC
| 分子 | 名称: GroTAC / タイプ: protein_or_peptide / ID: 2 / コピー数: 14 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 3.176783 KDa |
| 配列 | 文字列: CYRGGRPALR VVK(UNK)(UNK)(UNK)SWMT TPWGFHLP |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 4 mg/mL | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 8 構成要素:
詳細: ATP was added to the sample immediately before disposing on a grid | |||||||||||||||
| グリッド | モデル: Quantifoil R2/2 / 材質: COPPER / メッシュ: 200 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 60 sec. / 前処理 - 雰囲気: AIR / 前処理 - 気圧: 0.038 kPa | |||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS GLACIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 検出モード: COUNTING / デジタル化 - サイズ - 横: 4096 pixel / デジタル化 - サイズ - 縦: 4096 pixel / 撮影したグリッド数: 1 / 実像数: 4672 / 平均露光時間: 16.67 sec. / 平均電子線量: 40.0 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.2 µm / 最小 デフォーカス(公称値): 0.8 µm / 倍率(公称値): 240000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | Chain - Source name: Other / Chain - Initial model type: in silico model 詳細: The CA model was obtained by ModelAngelo (1.09) and then subsequently rebuilt to full-atom by cg2all (1.5). |
|---|---|
| 詳細 | The C-alpha model was obtained by two rounds of ab initio modeling using machine learning-based ModelAngelo 1.09 software. The incorrectly assigned GroEL residues were then manually corrected. The C-alpha trace of the GroTAC peptide was initially constructed by ModelAngelo, with residue identities suggested through a comparison to the 1MNF PDB structure. The reconstruction to full-atom was performed using cg2all 1.5 software. The first stage of refinement was done in Coot (0.9.8.5). The final refinement stage involved two rounds of phenix.real_space_refine (Phenix 1.21.5207). |
| 精密化 | 空間: REAL / プロトコル: AB INITIO MODEL / 当てはまり具合の基準: 0.88 |
| 得られたモデル |  PDB-8s32: |
 ムービー
ムービー コントローラー
コントローラー





 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)