+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Human UPF1 RNA helicase with AMPPNP and RNA | |||||||||
 マップデータ マップデータ | Human UPF1 bound to AMPPNP and RNA | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | RNA helicase / human / nonsense-mediated mRNA decay / RNA BINDING PROTEIN | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
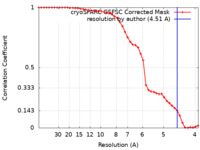
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.51 Å | |||||||||
 データ登録者 データ登録者 | Langer LM / Conti E | |||||||||
| 資金援助 |  ドイツ, 1件 ドイツ, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nucleic Acids Res / 年: 2024 ジャーナル: Nucleic Acids Res / 年: 2024タイトル: UPF1 helicase orchestrates mutually exclusive interactions with the SMG6 endonuclease and UPF2. 著者: Lukas M Langer / Katharina Kurscheidt / Jérôme Basquin / Fabien Bonneau / Iuliia Iermak / Claire Basquin / Elena Conti /  要旨: Nonsense-mediated mRNA decay (NMD) is a conserved co-translational mRNA surveillance and turnover pathway across eukaryotes. NMD has a central role in degrading defective mRNAs and also regulates the ...Nonsense-mediated mRNA decay (NMD) is a conserved co-translational mRNA surveillance and turnover pathway across eukaryotes. NMD has a central role in degrading defective mRNAs and also regulates the stability of a significant portion of the transcriptome. The pathway is organized around UPF1, an RNA helicase that can interact with several NMD-specific factors. In human cells, degradation of the targeted mRNAs begins with a cleavage event that requires the recruitment of the SMG6 endonuclease to UPF1. Previous studies have identified functional links between SMG6 and UPF1, but the underlying molecular mechanisms have remained elusive. Here, we used mass spectrometry, structural biology and biochemical approaches to identify and characterize a conserved short linear motif in SMG6 that interacts with the cysteine/histidine-rich (CH) domain of UPF1. Unexpectedly, we found that the UPF1-SMG6 interaction is precluded when the UPF1 CH domain is engaged with another NMD factor, UPF2. Based on cryo-EM data, we propose that the formation of distinct SMG6-containing and UPF2-containing NMD complexes may be dictated by different conformational states connected to the RNA-binding status of UPF1. Our findings rationalize a key event in metazoan NMD and advance our understanding of mechanisms regulating activity and guiding substrate recognition by the SMG6 endonuclease. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_19451.map.gz emd_19451.map.gz | 7.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-19451-v30.xml emd-19451-v30.xml emd-19451.xml emd-19451.xml | 14 KB 14 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_19451_fsc.xml emd_19451_fsc.xml | 4.2 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_19451.png emd_19451.png | 100.7 KB | ||
| Filedesc metadata |  emd-19451.cif.gz emd-19451.cif.gz | 5.2 KB | ||
| その他 |  emd_19451_half_map_1.map.gz emd_19451_half_map_1.map.gz emd_19451_half_map_2.map.gz emd_19451_half_map_2.map.gz | 7.4 MB 7.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-19451 http://ftp.pdbj.org/pub/emdb/structures/EMD-19451 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-19451 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-19451 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_19451_validation.pdf.gz emd_19451_validation.pdf.gz | 695.5 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_19451_full_validation.pdf.gz emd_19451_full_validation.pdf.gz | 695 KB | 表示 | |
| XML形式データ |  emd_19451_validation.xml.gz emd_19451_validation.xml.gz | 10.9 KB | 表示 | |
| CIF形式データ |  emd_19451_validation.cif.gz emd_19451_validation.cif.gz | 13.7 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-19451 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-19451 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-19451 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-19451 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_19451.map.gz / 形式: CCP4 / 大きさ: 8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_19451.map.gz / 形式: CCP4 / 大きさ: 8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Human UPF1 bound to AMPPNP and RNA | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.885 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #1
| ファイル | emd_19451_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
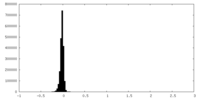
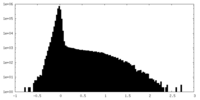
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_19451_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
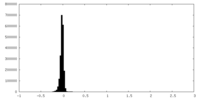
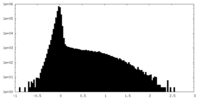
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : UPF1 bound to AMPPNP
| 全体 | 名称: UPF1 bound to AMPPNP |
|---|---|
| 要素 |
|
-超分子 #1: UPF1 bound to AMPPNP
| 超分子 | 名称: UPF1 bound to AMPPNP / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 124 KDa |
-分子 #1: UPF1, RENT1
| 分子 | 名称: UPF1, RENT1 / タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MSVEAYGPSS QTLTFLDTEE AELLGADTQG SEFEFTDFTL PSQTQTPPGG PGGPGGGGAG GPGGAGAGAA AGQLDAQVGP EGILQNGAVD DSVAKTSQLL AELNFEEDEE DTYYTKDLPI HACSYCGIHD PACVVYCNTS KKWFCNGRGN TSGSHIVNHL VRAKCKEVTL ...文字列: MSVEAYGPSS QTLTFLDTEE AELLGADTQG SEFEFTDFTL PSQTQTPPGG PGGPGGGGAG GPGGAGAGAA AGQLDAQVGP EGILQNGAVD DSVAKTSQLL AELNFEEDEE DTYYTKDLPI HACSYCGIHD PACVVYCNTS KKWFCNGRGN TSGSHIVNHL VRAKCKEVTL HKDGPLGETV LECYNCGCRN VFLLGFIPAK ADSVVVLLCR QPCASQSSLK DINWDSSQWQ PLIQDRCFLS WLVKIPSEQE QLRARQITAQ QINKLEELWK ENPSATLEDL EKPGVDEEPQ HVLLRYEDAY QYQNIFGPLV KLEADYDKKL KESQTQDNIT VRWDLGLNKK RIAYFTLPKT DSDMRLMQGD EICLRYKGDL APLWKGIGHV IKVPDNYGDE IAIELRSSVG APVEVTHNFQ VDFVWKSTSF DRMQSALKTF AVDETSVSGY IYHKLLGHEV EDVIIKCQLP KRFTAQGLPD LNHSQVYAVK TVLQRPLSLI QGPPGTGKTV TSATIVYHLA RQGNGPVLVC APSNIAVDQL TEKIHQTGLK VVRLCAKSRE AIDSPVSFLA LHNQIRNMDS MPELQKLQQL KDETGELSSA DEKRYRALKR TAERELLMNA DVICCTCVGA GDPRLAKMQF RSILIDESTQ ATEPECMVPV VLGAKQLILV GDHCQLGPVV MCKKAAKAGL SQSLFERLVV LGIRPIRLQV QYRMHPALSA FPSNIFYEGS LQNGVTAADR VKKGFDFQWP QPDKPMFFYV TQGQEEIASS GTSYLNRTEA ANVEKITTKL LKAGAKPDQI GIITPYEGQR SYLVQYMQFS GSLHTKLYQE VEIASVDAFQ GREKDFIILS CVRANEHQGI GFLNDPRRLN VALTRARYGV IIVGNPKALS KQPLWNHLLN YYKEQKVLVE GPLNNLRESL MQFSKPRKLV NTINPGARFM TTAMYDAREA IIPGSVYDRS SQGRPSSMYF QTHDQIGMIS AGPSHVAAMN IPIPFNLVMP PMPPPGYFGQ ANGPAAGRGT PKGKTGRGGR QKNRFGLPGP SQTNLPNSQA SQDVASQPFS QGALTQGYIS MSQPSQMSQP GLSQPELSQD SYLGDEFKSQ IDVALSQDST YQGERAYQHG GVTGLSQY |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 |
|---|---|
| 凍結 | 凍結剤: ETHANE-PROPANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS GLACIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / 平均電子線量: 60.0 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 1.5 µm / 倍率(公称値): 22000 |
| 試料ステージ | ホルダー冷却材: NITROGEN |
 ムービー
ムービー コントローラー
コントローラー






 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)