+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | TadA/CpaF with ADP | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | pilus / secretion / ATPase / MOTOR PROTEIN | |||||||||
| 機能・相同性 | : / Type II/IV secretion system protein / Type II/IV secretion system protein / ATP hydrolysis activity / P-loop containing nucleoside triphosphate hydrolase / Pilus assembly ATPase CpaF 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  Caulobacter vibrioides NA1000 (バクテリア) Caulobacter vibrioides NA1000 (バクテリア) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.1 Å | |||||||||
 データ登録者 データ登録者 | Hohl M / Low H | |||||||||
| 資金援助 |  英国, 1件 英国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2024 ジャーナル: Nat Commun / 年: 2024タイトル: Bidirectional pilus processing in the Tad pilus system motor CpaF. 著者: Michael Hohl / Emma J Banks / Max P Manley / Tung B K Le / Harry H Low /  要旨: The bacterial tight adherence pilus system (TadPS) assembles surface pili essential for adhesion and colonisation in many human pathogens. Pilus dynamics are powered by the ATPase CpaF (TadA), which ...The bacterial tight adherence pilus system (TadPS) assembles surface pili essential for adhesion and colonisation in many human pathogens. Pilus dynamics are powered by the ATPase CpaF (TadA), which drives extension and retraction cycles in Caulobacter crescentus through an unknown mechanism. Here we use cryogenic electron microscopy and cell-based light microscopy to characterise CpaF mechanism. We show that CpaF assembles into a hexamer with C2 symmetry in different nucleotide states. Nucleotide cycling occurs through an intra-subunit clamp-like mechanism that promotes sequential conformational changes between subunits. Moreover, a comparison of the active sites with different nucleotides bound suggests a mechanism for bidirectional motion. Conserved CpaF residues, predicted to interact with platform proteins CpaG (TadB) and CpaH (TadC), are mutated in vivo to establish their role in pilus processing. Our findings provide a model for how CpaF drives TadPS pilus dynamics and have broad implications for how other ancient type 4 filament family members power pilus assembly. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_19209.map.gz emd_19209.map.gz | 161.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-19209-v30.xml emd-19209-v30.xml emd-19209.xml emd-19209.xml | 14.1 KB 14.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_19209.png emd_19209.png | 173 KB | ||
| Filedesc metadata |  emd-19209.cif.gz emd-19209.cif.gz | 5.6 KB | ||
| その他 |  emd_19209_half_map_1.map.gz emd_19209_half_map_1.map.gz emd_19209_half_map_2.map.gz emd_19209_half_map_2.map.gz | 139.3 MB 138.9 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-19209 http://ftp.pdbj.org/pub/emdb/structures/EMD-19209 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-19209 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-19209 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_19209_validation.pdf.gz emd_19209_validation.pdf.gz | 926.7 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_19209_full_validation.pdf.gz emd_19209_full_validation.pdf.gz | 926.2 KB | 表示 | |
| XML形式データ |  emd_19209_validation.xml.gz emd_19209_validation.xml.gz | 15.2 KB | 表示 | |
| CIF形式データ |  emd_19209_validation.cif.gz emd_19209_validation.cif.gz | 17.7 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-19209 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-19209 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-19209 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-19209 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_19209.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_19209.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.829 Å | ||||||||||||||||||||||||||||||||||||
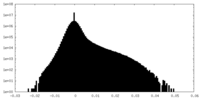
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #2
| ファイル | emd_19209_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
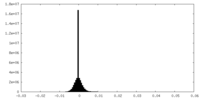
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_19209_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
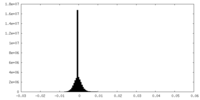
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Hexameric complex of TadA bound to ADP
| 全体 | 名称: Hexameric complex of TadA bound to ADP |
|---|---|
| 要素 |
|
-超分子 #1: Hexameric complex of TadA bound to ADP
| 超分子 | 名称: Hexameric complex of TadA bound to ADP / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  Caulobacter vibrioides NA1000 (バクテリア) Caulobacter vibrioides NA1000 (バクテリア) |
-分子 #1: Pilus assembly ATPase CpaF
| 分子 | 名称: Pilus assembly ATPase CpaF / タイプ: protein_or_peptide / ID: 1 / コピー数: 6 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Caulobacter vibrioides NA1000 (バクテリア) Caulobacter vibrioides NA1000 (バクテリア) |
| 分子量 | 理論値: 46.578355 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: DYYHATKTTI FNALLNTIDL SQLAQLDLKQ AGEEIRDIVA ELVAIKNVSM SVAEQEHLVQ DIINDVLGYG PLEPLLARDD IADIMVNGA HRVFIEVGGK VQLTNVRFRD NLQLMNICQR IVSQVGRRVD ESSPICDARL PDGSRVNVIA PPLALDGPTL T IRKFKKDK ...文字列: DYYHATKTTI FNALLNTIDL SQLAQLDLKQ AGEEIRDIVA ELVAIKNVSM SVAEQEHLVQ DIINDVLGYG PLEPLLARDD IADIMVNGA HRVFIEVGGK VQLTNVRFRD NLQLMNICQR IVSQVGRRVD ESSPICDARL PDGSRVNVIA PPLALDGPTL T IRKFKKDK LTMKNLVEFA SISPEGARVL GVIGACRCNL VISGGTGSGK TTLLNTMTAF IDPTERVVTC EDAAELQLQQ PH VVRLETR PPNLEGSGAV TMRDLVKNCL RMRPERIIVG EVRGPEAFDL LQAMNTGHDG SMGTLHANSP REAISRIESM ITM GGYGLP SKTIKEMIVG SVDVIIQAAR LRDGSRRITH ITEVVGLEGD VIVTQDLFVY EITGEDEHGK VVGKHRSTGI ARPR FWDRA RYYGLERELA EALDAAEAL UniProtKB: Pilus assembly ATPase CpaF |
-分子 #2: ADENOSINE-5'-DIPHOSPHATE
| 分子 | 名称: ADENOSINE-5'-DIPHOSPHATE / タイプ: ligand / ID: 2 / コピー数: 6 / 式: ADP |
|---|---|
| 分子量 | 理論値: 427.201 Da |
| Chemical component information |  ChemComp-ADP: |
-分子 #3: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 3 / コピー数: 6 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-分子 #4: 4-(2-HYDROXYETHYL)-1-PIPERAZINE ETHANESULFONIC ACID
| 分子 | 名称: 4-(2-HYDROXYETHYL)-1-PIPERAZINE ETHANESULFONIC ACID / タイプ: ligand / ID: 4 / コピー数: 4 / 式: EPE |
|---|---|
| 分子量 | 理論値: 238.305 Da |
| Chemical component information |  ChemComp-EPE: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 平均電子線量: 40.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 1.0 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: INSILICO MODEL |
|---|---|
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 3.1 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 114641 |
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
 ムービー
ムービー コントローラー
コントローラー










 X (Sec.)
X (Sec.) Y (Row.)
Y (Row.) Z (Col.)
Z (Col.)