+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | A DNA Robotic Switch with Regulated Autonomous Display of Cytotoxic Ligand Nanopatterns | |||||||||||||||||||||
 マップデータ マップデータ | From cryoSPARC 3DFlex reconstruct job | |||||||||||||||||||||
 試料 試料 |
| |||||||||||||||||||||
 キーワード キーワード | DNA / DNA origami / Lgand nanopattern / pH sensitive switch / Solid tumor / Death receptor clustering | |||||||||||||||||||||
| 生物種 | synthetic construct (人工物) /  Phage M13mp18 (ファージ) Phage M13mp18 (ファージ) | |||||||||||||||||||||
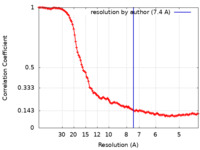
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 7.4 Å | |||||||||||||||||||||
 データ登録者 データ登録者 | Wang Y / Berzina I / Hogberg B | |||||||||||||||||||||
| 資金援助 |  スウェーデン, European Union, スウェーデン, European Union,  フィンランド, 6件 フィンランド, 6件
| |||||||||||||||||||||
 引用 引用 |  ジャーナル: Nat Nanotechnol / 年: 2024 ジャーナル: Nat Nanotechnol / 年: 2024タイトル: A DNA robotic switch with regulated autonomous display of cytotoxic ligand nanopatterns. 著者: Yang Wang / Igor Baars / Ieva Berzina / Iris Rocamonde-Lago / Boxuan Shen / Yunshi Yang / Marco Lolaico / Janine Waldvogel / Ioanna Smyrlaki / Keying Zhu / Robert A Harris / Björn Högberg /   要旨: The clustering of death receptors (DRs) at the membrane leads to apoptosis. With the goal of treating tumours, multivalent molecular tools that initiate this mechanism have been developed. However, ...The clustering of death receptors (DRs) at the membrane leads to apoptosis. With the goal of treating tumours, multivalent molecular tools that initiate this mechanism have been developed. However, DRs are also ubiquitously expressed in healthy tissue. Here we present a stimuli-responsive robotic switch nanodevice that can autonomously and selectively turn on the display of cytotoxic ligand patterns in tumour microenvironments. We demonstrate a switchable DNA origami that normally hides six ligands but displays them as a hexagonal pattern 10 nm in diameter once under higher acidity. This can effectively cluster DRs and trigger apoptosis of human breast cancer cells at pH 6.5 while remaining inert at pH 7.4. When administered to mice bearing human breast cancer xenografts, this nanodevice decreased tumour growth by up to 70%. The data demonstrate the feasibility and opportunities for developing ligand pattern switches as a path for targeted treatment. | |||||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_19129.map.gz emd_19129.map.gz | 117.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-19129-v30.xml emd-19129-v30.xml emd-19129.xml emd-19129.xml | 17.1 KB 17.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_19129_fsc.xml emd_19129_fsc.xml | 14.6 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_19129.png emd_19129.png | 62 KB | ||
| Filedesc metadata |  emd-19129.cif.gz emd-19129.cif.gz | 4.5 KB | ||
| その他 |  emd_19129_half_map_1.map.gz emd_19129_half_map_1.map.gz emd_19129_half_map_2.map.gz emd_19129_half_map_2.map.gz | 14.5 MB 14.5 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-19129 http://ftp.pdbj.org/pub/emdb/structures/EMD-19129 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-19129 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-19129 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_19129_validation.pdf.gz emd_19129_validation.pdf.gz | 657.1 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_19129_full_validation.pdf.gz emd_19129_full_validation.pdf.gz | 656.7 KB | 表示 | |
| XML形式データ |  emd_19129_validation.xml.gz emd_19129_validation.xml.gz | 18.4 KB | 表示 | |
| CIF形式データ |  emd_19129_validation.cif.gz emd_19129_validation.cif.gz | 24.5 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-19129 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-19129 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-19129 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-19129 | HTTPS FTP |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_19129.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_19129.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | From cryoSPARC 3DFlex reconstruct job | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 2.2 Å | ||||||||||||||||||||||||||||||||||||
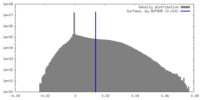
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: From cryoSPARC 3DFlex reconstruct job
| ファイル | emd_19129_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | From cryoSPARC 3DFlex reconstruct job | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
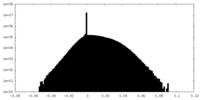
| 密度ヒストグラム |
-ハーフマップ: From cryoSPARC 3DFlex reconstruct job
| ファイル | emd_19129_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | From cryoSPARC 3DFlex reconstruct job | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : DNA origami
| 全体 | 名称: DNA origami |
|---|---|
| 要素 |
|
-超分子 #1: DNA origami
| 超分子 | 名称: DNA origami / タイプ: complex / ID: 1 / 親要素: 0 |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
-超分子 #2: ssDNA staples
| 超分子 | 名称: ssDNA staples / タイプ: complex / ID: 2 / 親要素: 1 |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
-超分子 #3: ssDNA scaffold
| 超分子 | 名称: ssDNA scaffold / タイプ: complex / ID: 3 / 親要素: 1 |
|---|---|
| 由来(天然) | 生物種:  Phage M13mp18 (ファージ) Phage M13mp18 (ファージ) |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1 mg/mL |
|---|---|
| 緩衝液 | pH: 7.4 / 詳細: 5 mM MgCl2, 5 mM NaCl, 5 mM TRIS, and 1 mM EDTA |
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 200 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: CONTINUOUS / 支持フィルム - Film thickness: 3 / 前処理 - タイプ: GLOW DISCHARGE |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 296 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS KRIOS |
|---|---|
| 特殊光学系 | 位相板: VOLTA PHASE PLATE / エネルギーフィルター - スリット幅: 10 eV |
| 撮影 | #0 - Image recording ID: 1 #0 - フィルム・検出器のモデル: GATAN K3 (6k x 4k) #0 - 撮影したグリッド数: 1 / #0 - 実像数: 6652 / #0 - 平均露光時間: 4.4 sec. / #0 - 平均電子線量: 40.0 e/Å2 / #1 - Image recording ID: 2 #1 - フィルム・検出器のモデル: GATAN K3 (6k x 4k) #1 - 撮影したグリッド数: 1 / #1 - 実像数: 5195 / #1 - 平均露光時間: 6.0 sec. / #1 - 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 100.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 1.5 µm / 最小 デフォーカス(公称値): 0.5 µm / 倍率(公称値): 81000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー




 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)