+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | BmrA E504-apo | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | BmrA / multidrug transporter / drug resistance / ABC transporter / TRANSPORT PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.3 Å | |||||||||
 データ登録者 データ登録者 | Gobet A / Zarkadas E / Schoehn G / Falson P / Chaptal V | |||||||||
| 資金援助 |  フランス, 1件 フランス, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2025 ジャーナル: Nat Commun / 年: 2025タイトル: Rhodamine6G and Hœchst33342 narrow BmrA conformational spectrum for a more efficient use of ATP. 著者: A Gobet / L Moissonnier / E Zarkadas / S Magnard / E Bettler / J Martin / R Terreux / G Schoehn / C Orelle / J M Jault / P Falson / V Chaptal /   要旨: Multidrug ABC transporters harness the energy of ATP binding and hydrolysis to translocate substrates out of the cell and detoxify them. While this involves a well-accepted alternating access ...Multidrug ABC transporters harness the energy of ATP binding and hydrolysis to translocate substrates out of the cell and detoxify them. While this involves a well-accepted alternating access mechanism, molecular details of this interplay are still elusive. Rhodamine6G binding on a catalytic inactive mutant of the homodimeric multidrug ABC transporter BmrA triggers a cooperative binding of ATP on the two identical nucleotide-binding-sites, otherwise michaelian. Here, we investigate this asymmetric behavior via a structural-enzymology approach, solving cryoEM structures of BmrA at defined ATP ratios, highlighting the plasticity of BmrA as it undergoes the transition from inward to outward facing conformations. Analysis of continuous heterogeneity within cryoEM data and structural dynamics, reveals that Rhodamine6G narrows the conformational spectrum explored by the nucleotide-binding domains. We observe the same behavior for the other drug Hœchst33342. Following on these findings, the effect of drug-binding showed an ATPase stimulation and a maximal transport activity of the wild-type protein at the concentration-range where the cooperative transition occurs. Altogether, these findings provide a description of the influence of drug binding on the ATP-binding sites through a change in conformational dynamics. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_19113.map.gz emd_19113.map.gz | 59.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-19113-v30.xml emd-19113-v30.xml emd-19113.xml emd-19113.xml | 18.7 KB 18.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_19113_fsc.xml emd_19113_fsc.xml | 8.4 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_19113.png emd_19113.png | 34.6 KB | ||
| Filedesc metadata |  emd-19113.cif.gz emd-19113.cif.gz | 6.4 KB | ||
| その他 |  emd_19113_half_map_1.map.gz emd_19113_half_map_1.map.gz emd_19113_half_map_2.map.gz emd_19113_half_map_2.map.gz | 59.1 MB 59.1 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-19113 http://ftp.pdbj.org/pub/emdb/structures/EMD-19113 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-19113 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-19113 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_19113_validation.pdf.gz emd_19113_validation.pdf.gz | 789.1 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_19113_full_validation.pdf.gz emd_19113_full_validation.pdf.gz | 788.7 KB | 表示 | |
| XML形式データ |  emd_19113_validation.xml.gz emd_19113_validation.xml.gz | 16.2 KB | 表示 | |
| CIF形式データ |  emd_19113_validation.cif.gz emd_19113_validation.cif.gz | 21.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-19113 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-19113 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-19113 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-19113 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8rezMC  8rf1C  8rg7C  8rgaC  8rgnC  8ri1C  8riaC  9gsjC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_19113.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_19113.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.839 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: half map A
| ファイル | emd_19113_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map A | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: half map B
| ファイル | emd_19113_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map B | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Multidrug resistance ABC transporter ATP-binding/permease protein BmrA
| 全体 | 名称: Multidrug resistance ABC transporter ATP-binding/permease protein BmrA |
|---|---|
| 要素 |
|
-超分子 #1: Multidrug resistance ABC transporter ATP-binding/permease protein BmrA
| 超分子 | 名称: Multidrug resistance ABC transporter ATP-binding/permease protein BmrA タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all / 詳細: Apo form, mutant E504A |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 130 KDa |
-分子 #1: Lipid A export ATP-binding/permease protein MsbA
| 分子 | 名称: Lipid A export ATP-binding/permease protein MsbA / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO EC番号: 加水分解酵素; 酸無水物に作用; 酸無水物に作用・物質の膜輸送を触媒する |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 65.747141 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MSSSHHHHHH MPTKKQKSKS KLKPFFALVR RTNPSYGKLA FALALSVVTT LVSLLIPLLT KQLVDGFSMS NLSGTQIGLI ALVFFVQAG LSAYATYALN YNGQKIISGL RELLWKKLIK LPVSYFDTNA SGETVSRVTN DTMVVKELIT THISGFITGI I SVIGSLTI ...文字列: MSSSHHHHHH MPTKKQKSKS KLKPFFALVR RTNPSYGKLA FALALSVVTT LVSLLIPLLT KQLVDGFSMS NLSGTQIGLI ALVFFVQAG LSAYATYALN YNGQKIISGL RELLWKKLIK LPVSYFDTNA SGETVSRVTN DTMVVKELIT THISGFITGI I SVIGSLTI LFIMNWKLTL LVLVVVPLAA LILVPIGRKM FSISRETQDE TARFTGLLNQ ILPEIRLVKA SNAEDVEYGR GK MGISSLF KLGVREAKVQ SLVGPLISLV LMAALVAVIG YGGMQVSSGE LTAGALVAFI LYLFQIIMPM GQITTFFTQL QKS IGATER MIEILAEEEE DTVTGKQIEN AHLPIQLDRV SFGYKPDQLI LKEVSAVIEA GKVTAIVGPS GGGKTTLFKL LERF YSPTA GTIRLGDEPV DTYSLESWRE HIGYVSQESP LMSGTIRENI CYGLERDVTD AEIEKAAEMA YALNFIKELP NQFDT EVGE RGIMLSGGQR QRIAIARALL RNPSILMLDA ATSSLDSQSE KSVQQALEVL MEGRTTIVIA HRLSTVVDAD QLLFVE KGE ITGRGTHHEL MASHGLYRDF AEQQLKMNAD LENKAG UniProtKB: Lipid A export ATP-binding/permease protein MsbA |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 4 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 |
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: GOLD / メッシュ: 300 / 前処理 - タイプ: GLOW DISCHARGE |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 293 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 実像数: 25123 / 平均露光時間: 1.9 sec. / 平均電子線量: 40.12 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 倍率(補正後): 105000 / 照射モード: OTHER / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.4 µm / 最小 デフォーカス(公称値): 1.0 µm |
| 試料ステージ | ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー



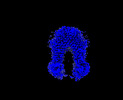









 Z (Sec.)
Z (Sec.) X (Row.)
X (Row.) Y (Col.)
Y (Col.)






































