登録情報 データベース : EMDB / ID : EMD-18764タイトル SWR1-hexasome complex 複合体 : SWR1-hexasome complexタンパク質・ペプチド : x 11種DNA : x 2種リガンド : x 4種 / / 機能・相同性 分子機能 ドメイン・相同性 構成要素
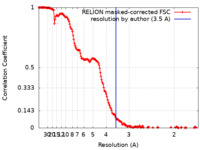
/ / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / 生物種 Saccharomyces cerevisiae S288C (パン酵母) / synthetic construct (人工物) 手法 / / 解像度 : 3.5 Å Jalal ASB / Wigley DB 資金援助 Organization Grant number 国 Wellcome Trust 095519/Z/11/Z & 209327/Z/17/Z Cancer Research UK C6913/A21608 Medical Research Council (MRC, United Kingdom) MR/N009258/1 & MR/R009023/1
ジャーナル : Mol Cell / 年 : 2024タイトル : Stabilization of the hexasome intermediate during histone exchange by yeast SWR1 complex.著者 : Adam S B Jalal / Paul Girvan / Eugene Y D Chua / Lexin Liu / Shijie Wang / Elizabeth A McCormack / Michael T Skehan / Carol L Knight / David S Rueda / Dale B Wigley / 要旨 : The yeast SWR1 complex catalyzes the exchange of histone H2A/H2B dimers in nucleosomes with Htz1/H2B dimers. We use cryoelectron microscopy to determine the structure of an enzyme-bound hexasome ... The yeast SWR1 complex catalyzes the exchange of histone H2A/H2B dimers in nucleosomes with Htz1/H2B dimers. We use cryoelectron microscopy to determine the structure of an enzyme-bound hexasome intermediate in the reaction pathway of histone exchange, in which an H2A/H2B dimer has been extracted from a nucleosome prior to the insertion of a dimer comprising Htz1/H2B. The structure reveals a key role for the Swc5 subunit in stabilizing the unwrapping of DNA from the histone core of the hexasome. By engineering a crosslink between an Htz1/H2B dimer and its chaperone protein Chz1, we show that this blocks histone exchange by SWR1 but allows the incoming chaperone-dimer complex to insert into the hexasome. We use this reagent to trap an SWR1/hexasome complex with an incoming Htz1/H2B dimer that shows how the reaction progresses to the next step. Taken together the structures reveal insights into the mechanism of histone exchange by SWR1 complex. 履歴 登録 2023年10月26日 - ヘッダ(付随情報) 公開 2024年10月2日 - マップ公開 2024年10月2日 - 更新 2024年10月30日 - 現状 2024年10月30日 処理サイト : PDBe / 状態 : 公開
すべて表示 表示を減らす
 データを開く
データを開く 基本情報
基本情報
 マップデータ
マップデータ 試料
試料 キーワード
キーワード 機能・相同性情報
機能・相同性情報
 データ登録者
データ登録者 英国, 3件
英国, 3件  引用
引用 ジャーナル: Mol Cell / 年: 2024
ジャーナル: Mol Cell / 年: 2024
 構造の表示
構造の表示 ダウンロードとリンク
ダウンロードとリンク emd_18764.map.gz
emd_18764.map.gz EMDBマップデータ形式
EMDBマップデータ形式 emd-18764-v30.xml
emd-18764-v30.xml emd-18764.xml
emd-18764.xml EMDBヘッダ
EMDBヘッダ emd_18764_fsc.xml
emd_18764_fsc.xml FSCデータファイル
FSCデータファイル emd_18764.png
emd_18764.png emd-18764.cif.gz
emd-18764.cif.gz emd_18764_half_map_1.map.gz
emd_18764_half_map_1.map.gz emd_18764_half_map_2.map.gz
emd_18764_half_map_2.map.gz http://ftp.pdbj.org/pub/emdb/structures/EMD-18764
http://ftp.pdbj.org/pub/emdb/structures/EMD-18764 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-18764
ftp://ftp.pdbj.org/pub/emdb/structures/EMD-18764 emd_18764_validation.pdf.gz
emd_18764_validation.pdf.gz EMDB検証レポート
EMDB検証レポート emd_18764_full_validation.pdf.gz
emd_18764_full_validation.pdf.gz emd_18764_validation.xml.gz
emd_18764_validation.xml.gz emd_18764_validation.cif.gz
emd_18764_validation.cif.gz https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18764
https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18764 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18764
ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18764 リンク
リンク EMDB (EBI/PDBe) /
EMDB (EBI/PDBe) /  EMDataResource
EMDataResource マップ
マップ ダウンロード / ファイル: emd_18764.map.gz / 形式: CCP4 / 大きさ: 421.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES)
ダウンロード / ファイル: emd_18764.map.gz / 形式: CCP4 / 大きさ: 421.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) 試料の構成要素
試料の構成要素 解析
解析 試料調製
試料調製 電子顕微鏡法
電子顕微鏡法 FIELD EMISSION GUN
FIELD EMISSION GUN
 ムービー
ムービー コントローラー
コントローラー




















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)































 Trichoplusia ni (イラクサキンウワバ)
Trichoplusia ni (イラクサキンウワバ)