+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Subtomogram average of Ebola virus nucleocapsid obtained from cryo-FIB milled Ebola virus infected Huh7 cells at 22 hours post infection | |||||||||
 マップデータ マップデータ | Subtomogram average Ebola condensed nucleocapsid from Ebola virus infected cells (low pass filtered to 21A resolution) | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Ebola virus nucleocapsid / VIRUS | |||||||||
| 生物種 |  | |||||||||
| 手法 | サブトモグラム平均法 / クライオ電子顕微鏡法 / 解像度: 21.0 Å | |||||||||
 データ登録者 データ登録者 | Vallbracht M / Chlanda P | |||||||||
| 資金援助 |  ドイツ, 1件 ドイツ, 1件
| |||||||||
 引用 引用 |  ジャーナル: Cell / 年: 2025 ジャーナル: Cell / 年: 2025タイトル: Nucleocapsid assembly drives Ebola viral factory maturation and dispersion. 著者: Melina Vallbracht / Bianca S Bodmer / Konstantin Fischer / Jana Makroczyova / Sophie L Winter / Lisa Wendt / Moritz Wachsmuth-Melm / Thomas Hoenen / Petr Chlanda /  要旨: Replication and genome encapsidation of many negative-sense RNA viruses take place in virus-induced membraneless organelles termed viral factories (VFs). Although liquid properties of VFs are ...Replication and genome encapsidation of many negative-sense RNA viruses take place in virus-induced membraneless organelles termed viral factories (VFs). Although liquid properties of VFs are believed to control the transition from genome replication to nucleocapsid (NC) assembly, VF maturation and interactions with the cellular environment remain elusive. Here, we apply in situ cryo-correlative light and electron tomography to follow NC assembly and changes in VF morphology and their liquid properties during Ebola virus infection. We show that viral NCs transition from loosely packed helical assemblies in early VFs to compact cylinders that arrange into highly organized parallel bundles later in infection. Early VFs associate with intermediate filaments and are devoid of other host material but become progressively accessible to cellular components. Our data suggest that this process is coupled to VF solidification, loss of sphericity, and dispersion and promotes cytoplasmic exposure of NCs to facilitate their transport to budding sites. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_18697.map.gz emd_18697.map.gz | 58.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-18697-v30.xml emd-18697-v30.xml emd-18697.xml emd-18697.xml | 14.8 KB 14.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_18697_fsc.xml emd_18697_fsc.xml | 11.8 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_18697.png emd_18697.png | 109.7 KB | ||
| マスクデータ |  emd_18697_msk_1.map emd_18697_msk_1.map | 64 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-18697.cif.gz emd-18697.cif.gz | 4.6 KB | ||
| その他 |  emd_18697_half_map_1.map.gz emd_18697_half_map_1.map.gz emd_18697_half_map_2.map.gz emd_18697_half_map_2.map.gz | 58.9 MB 59 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-18697 http://ftp.pdbj.org/pub/emdb/structures/EMD-18697 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-18697 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-18697 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_18697_validation.pdf.gz emd_18697_validation.pdf.gz | 1 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_18697_full_validation.pdf.gz emd_18697_full_validation.pdf.gz | 1 MB | 表示 | |
| XML形式データ |  emd_18697_validation.xml.gz emd_18697_validation.xml.gz | 16.3 KB | 表示 | |
| CIF形式データ |  emd_18697_validation.cif.gz emd_18697_validation.cif.gz | 21.8 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18697 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18697 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18697 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18697 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_18697.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_18697.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Subtomogram average Ebola condensed nucleocapsid from Ebola virus infected cells (low pass filtered to 21A resolution) | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 2.671 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_18697_msk_1.map emd_18697_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
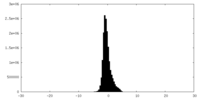
| 密度ヒストグラム |
-ハーフマップ: Subtomogram average half map of Ebola condensed nucleocapsid
| ファイル | emd_18697_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Subtomogram average half map of Ebola condensed nucleocapsid | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
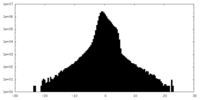
| 密度ヒストグラム |
-ハーフマップ: Subtomogram average half map of Ebola condensed nucleocapsid
| ファイル | emd_18697_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Subtomogram average half map of Ebola condensed nucleocapsid | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Ebola virus - Mayinga, Zaire, 1976
| 全体 | 名称:  |
|---|---|
| 要素 |
|
-超分子 #1: Ebola virus - Mayinga, Zaire, 1976
| 超分子 | 名称: Ebola virus - Mayinga, Zaire, 1976 / タイプ: virus / ID: 1 / 親要素: 0 / NCBI-ID: 128952 / 生物種: Ebola virus - Mayinga, Zaire, 1976 / ウイルスタイプ: VIRION / ウイルス・単離状態: STRAIN / ウイルス・エンベロープ: Yes / ウイルス・中空状態: No |
|---|---|
| ウイルス殻 | Shell ID: 1 / 名称: Nucleocapsid / 直径: 500.0 Å |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | サブトモグラム平均法 |
| 試料の集合状態 | cell |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 |
|---|---|
| グリッド | モデル: Quantifoil / 材質: GOLD / メッシュ: 200 / 前処理 - タイプ: GLOW DISCHARGE |
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 3.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 4.0 µm / 最小 デフォーカス(公称値): 2.5 µm / 倍率(公称値): 33000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー











 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)