+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-electron tomogram of lift-out lamella from cell-derived extracellular matrix (example 2) | |||||||||||||||
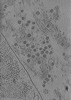
 マップデータ マップデータ | Cryo-electron tomogram containing cell-derived extracellular matrix obtained via lift-out focused ion beam milling | |||||||||||||||
 試料 試料 |
| |||||||||||||||
 キーワード キーワード | Extracellular matrix / cell-derived matrix / collagen / PROTEIN FIBRIL | |||||||||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||||||||
| 手法 | 電子線トモグラフィー法 / クライオ電子顕微鏡法 | |||||||||||||||
 データ登録者 データ登録者 | Zens B / Faessler F / Hansen J / Hauschild R / Datler J / Hodirnau V-V / Zheden V / Alanko J / Sixt MK / Schur FKM | |||||||||||||||
| 資金援助 |  オーストリア, オーストリア,  米国, European Union, 4件 米国, European Union, 4件
| |||||||||||||||
 引用 引用 |  ジャーナル: J Cell Biol / 年: 2024 ジャーナル: J Cell Biol / 年: 2024タイトル: Lift-out cryo-FIBSEM and cryo-ET reveal the ultrastructural landscape of extracellular matrix. 著者: Bettina Zens / Florian Fäßler / Jesse M Hansen / Robert Hauschild / Julia Datler / Victor-Valentin Hodirnau / Vanessa Zheden / Jonna Alanko / Michael Sixt / Florian K M Schur /  要旨: The extracellular matrix (ECM) serves as a scaffold for cells and plays an essential role in regulating numerous cellular processes, including cell migration and proliferation. Due to limitations in ...The extracellular matrix (ECM) serves as a scaffold for cells and plays an essential role in regulating numerous cellular processes, including cell migration and proliferation. Due to limitations in specimen preparation for conventional room-temperature electron microscopy, we lack structural knowledge on how ECM components are secreted, remodeled, and interact with surrounding cells. We have developed a 3D-ECM platform compatible with sample thinning by cryo-focused ion beam milling, the lift-out extraction procedure, and cryo-electron tomography. Our workflow implements cell-derived matrices (CDMs) grown on EM grids, resulting in a versatile tool closely mimicking ECM environments. This allows us to visualize ECM for the first time in its hydrated, native context. Our data reveal an intricate network of extracellular fibers, their positioning relative to matrix-secreting cells, and previously unresolved structural entities. Our workflow and results add to the structural atlas of the ECM, providing novel insights into its secretion and assembly. | |||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_18491.map.gz emd_18491.map.gz | 295.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-18491-v30.xml emd-18491-v30.xml emd-18491.xml emd-18491.xml | 14.6 KB 14.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_18491.png emd_18491.png | 358.4 KB | ||
| Filedesc metadata |  emd-18491.cif.gz emd-18491.cif.gz | 5.1 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-18491 http://ftp.pdbj.org/pub/emdb/structures/EMD-18491 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-18491 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-18491 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_18491_validation.pdf.gz emd_18491_validation.pdf.gz | 486.2 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_18491_full_validation.pdf.gz emd_18491_full_validation.pdf.gz | 485.7 KB | 表示 | |
| XML形式データ |  emd_18491_validation.xml.gz emd_18491_validation.xml.gz | 4.6 KB | 表示 | |
| CIF形式データ |  emd_18491_validation.cif.gz emd_18491_validation.cif.gz | 5 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18491 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18491 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18491 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18491 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_18491.map.gz / 形式: CCP4 / 大きさ: 330.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_18491.map.gz / 形式: CCP4 / 大きさ: 330.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Cryo-electron tomogram containing cell-derived extracellular matrix obtained via lift-out focused ion beam milling | ||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 これらの図は立方格子座標系で作成されたものです | ||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 17.096 Å | ||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Cryo-lamella prepared by lift-out cryo-FIB milling of cell-derive...
| 全体 | 名称: Cryo-lamella prepared by lift-out cryo-FIB milling of cell-derived matrix from human telomerase-immortalized foreskin fibroblasts (TIFFs) |
|---|---|
| 要素 |
|
-超分子 #1: Cryo-lamella prepared by lift-out cryo-FIB milling of cell-derive...
| 超分子 | 名称: Cryo-lamella prepared by lift-out cryo-FIB milling of cell-derived matrix from human telomerase-immortalized foreskin fibroblasts (TIFFs) タイプ: cell / ID: 1 / 親要素: 0 詳細: Cell-derived matrix was generated by growing TIFF cells on EM grids over 14 days of growth time. |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) / 器官: Skin / 組織: Foreskin Homo sapiens (ヒト) / 器官: Skin / 組織: Foreskin |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 電子線トモグラフィー法 |
| 試料の集合状態 | cell |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 / 構成要素 - 濃度: 10.0 Percent / 構成要素 - 名称: Dextran / 詳細: 10% Dextran in 0.1M Phosphate Buffer, pH 7.4 |
|---|---|
| グリッド | モデル: Quantifoil R2/2 / 材質: GOLD / メッシュ: 200 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 120 sec. / 前処理 - 雰囲気: AIR / 詳細: ELMO Glow Discharge unit |
| 凍結 | 凍結剤: NITROGEN |
| 詳細 | Cell-derived matrix obtained from TIFF-cells. |
| 加圧凍結法 | 装置: OTHER 詳細: High pressure freezing carriers were 3.0mm in diameter and 0.5mm thick. One carrier was flat, the second carrier had a central cavity of 2.0mm diameter and 0.02mm depth. Carriers were coated ...詳細: High pressure freezing carriers were 3.0mm in diameter and 0.5mm thick. One carrier was flat, the second carrier had a central cavity of 2.0mm diameter and 0.02mm depth. Carriers were coated in 1-hexadecene prior to high pressure freezing.. The value given for _em_high_pressure_freezing.instrument is Bal-tec HPM010. This is not in a list of allowed values {'BAL-TEC HPM 010', 'EMS-002 RAPID IMMERSION FREEZER', 'LEICA EM PACT', 'LEICA EM PACT2', 'LEICA EM HPM100', 'OTHER'} so OTHER is written into the XML file. |
| Cryo protectant | 10% Dextran (high MW) |
| 切片作成 | 集束イオンビーム - 装置: OTHER / 集束イオンビーム - イオン: OTHER / 集束イオンビーム - 電圧: 30 / 集束イオンビーム - 電流: 0.03 / 集束イオンビーム - 時間: 3600 / 集束イオンビーム - 温度: 80 K / 集束イオンビーム - Initial thickness: 1000 / 集束イオンビーム - 最終 厚さ: 250 集束イオンビーム - 詳細: A block of sample was extracted from the sample bulk by cryo-lift out FIB milling, with a thickness of 5000-8000nm. It was attached to a second grid, where it was ...集束イオンビーム - 詳細: A block of sample was extracted from the sample bulk by cryo-lift out FIB milling, with a thickness of 5000-8000nm. It was attached to a second grid, where it was then milled down to 200-250nm thickness step by step. Lamellae were milled in consecutive steps with 1nA (down to 3000nm thickness), 0.5nA (2000nm thickness), 0.1nA (1000nm), 50pA (500nm) and 30pA (200nm). See associated publication for details.. The value given for _em_focused_ion_beam.instrument is TFS Aquilos II. This is not in a list of allowed values {'OTHER', 'DB235'} so OTHER is written into the XML file. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS KRIOS |
|---|---|
| 温度 | 最低: 80.0 K / 最高: 83.0 K |
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) デジタル化 - サイズ - 横: 5760 pixel / デジタル化 - サイズ - 縦: 4092 pixel / 平均露光時間: 0.5 sec. / 平均電子線量: 2.137 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 倍率(補正後): 42000 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 8.0 µm / 最小 デフォーカス(公称値): 8.0 µm / 倍率(公称値): 42000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 最終 再構成 | アルゴリズム: BACK PROJECTION 詳細: Reconstructed with AreTomo (version 1.3.4., Feb22) and processed with IsoNet (version 0.2). 使用した粒子像数: 61 |
|---|
 ムービー
ムービー コントローラー
コントローラー








 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)