[日本語] English
 万見
万見- EMDB-18328: CryoEM structure of a S. Cerevisiae Ski2387 complex in the open state -
+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | CryoEM structure of a S. Cerevisiae Ski2387 complex in the open state | |||||||||
 マップデータ マップデータ | Main map obtained from signal subtraction of Ski3-Nt followed by local refinement | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Helicase / RNA binding / RNA degradation / HYDROLASE | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報mRNA decay by 3' to 5' exoribonuclease / protein-DNA complex assembly / Ski complex / nuclear-transcribed mRNA catabolic process, non-stop decay / nuclear-transcribed mRNA catabolic process, 3'-5' exonucleolytic nonsense-mediated decay / reciprocal meiotic recombination / nonfunctional rRNA decay / nuclear chromosome / mRNA catabolic process / nuclear-transcribed mRNA catabolic process ...mRNA decay by 3' to 5' exoribonuclease / protein-DNA complex assembly / Ski complex / nuclear-transcribed mRNA catabolic process, non-stop decay / nuclear-transcribed mRNA catabolic process, 3'-5' exonucleolytic nonsense-mediated decay / reciprocal meiotic recombination / nonfunctional rRNA decay / nuclear chromosome / mRNA catabolic process / nuclear-transcribed mRNA catabolic process / protein catabolic process / regulation of translation / protein-containing complex assembly / protein-macromolecule adaptor activity / defense response to virus / RNA helicase activity / RNA helicase / translation / mRNA binding / GTPase activity / GTP binding / protein-containing complex binding / ATP hydrolysis activity / DNA binding / ATP binding / nucleus / cytosol / cytoplasm 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
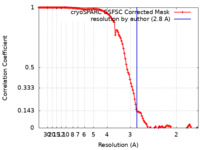
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.8 Å | |||||||||
 データ登録者 データ登録者 | Keidel A / Koegel A / Reichelt P / Kowalinski E / Schaefer IB / Conti E | |||||||||
| 資金援助 |  ドイツ, 1件 ドイツ, 1件
| |||||||||
 引用 引用 |  ジャーナル: Mol Cell / 年: 2023 ジャーナル: Mol Cell / 年: 2023タイトル: Concerted structural rearrangements enable RNA channeling into the cytoplasmic Ski238-Ski7-exosome assembly. 著者: Achim Keidel / Alexander Kögel / Peter Reichelt / Eva Kowalinski / Ingmar B Schäfer / Elena Conti /   要旨: The Ski2-Ski3-Ski8 (Ski238) helicase complex directs cytoplasmic mRNAs toward the nucleolytic exosome complex for degradation. In yeast, the interaction between Ski238 and exosome requires the ...The Ski2-Ski3-Ski8 (Ski238) helicase complex directs cytoplasmic mRNAs toward the nucleolytic exosome complex for degradation. In yeast, the interaction between Ski238 and exosome requires the adaptor protein Ski7. We determined different cryo-EM structures of the Ski238 complex depicting the transition from a rigid autoinhibited closed conformation to a flexible active open conformation in which the Ski2 helicase module has detached from the rest of Ski238. The open conformation favors the interaction of the Ski3 subunit with exosome-bound Ski7, leading to the recruitment of the exosome. In the Ski238-Ski7-exosome holocomplex, the Ski2 helicase module binds the exosome cap, enabling the RNA to traverse from the helicase through the internal exosome channel to the Rrp44 exoribonuclease. Our study pinpoints how conformational changes within the Ski238 complex regulate exosome recruitment for RNA degradation. We also reveal the remarkable conservation of helicase-exosome RNA channeling mechanisms throughout eukaryotic nuclear and cytoplasmic exosome complexes. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_18328.map.gz emd_18328.map.gz | 156.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-18328-v30.xml emd-18328-v30.xml emd-18328.xml emd-18328.xml | 27.6 KB 27.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_18328_fsc.xml emd_18328_fsc.xml | 11.6 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_18328.png emd_18328.png | 25.9 KB | ||
| マスクデータ |  emd_18328_msk_1.map emd_18328_msk_1.map | 166.4 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-18328.cif.gz emd-18328.cif.gz | 8.6 KB | ||
| その他 |  emd_18328_additional_1.map.gz emd_18328_additional_1.map.gz emd_18328_additional_2.map.gz emd_18328_additional_2.map.gz emd_18328_half_map_1.map.gz emd_18328_half_map_1.map.gz emd_18328_half_map_2.map.gz emd_18328_half_map_2.map.gz | 148.7 MB 156.9 MB 154.2 MB 154.2 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-18328 http://ftp.pdbj.org/pub/emdb/structures/EMD-18328 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-18328 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-18328 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_18328_validation.pdf.gz emd_18328_validation.pdf.gz | 982.7 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_18328_full_validation.pdf.gz emd_18328_full_validation.pdf.gz | 982.3 KB | 表示 | |
| XML形式データ |  emd_18328_validation.xml.gz emd_18328_validation.xml.gz | 19.5 KB | 表示 | |
| CIF形式データ |  emd_18328_validation.cif.gz emd_18328_validation.cif.gz | 25.1 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18328 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18328 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18328 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18328 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_18328.map.gz / 形式: CCP4 / 大きさ: 166.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_18328.map.gz / 形式: CCP4 / 大きさ: 166.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Main map obtained from signal subtraction of Ski3-Nt followed by local refinement | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.8512 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
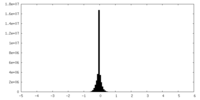
| ファイル |  emd_18328_msk_1.map emd_18328_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: sharpened with deepEMhancer software (tightTarget)
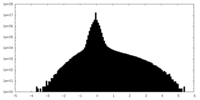
| ファイル | emd_18328_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | sharpened with deepEMhancer software (tightTarget) | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: full map prior to signal subtraction
| ファイル | emd_18328_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | full map prior to signal subtraction | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
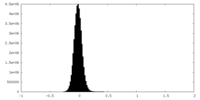
| ファイル | emd_18328_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
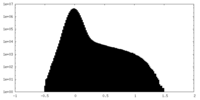
| ファイル | emd_18328_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Ski2delarch387 PolyU RNA
| 全体 | 名称: Ski2delarch387 PolyU RNA |
|---|---|
| 要素 |
|
-超分子 #1: Ski2delarch387 PolyU RNA
| 超分子 | 名称: Ski2delarch387 PolyU RNA / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: Antiviral helicase SKI2
| 分子 | 名称: Antiviral helicase SKI2 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO / EC番号: RNA helicase |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 146.259094 KDa |
| 組換発現 | 生物種:  Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 配列 | 文字列: MSEGFSSSSI QELYQSLKEI TNNADVELFE DRITKLDFES TDEPKHANDI IKDRFLRPSN ALPWSLLDMV QDVPHTSSPE DCSGKLDYK ELLKVPDPIN RTSYQFKRTG LEGKISGYKE EVDLKEVANA NASNSLSITR SINHNQNSVR GSTAQLPFTP G GIPMKSVK ...文字列: MSEGFSSSSI QELYQSLKEI TNNADVELFE DRITKLDFES TDEPKHANDI IKDRFLRPSN ALPWSLLDMV QDVPHTSSPE DCSGKLDYK ELLKVPDPIN RTSYQFKRTG LEGKISGYKE EVDLKEVANA NASNSLSITR SINHNQNSVR GSTAQLPFTP G GIPMKSVK TDSEQNGSST MANATKLLHK DGQGLFDIPE GMNRGIKPMD SPAENEDQNG QFKELKQLNE IDNELDIRIE AN EAKLKEE EKSAKSISEE IMEEATEETT ADNADDAEID ELLPIGIDFG RTKPVSKSVP VKKEWAHVVD LNHKIENFDE LIP NPARSW PFELDTFQKE AVYHLEQGDS VFVAAHTSAG KTVVAEYAIA MAHRNMTKTI YTSPIKALSN QKFRDFKETF DDVN IGLIT GDVQINPDAN CLIMTTEILR SMLYRGADLI RDVEFVIFDE VHYVNDQDRG VVWEEVIIML PQHVKFILLS ATVPN TYEF ANWIGRTKQK NIYVISTPKR PVPLEINIWA KKELIPVINQ NSEFLEANFR KHKEILNGES AKGAPSKTDN GRGGST ARG GRGGSNTRDG RGGRGNSTRG GANRGGSRGA GAIGSNKRKF FTQDGPSKKT WPEIVNYLRK RELLPMVVFV FSKKRCE EY ADWLEGINFC NNKEKSQIHM FIEKSITRLK KEDRDLPQIL KTRSLLERGI AVHHGGLLPI VKELIEILFS KGFIKVLF A TETFAMGLNL PTRTVIFSSI RKHDGNGLRE LTPGEFTQMA GRAGRRGLDS TGTVIVMAYN SPLSIATFKE VTMGVPTRL QSQFRLTYNM ILNLLRIEAL RVEEMIKYSF SENAKETLQP EHEKQIKVLQ EELQTIEYKS CEICDNDIEK FLELMLAYKE ATVNLMQEM VKSPSILHIL KEGRLVAFRD PNDCLKLGFV FKVSLKDAVC VIMTFTKPYK LPNGEPNHLI YFPKADGYRR R NFPKFQKT DFYMEEVPVT AIEVITKRKF AAPLGKVIKK DVAALNEFNA ETNNILDGKT LKEAINIEKQ GLKIHQILLD RT NIRDEIF KLKSIKCPNL SQHIVPKFKA HVIKKKIEEL YHLMSDQNLS LLPDYEKRLA VLKDTEFIDQ NHNVLLKGRV ACE INSGYE LVLTELILDN FLGSFEPEEI VALLSVFVYE GKTREEEPPI VTPRLAKGKQ RIEEIYKKML CVFNTHQIPL TQDE AEFLD RKRFAMMNVV YEWARGLSFK EIMEMSPEAE GTVVRVITWL DEICREVKTA SIIIGNSTLH MKMSRAQELI KRDIV FAAS LYL UniProtKB: Antiviral helicase SKI2 |
-分子 #2: Superkiller protein 3
| 分子 | 名称: Superkiller protein 3 / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 164.278484 KDa |
| 組換発現 | 生物種:  Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 配列 | 文字列: GPDSMSDIKQ LLKEAKQELT NRDYEETIEI SEKVLKLDPD NYFAHIFLGK ALSSLPASNN VSSNRNLERA TNHYVSAAKL VPDNLLAWK GLFLLFRTTE VVPDILSYDE YFDLCGQYAD ALLKQEQSQV ELINDIKLLK KTHPDCQKAF YQHLKPGSLM A ETIGRHLS ...文字列: GPDSMSDIKQ LLKEAKQELT NRDYEETIEI SEKVLKLDPD NYFAHIFLGK ALSSLPASNN VSSNRNLERA TNHYVSAAKL VPDNLLAWK GLFLLFRTTE VVPDILSYDE YFDLCGQYAD ALLKQEQSQV ELINDIKLLK KTHPDCQKAF YQHLKPGSLM A ETIGRHLS TPQDALLNLI KILSNIETTE IGKTLSQNRL KLKASDPDYQ IKLNSFSWEI IKNSEIDQLY NQLVNILADD QK RSEIENQ WLEYRIKVLK SMPLDVKKDF FTKVKEMVED MVLVNHQSLL AWQKYFEWTD YEDLDNMDAP LIIKYFKKFP KDP LAMILY SWLSSKLSKY DIKSLESANK PPEGHKKTEK ETDIKDVDET NEDEVKDRVE DEVKDRVEDE VKDQDEEAKE DEEE DLDDI EIGLLEEEVV TVLTENIVKC KNNILAHRIL CQYYLLTKEY EAALPYIKNG ISLIAYNIKD LGVHLPLTKR EFSLD LATV YTYVDAPKDH NAALKLYDNI LSGDFSNIQA KMGKGIIFIE RKNWKDAMTL LTQVHEQSPN NLEVLSELSW SKAHMG YMD EALAGLDTVI KGIKGMDLRS IDFRALNLWR QAKVYIMKHA SINDAKQENV KCAFKLLIQS IKILDTFAPG FSTLGDI YC HYYKDHLRAF KCYFKAFDLD AGDYTAAKYI TETYASKPNW QAASSIASRL IKGEKAKAEL RSNNWPFRVV GIAHLEKQ E ESDSIEWFQS ALRVDPNDVE SWVGLGQAYH ACGRIEASIK VFDKAIQLRP SHTFAQYFKA ISLCDVGEYL ESLDILEKV CQEAATEESF QIGLVEVLMR CSLDLYSQGF LLKSVSIAKD TIERIKIIIS ELKCENQQVW IYLSQVLRLF IWIESKVDTL PVESLVSIF ENSQFSGSEE IDSVDNIKID TLLDSTTDDN VSIACKFLIL ASKYSVSDQK FTDIAGTVRA SYWYNIGISE L TAFITLKE PQYRDAAIFA FKKSIQLQSN TSETWIGLGI ATMDINFRVS QHCFIKATAL EPKATNTWFN LAMLGLKKKD TE FAQQVLN KLQSLAPQDS SPWLGMALIL EEQGDIIGSS KLFAHSFILS NGRSKAAQFM YAKNVLENHI NNGDDERDIE TVE KLTTAS IALEQFFKKS PDSQFALQCA LLTLERLHHY ENANELANRL IGILEKKFEK TQDERELFNF AIIKGQFARI HLGL GNFEL SIENADLSQG IISESSDEKS MKTKISNHIC LGLSYFFLND FDQTLNQFQE LLSISKDSKH LVVLIAKVLY DVGES DTKE IALQELTEYI ATSGADLLVT LTIAAMSILD DKREDLSIIL EELKALPLSK QIIDKHKDAP YLIEEITKRL YRNDTG KQV WQRSAYFFPN NLKVWERLDK NIQRRIASNG QNKVTAEEMS KLYCESKNLR SIQRGMFLCP WNVTAVKALN ECF UniProtKB: Superkiller protein 3 |
-分子 #3: Antiviral protein SKI8
| 分子 | 名称: Antiviral protein SKI8 / タイプ: protein_or_peptide / ID: 3 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 44.283527 KDa |
| 組換発現 | 生物種:  Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 配列 | 文字列: MSKVFIATAN AGKAHDADIF SVSACNSFTV SCSGDGYLKV WDNKLLDNEN PKDKSYSHFV HKSGLHHVDV LQAIERDAFE LCLVATTSF SGDLLFYRIT REDETKKVIF EKLDLLDSDM KKHSFWALKW GASNDRLLSH RLVATDVKGT TYIWKFHPFA D ESNSLTLN ...文字列: MSKVFIATAN AGKAHDADIF SVSACNSFTV SCSGDGYLKV WDNKLLDNEN PKDKSYSHFV HKSGLHHVDV LQAIERDAFE LCLVATTSF SGDLLFYRIT REDETKKVIF EKLDLLDSDM KKHSFWALKW GASNDRLLSH RLVATDVKGT TYIWKFHPFA D ESNSLTLN WSPTLELQGT VESPMTPSQF ATSVDISERG LIATGFNNGT VQISELSTLR PLYNFESQHS MINNSNSIRS VK FSPQGSL LAIAHDSNSF GCITLYETEF GERIGSLSVP THSSQASLGE FAHSSWVMSL SFNDSGETLC SAGWDGKLRF WDV KTKERI TTLNMHCDDI EIEEDILAVD EHGDSLAEPG VFDVKFLKKG WRSGMGADLN ESLCCVCLDR SIRWFREAGG K UniProtKB: Antiviral protein SKI8 |
-分子 #4: Superkiller protein 7
| 分子 | 名称: Superkiller protein 7 / タイプ: protein_or_peptide / ID: 4 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 28.345137 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: GPDSMSLLEQ LARKRIEKSK GLLSADQSHS TSKSASLLER LHKNRETKDN NAETKRKDLK TLLAKDKVKR SDFTPNQHSV SLSLKLSAL KKSNSDLEKQ GKSVTLDSKE NELPTKRKSP DDKLNLEESW KAIKEMNHYC FLKNDPCINQ TDDFAFTNFI I KDKKNSLS ...文字列: GPDSMSLLEQ LARKRIEKSK GLLSADQSHS TSKSASLLER LHKNRETKDN NAETKRKDLK TLLAKDKVKR SDFTPNQHSV SLSLKLSAL KKSNSDLEKQ GKSVTLDSKE NELPTKRKSP DDKLNLEESW KAIKEMNHYC FLKNDPCINQ TDDFAFTNFI I KDKKNSLS TSIPLSSQNS SFLSLKKHNN ELLGIFVPCN LPKTTRKVAI ENFNRPSPDD IIQSAQLNAF NEKLENLNIK SA GSWSHPQ FEK UniProtKB: Superkiller protein 7 |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 |
|---|---|
| 凍結 | 凍結剤: ETHANE-PROPANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 実像数: 7127 / 平均電子線量: 77.7 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.2 µm / 最小 デフォーカス(公称値): 0.6 µm / 倍率(公称値): 105000 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー



















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)