+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | consensus map of the ASFV RNA polymerase in open conformation | ||||||||||||
 マップデータ マップデータ | 3D refinement map (not sharpened) | ||||||||||||
 試料 試料 |
| ||||||||||||
 キーワード キーワード | RNA polymerase / ASFV / transcription / eukaryotic virus | ||||||||||||
| 生物種 |  African swine fever virus BA71V (ウイルス) African swine fever virus BA71V (ウイルス) | ||||||||||||
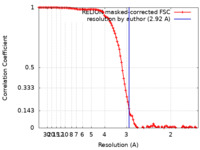
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.92 Å | ||||||||||||
 データ登録者 データ登録者 | Pilotto S / Sykora M / Cackett G / Werner F | ||||||||||||
| 資金援助 |  英国, 3件 英国, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2024 ジャーナル: Nat Commun / 年: 2024タイトル: Structure of the recombinant RNA polymerase from African Swine Fever Virus. 著者: Simona Pilotto / Michal Sýkora / Gwenny Cackett / Christopher Dulson / Finn Werner /  要旨: African Swine Fever Virus is a Nucleo-Cytoplasmic Large DNA Virus that causes an incurable haemorrhagic fever in pigs with a high impact on global food security. ASFV replicates in the cytoplasm of ...African Swine Fever Virus is a Nucleo-Cytoplasmic Large DNA Virus that causes an incurable haemorrhagic fever in pigs with a high impact on global food security. ASFV replicates in the cytoplasm of the infected cell and encodes its own transcription machinery that is independent of cellular factors, however, not much is known about how this system works at a molecular level. Here, we present methods to produce recombinant ASFV RNA polymerase, functional assays to screen for inhibitors, and high-resolution cryo-electron microscopy structures of the ASFV RNAP in different conformational states. The ASFV RNAP bears a striking resemblance to RNAPII with bona fide homologues of nine of its twelve subunits. Key differences include the fusion of the ASFV assembly platform subunits RPB3 and RPB11, and an unusual C-terminal domain of the stalk subunit vRPB7 that is related to the eukaryotic mRNA cap 2´-O-methyltransferase 1. Despite the high degree of structural conservation with cellular RNA polymerases, the ASFV RNAP is resistant to the inhibitors rifampicin and alpha-amanitin. The cryo-EM structures and fully recombinant RNAP system together provide an important tool for the design, development, and screening of antiviral drugs in a low biosafety containment environment. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_18163.map.gz emd_18163.map.gz | 80.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-18163-v30.xml emd-18163-v30.xml emd-18163.xml emd-18163.xml | 18.8 KB 18.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_18163_fsc.xml emd_18163_fsc.xml | 10.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_18163.png emd_18163.png | 105.1 KB | ||
| Filedesc metadata |  emd-18163.cif.gz emd-18163.cif.gz | 5.1 KB | ||
| その他 |  emd_18163_half_map_1.map.gz emd_18163_half_map_1.map.gz emd_18163_half_map_2.map.gz emd_18163_half_map_2.map.gz | 81 MB 81 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-18163 http://ftp.pdbj.org/pub/emdb/structures/EMD-18163 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-18163 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-18163 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_18163_validation.pdf.gz emd_18163_validation.pdf.gz | 969.3 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_18163_full_validation.pdf.gz emd_18163_full_validation.pdf.gz | 968.9 KB | 表示 | |
| XML形式データ |  emd_18163_validation.xml.gz emd_18163_validation.xml.gz | 17.6 KB | 表示 | |
| CIF形式データ |  emd_18163_validation.cif.gz emd_18163_validation.cif.gz | 22.8 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18163 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18163 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18163 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18163 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_18163.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_18163.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | 3D refinement map (not sharpened) | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.828 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: half 2 map
| ファイル | emd_18163_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
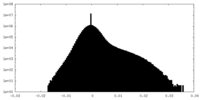
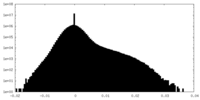
| 注釈 | half 2 map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: half 1 map
| ファイル | emd_18163_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
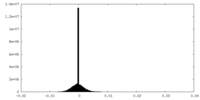
| 注釈 | half 1 map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : apo-form of the 8-subunit RNA polymerase from African Swine Fever...
| 全体 | 名称: apo-form of the 8-subunit RNA polymerase from African Swine Fever Virus |
|---|---|
| 要素 |
|
-超分子 #1: apo-form of the 8-subunit RNA polymerase from African Swine Fever...
| 超分子 | 名称: apo-form of the 8-subunit RNA polymerase from African Swine Fever Virus タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#8 |
|---|---|
| 由来(天然) | 生物種:  African swine fever virus BA71V (ウイルス) African swine fever virus BA71V (ウイルス) |
| 分子量 | 理論値: 450 KDa |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.05 mg/mL | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 8 構成要素:
| ||||||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 95 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV | ||||||||||||||||||
| 詳細 | This sample was monodisparse |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 実像数: 14638 / 平均露光時間: 2.6 sec. / 平均電子線量: 48.152 e/Å2 詳細: Images were collected in movie-mode for a total of 50 frames per image. The data collection was carried out in super-resolution mode and binned 2 on-the-fly. |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.7 µm / 最小 デフォーカス(公称値): 1.5 µm / 倍率(公称値): 105000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | PDB ID: Chain - Source name: Other / Chain - Initial model type: experimental model 詳細: The complete biological assembly for the PDB entry 8Q3B was used for the docking in the new map |
|---|---|
| 精密化 | 空間: REAL / プロトコル: FLEXIBLE FIT / 温度因子: 84.5995 当てはまり具合の基準: cross-correlation coefficient |
 ムービー
ムービー コントローラー
コントローラー









 Z
Z Y
Y X
X