+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | ||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
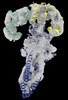
| タイトル | GBP1 bound by 14-3-3sigma | ||||||||||||||||||||||||||||||
 マップデータ マップデータ | |||||||||||||||||||||||||||||||
 試料 試料 |
| ||||||||||||||||||||||||||||||
 キーワード キーワード | Protein complex / Phosphorylation / IMMUNE SYSTEM | ||||||||||||||||||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報GDP phosphatase activity / non-canonical inflammasome complex assembly / negative regulation of substrate adhesion-dependent cell spreading / protein localization to vacuole / symbiont cell surface / cytolysis in another organism / positive regulation of pyroptotic inflammatory response / vesicle membrane / negative regulation of interleukin-2 production / negative regulation of T cell receptor signaling pathway ...GDP phosphatase activity / non-canonical inflammasome complex assembly / negative regulation of substrate adhesion-dependent cell spreading / protein localization to vacuole / symbiont cell surface / cytolysis in another organism / positive regulation of pyroptotic inflammatory response / vesicle membrane / negative regulation of interleukin-2 production / negative regulation of T cell receptor signaling pathway / spectrin binding / defense response to protozoan / regulation of epidermal cell division / protein kinase C inhibitor activity / positive regulation of epidermal cell differentiation / keratinocyte development / keratinization / cytokine binding / regulation of cell-cell adhesion / cAMP/PKA signal transduction / Regulation of localization of FOXO transcription factors / keratinocyte proliferation / cellular response to interleukin-1 / phosphoserine residue binding / Activation of BAD and translocation to mitochondria / negative regulation of keratinocyte proliferation / establishment of skin barrier / negative regulation of protein localization to plasma membrane / 加水分解酵素; 酸無水物に作用; リン含有酸無水物に作用 / Chk1/Chk2(Cds1) mediated inactivation of Cyclin B:Cdk1 complex / SARS-CoV-2 targets host intracellular signalling and regulatory pathways / regulation of protein localization to plasma membrane / negative regulation of protein kinase activity / negative regulation of stem cell proliferation / SARS-CoV-1 targets host intracellular signalling and regulatory pathways / RHO GTPases activate PKNs / positive regulation of protein localization / regulation of calcium-mediated signaling / positive regulation of cell adhesion / protein sequestering activity / protein export from nucleus / negative regulation of innate immune response / cytoplasmic vesicle membrane / TP53 Regulates Transcription of Genes Involved in G2 Cell Cycle Arrest / release of cytochrome c from mitochondria / positive regulation of protein export from nucleus / stem cell proliferation / TP53 Regulates Metabolic Genes / Translocation of SLC2A4 (GLUT4) to the plasma membrane / lipopolysaccharide binding / Hsp90 protein binding / negative regulation of ERK1 and ERK2 cascade / cellular response to type II interferon / Interferon gamma signaling / intrinsic apoptotic signaling pathway in response to DNA damage / cellular response to tumor necrosis factor / GDP binding / intracellular protein localization / actin cytoskeleton / regulation of protein localization / actin binding / G protein activity / positive regulation of cell growth / cytoplasmic vesicle / defense response to virus / 加水分解酵素; 酸無水物に作用; GTPに作用・細胞または細胞小器官の運動に関与 / regulation of cell cycle / defense response to bacterium / cadherin binding / Golgi membrane / innate immune response / GTPase activity / protein kinase binding / GTP binding / enzyme binding / negative regulation of transcription by RNA polymerase II / Golgi apparatus / signal transduction / protein homodimerization activity / extracellular space / extracellular exosome / extracellular region / identical protein binding / nucleus / plasma membrane / cytoplasm / cytosol 類似検索 - 分子機能 | ||||||||||||||||||||||||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||||||||||||||||||||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 5.12 Å | ||||||||||||||||||||||||||||||
 データ登録者 データ登録者 | Pfleiderer MM / Liu X / Fisch D / Anastasakou E / Frickel EM / Galej WP | ||||||||||||||||||||||||||||||
| 資金援助 |  ドイツ, ドイツ,  フランス, European Union, フランス, European Union,  英国, 9件 英国, 9件
| ||||||||||||||||||||||||||||||
 引用 引用 |  ジャーナル: Science / 年: 2023 ジャーナル: Science / 年: 2023タイトル: PIM1 controls GBP1 activity to limit self-damage and to guard against pathogen infection. 著者: Daniel Fisch / Moritz M Pfleiderer / Eleni Anastasakou / Gillian M Mackie / Fabian Wendt / Xiangyang Liu / Barbara Clough / Samuel Lara-Reyna / Vesela Encheva / Ambrosius P Snijders / ...著者: Daniel Fisch / Moritz M Pfleiderer / Eleni Anastasakou / Gillian M Mackie / Fabian Wendt / Xiangyang Liu / Barbara Clough / Samuel Lara-Reyna / Vesela Encheva / Ambrosius P Snijders / Hironori Bando / Masahiro Yamamoto / Andrew D Beggs / Jason Mercer / Avinash R Shenoy / Bernd Wollscheid / Kendle M Maslowski / Wojtek P Galej / Eva-Maria Frickel /      要旨: Disruption of cellular activities by pathogen virulence factors can trigger innate immune responses. Interferon-γ (IFN-γ)-inducible antimicrobial factors, such as the guanylate binding proteins ...Disruption of cellular activities by pathogen virulence factors can trigger innate immune responses. Interferon-γ (IFN-γ)-inducible antimicrobial factors, such as the guanylate binding proteins (GBPs), promote cell-intrinsic defense by attacking intracellular pathogens and by inducing programmed cell death. Working in human macrophages, we discovered that GBP1 expression in the absence of IFN-γ killed the cells and induced Golgi fragmentation. IFN-γ exposure improved macrophage survival through the activity of the kinase PIM1. PIM1 phosphorylated GBP1, leading to its sequestration by 14-3-3σ, which thereby prevented GBP1 membrane association. During infection, the virulence protein TgIST interfered with IFN-γ signaling and depleted PIM1, thereby increasing GBP1 activity. Although infected cells can restrain pathogens in a GBP1-dependent manner, this mechanism can protect uninfected bystander cells. Thus, PIM1 can provide a bait for pathogen virulence factors, guarding the integrity of IFN-γ signaling. | ||||||||||||||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_18149.map.gz emd_18149.map.gz | 401.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-18149-v30.xml emd-18149-v30.xml emd-18149.xml emd-18149.xml | 20 KB 20 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_18149.png emd_18149.png | 90.9 KB | ||
| Filedesc metadata |  emd-18149.cif.gz emd-18149.cif.gz | 6.7 KB | ||
| その他 |  emd_18149_half_map_1.map.gz emd_18149_half_map_1.map.gz emd_18149_half_map_2.map.gz emd_18149_half_map_2.map.gz | 763.8 MB 763.8 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-18149 http://ftp.pdbj.org/pub/emdb/structures/EMD-18149 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-18149 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-18149 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_18149_validation.pdf.gz emd_18149_validation.pdf.gz | 937 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_18149_full_validation.pdf.gz emd_18149_full_validation.pdf.gz | 936.5 KB | 表示 | |
| XML形式データ |  emd_18149_validation.xml.gz emd_18149_validation.xml.gz | 21.2 KB | 表示 | |
| CIF形式データ |  emd_18149_validation.cif.gz emd_18149_validation.cif.gz | 25.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18149 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18149 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18149 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18149 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8q4lMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_18149.map.gz / 形式: CCP4 / 大きさ: 824 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_18149.map.gz / 形式: CCP4 / 大きさ: 824 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.638 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #2
| ファイル | emd_18149_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
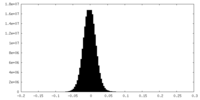
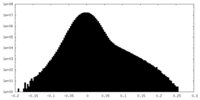
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_18149_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
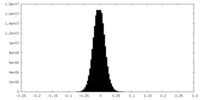
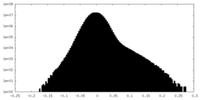
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : GBP1 bound by 14-3-3sigma
| 全体 | 名称: GBP1 bound by 14-3-3sigma |
|---|---|
| 要素 |
|
-超分子 #1: GBP1 bound by 14-3-3sigma
| 超分子 | 名称: GBP1 bound by 14-3-3sigma / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 120 KDa |
-分子 #1: Guanylate-binding protein 1
| 分子 | 名称: Guanylate-binding protein 1 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 66.275539 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MTGPMCLIEN TNGRLMANPE ALKILSAITQ PMVVVAIVGL YRTGKSYLMN KLAGKKKGFS LGSTVQSHTK GIWMWCVPHP KKPGHILVL LDTEGLGDVE KGDNQNDSWI FALAVLLSST FVYNSIGTIN QQAMDQLYYV TELTHRIRSK SSPDENENEV E DSADFVSF ...文字列: MTGPMCLIEN TNGRLMANPE ALKILSAITQ PMVVVAIVGL YRTGKSYLMN KLAGKKKGFS LGSTVQSHTK GIWMWCVPHP KKPGHILVL LDTEGLGDVE KGDNQNDSWI FALAVLLSST FVYNSIGTIN QQAMDQLYYV TELTHRIRSK SSPDENENEV E DSADFVSF FPDFVWTLRD FSLDLEADGQ PLTPDEYLTY SLKLKKGTSQ KDETFNLPRL CIRKFFPKKK CFVFDRPVHR RK LAQLEKL QDEELDPEFV QQVADFCSYI FSNSKTKTLS GGIQVNGPRL ESLVLTYVNA ISSGDLPCME NAVLALAQIE NSA AVQKAI AHYEQQMGQK VQLPTETLQE LLDLHRDSER EAIEVFIRSS FKDVDHLFQK ELAAQLEKKR DDFCKQNQEA SSDR CSALL QVIFSPLEEE VKAGIYSKPG GYRLFVQKLQ DLKKKYYEEP RKGIQAEEIL QTYLKSKESM TDAILQTDQT LTEKE KEIE VERVKAESAQ ASAKMLQEMQ RKNEQMMEQK ERSYQEHLKQ LTEKMENDRV QLLKEQERTL ALKLQEQEQL LKEGFQ KES RIMKNEIQDL QTKM UniProtKB: Guanylate-binding protein 1 |
-分子 #2: 14-3-3 protein sigma
| 分子 | 名称: 14-3-3 protein sigma / タイプ: protein_or_peptide / ID: 2 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 26.139461 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MERASLIQKA KLAEQAERYE DMAAFMKGAV EKGEELSCEE RNLLSVAYKN VVGGQRAAWR VLSSIEQKSN EEGSEEKGPE VREYREKVE TELQGVCDTV LGLLDSHLIK EAGDAESRVF YLKMKGDYYR YLAEVATGDD KKRIIDSARS AYQEAMDISK K EMPPTNPI ...文字列: MERASLIQKA KLAEQAERYE DMAAFMKGAV EKGEELSCEE RNLLSVAYKN VVGGQRAAWR VLSSIEQKSN EEGSEEKGPE VREYREKVE TELQGVCDTV LGLLDSHLIK EAGDAESRVF YLKMKGDYYR YLAEVATGDD KKRIIDSARS AYQEAMDISK K EMPPTNPI RLGLALNFSV FHYEIANSPE EAISLAKTTF DEAMADLHTL SEDSYKDSTL IMQLLRDNLT LWT UniProtKB: 14-3-3 protein sigma |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.25 mg/mL | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.8 構成要素:
詳細: 150 mM KCl, 20 mM HEPES-KOH pH 7.8 | |||||||||
| グリッド | 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 20 sec. / 前処理 - 気圧: 30.0 kPa 詳細: Glow discharged for 20 seconds at 25 mA and 0.3 bar using a Pelco EasyGlow device. | |||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 QUANTUM (4k x 4k) 検出モード: COUNTING / 撮影したグリッド数: 1 / 実像数: 14974 / 平均露光時間: 8.0 sec. / 平均電子線量: 65.5 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.0 µm |
| 試料ステージ | ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー














 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)