+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
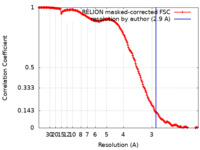
| タイトル | Inward-facing, open1 proteoliposome complex I at 2.9 A. Initially purified in LMNG. | |||||||||
 マップデータ マップデータ | Locally sharpened consensus map | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Complex I / Oxidoreductase / Proteoliposomes / Membrane-bound / metabolism / MEMBRANE PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報Complex I biogenesis / Mitochondrial protein import / RHOG GTPase cycle / ubiquinone biosynthetic process / Respiratory electron transport / cellular response to oxygen levels / mitochondrial large ribosomal subunit binding / gliogenesis / neural precursor cell proliferation / [2Fe-2S] cluster assembly ...Complex I biogenesis / Mitochondrial protein import / RHOG GTPase cycle / ubiquinone biosynthetic process / Respiratory electron transport / cellular response to oxygen levels / mitochondrial large ribosomal subunit binding / gliogenesis / neural precursor cell proliferation / [2Fe-2S] cluster assembly / oxygen sensor activity / Neutrophil degranulation / NADH dehydrogenase activity / Mitochondrial protein degradation / acyl binding / mitochondrial ATP synthesis coupled electron transport / ubiquinone binding / acyl carrier activity / electron transport coupled proton transport / NADH:ubiquinone reductase (H+-translocating) / mitochondrial respiratory chain complex I assembly / mitochondrial electron transport, NADH to ubiquinone / respiratory chain complex I / response to cAMP / NADH dehydrogenase (ubiquinone) activity / quinone binding / ATP synthesis coupled electron transport / neurogenesis / reactive oxygen species metabolic process / aerobic respiration / fatty acid binding / respiratory electron transport chain / electron transport chain / circadian rhythm / mitochondrial membrane / brain development / mitochondrial intermembrane space / 2 iron, 2 sulfur cluster binding / NAD binding / fatty acid biosynthetic process / FMN binding / 4 iron, 4 sulfur cluster binding / oxidoreductase activity / mitochondrial inner membrane / mitochondrial matrix / negative regulation of DNA-templated transcription / apoptotic process / protein-containing complex binding / mitochondrion / nucleoplasm / metal ion binding / cytoplasm 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.9 Å | |||||||||
 データ登録者 データ登録者 | Grba DN / Hirst J | |||||||||
| 資金援助 |  英国, 2件 英国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Science / 年: 2024 ジャーナル: Science / 年: 2024タイトル: Molecular mechanism of the ischemia-induced regulatory switch in mammalian complex I. 著者: Daniel N Grba / John J Wright / Zhan Yin / William Fisher / Judy Hirst /  要旨: Respiratory complex I is an efficient driver for oxidative phosphorylation in mammalian mitochondria, but its uncontrolled catalysis under challenging conditions leads to oxidative stress and ...Respiratory complex I is an efficient driver for oxidative phosphorylation in mammalian mitochondria, but its uncontrolled catalysis under challenging conditions leads to oxidative stress and cellular damage. Ischemic conditions switch complex I from rapid, reversible catalysis into a dormant state that protects upon reoxygenation, but the molecular basis for the switch is unknown. We combined precise biochemical definition of complex I catalysis with high-resolution cryo-electron microscopy structures in the phospholipid bilayer of coupled vesicles to reveal the mechanism of the transition into the dormant state, modulated by membrane interactions. By implementing a versatile membrane system to unite structure and function, attributing catalytic and regulatory properties to specific structural states, we define how a conformational switch in complex I controls its physiological roles. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_18140.map.gz emd_18140.map.gz | 276.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-18140-v30.xml emd-18140-v30.xml emd-18140.xml emd-18140.xml | 74.6 KB 74.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_18140_fsc.xml emd_18140_fsc.xml | 15.9 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_18140.png emd_18140.png | 120.9 KB | ||
| マスクデータ |  emd_18140_msk_1.map emd_18140_msk_1.map | 347.6 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-18140.cif.gz emd-18140.cif.gz | 15.2 KB | ||
| その他 |  emd_18140_additional_1.map.gz emd_18140_additional_1.map.gz emd_18140_additional_2.map.gz emd_18140_additional_2.map.gz emd_18140_half_map_1.map.gz emd_18140_half_map_1.map.gz emd_18140_half_map_2.map.gz emd_18140_half_map_2.map.gz | 278.7 MB 20.7 MB 279.8 MB 279.8 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-18140 http://ftp.pdbj.org/pub/emdb/structures/EMD-18140 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-18140 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-18140 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_18140_validation.pdf.gz emd_18140_validation.pdf.gz | 1 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_18140_full_validation.pdf.gz emd_18140_full_validation.pdf.gz | 1 MB | 表示 | |
| XML形式データ |  emd_18140_validation.xml.gz emd_18140_validation.xml.gz | 23.4 KB | 表示 | |
| CIF形式データ |  emd_18140_validation.cif.gz emd_18140_validation.cif.gz | 31.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18140 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18140 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18140 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18140 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8q47MC  8q0aC  8q0fC  8q0jC  8q0mC  8q0oC  8q0qC  8q1pC  8q1uC  8q1yC  8q25C  8q45C  8q46C  8q48C  8q49C  8q4aC C: 同じ文献を引用 ( M: このマップから作成された原子モデル |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_18140.map.gz / 形式: CCP4 / 大きさ: 347.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_18140.map.gz / 形式: CCP4 / 大きさ: 347.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Locally sharpened consensus map | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.066 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_18140_msk_1.map emd_18140_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: Unfiltered consensus map
| ファイル | emd_18140_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Unfiltered consensus map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: Globally sharpened/postprocessed consensus map
| ファイル | emd_18140_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Globally sharpened/postprocessed consensus map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: half map 1
| ファイル | emd_18140_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: half map 2
| ファイル | emd_18140_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
+全体 : Complex I from Bos taurus reconstituted into proteoliposome.
+超分子 #1: Complex I from Bos taurus reconstituted into proteoliposome.
+分子 #1: NADH-ubiquinone oxidoreductase chain 3
+分子 #2: NADH dehydrogenase [ubiquinone] iron-sulfur protein 7, mitochondrial
+分子 #3: NADH dehydrogenase [ubiquinone] iron-sulfur protein 3, mitochondrial
+分子 #4: NADH dehydrogenase [ubiquinone] iron-sulfur protein 2, mitochondrial
+分子 #5: NADH dehydrogenase [ubiquinone] flavoprotein 2, mitochondrial
+分子 #6: NADH dehydrogenase [ubiquinone] flavoprotein 1, mitochondrial
+分子 #7: NADH-ubiquinone oxidoreductase 75 kDa subunit, mitochondrial
+分子 #8: NADH-ubiquinone oxidoreductase chain 1
+分子 #9: NADH dehydrogenase [ubiquinone] iron-sulfur protein 8, mitochondrial
+分子 #10: NADH-ubiquinone oxidoreductase chain 6
+分子 #11: NADH-ubiquinone oxidoreductase chain 4L
+分子 #12: NADH-ubiquinone oxidoreductase chain 5
+分子 #13: NADH-ubiquinone oxidoreductase chain 4
+分子 #14: NADH-ubiquinone oxidoreductase chain 2
+分子 #15: NADH dehydrogenase [ubiquinone] 1 alpha subcomplex subunit 10, mi...
+分子 #16: NADH dehydrogenase [ubiquinone] 1 alpha subcomplex subunit 9, mit...
+分子 #17: NADH dehydrogenase [ubiquinone] iron-sulfur protein 4, mitochondrial
+分子 #18: NADH dehydrogenase [ubiquinone] iron-sulfur protein 6, mitochondrial
+分子 #19: NADH dehydrogenase [ubiquinone] 1 alpha subcomplex subunit 2
+分子 #20: Acyl carrier protein, mitochondrial
+分子 #21: NADH dehydrogenase [ubiquinone] 1 alpha subcomplex subunit 5
+分子 #22: NADH dehydrogenase [ubiquinone] 1 alpha subcomplex subunit 6
+分子 #23: NADH dehydrogenase [ubiquinone] 1 alpha subcomplex subunit 8
+分子 #24: NADH dehydrogenase [ubiquinone] 1 alpha subcomplex subunit 11
+分子 #25: NADH dehydrogenase [ubiquinone] 1 alpha subcomplex subunit 13
+分子 #26: NADH dehydrogenase [ubiquinone] 1 alpha subcomplex subunit 1
+分子 #27: NADH dehydrogenase [ubiquinone] 1 alpha subcomplex subunit 3
+分子 #28: NADH dehydrogenase [ubiquinone] 1 subunit C1, mitochondrial
+分子 #29: NADH dehydrogenase [ubiquinone] 1 subunit C2
+分子 #30: NADH dehydrogenase [ubiquinone] iron-sulfur protein 5
+分子 #31: NADH dehydrogenase [ubiquinone] 1 beta subcomplex subunit 1
+分子 #32: NADH dehydrogenase [ubiquinone] 1 beta subcomplex subunit 11, mit...
+分子 #33: NADH dehydrogenase [ubiquinone] 1 beta subcomplex subunit 5, mito...
+分子 #34: NADH dehydrogenase [ubiquinone] 1 beta subcomplex subunit 6
+分子 #35: NADH dehydrogenase [ubiquinone] 1 beta subcomplex subunit 2, mito...
+分子 #36: NADH dehydrogenase [ubiquinone] 1 beta subcomplex subunit 3
+分子 #37: NADH dehydrogenase [ubiquinone] 1 beta subcomplex subunit 8, mito...
+分子 #38: NADH dehydrogenase [ubiquinone] 1 beta subcomplex subunit 4
+分子 #39: NADH dehydrogenase [ubiquinone] 1 beta subcomplex subunit 9
+分子 #40: NADH dehydrogenase [ubiquinone] 1 beta subcomplex subunit 7
+分子 #41: NADH dehydrogenase [ubiquinone] 1 beta subcomplex subunit 10
+分子 #42: NADH dehydrogenase [ubiquinone] 1 alpha subcomplex subunit 12
+分子 #43: NADH dehydrogenase [ubiquinone] 1 alpha subcomplex subunit 7
+分子 #44: NADH dehydrogenase [ubiquinone] flavoprotein 3, mitochondrial
+分子 #45: 1,2-DIACYL-SN-GLYCERO-3-PHOSPHOCHOLINE
+分子 #46: 1,2-Distearoyl-sn-glycerophosphoethanolamine
+分子 #47: IRON/SULFUR CLUSTER
+分子 #48: DIUNDECYL PHOSPHATIDYL CHOLINE
+分子 #49: FE2/S2 (INORGANIC) CLUSTER
+分子 #50: FLAVIN MONONUCLEOTIDE
+分子 #51: POTASSIUM ION
+分子 #52: UBIQUINONE-10
+分子 #53: CARDIOLIPIN
+分子 #54: ZINC ION
+分子 #55: 2'-DEOXYGUANOSINE-5'-TRIPHOSPHATE
+分子 #56: MAGNESIUM ION
+分子 #57: NADPH DIHYDRO-NICOTINAMIDE-ADENINE-DINUCLEOTIDE PHOSPHATE
+分子 #58: ~{S}-[2-[3-[[(2~{R})-3,3-dimethyl-2-oxidanyl-4-phosphonooxy-butan...
+分子 #59: CHOLIC ACID
+分子 #60: MYRISTIC ACID
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.2 mg/mL | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
| |||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 95 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK III 詳細: Sample was incubated on grid for 30 s before plunge freezing. | |||||||||
| 詳細 | Complex I proteoliposomes on a graphene-oxide coated gold grid with even liposome distribution. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) デジタル化 - サイズ - 横: 5760 pixel / デジタル化 - サイズ - 縦: 4092 pixel / 撮影したグリッド数: 1 / 実像数: 27014 / 平均露光時間: 2.67 sec. / 平均電子線量: 45.4 e/Å2 詳細: The data were collected with aberration-free image shift (AFIS). |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm 最大 デフォーカス(公称値): 2.3000000000000003 µm 最小 デフォーカス(公称値): 0.9 µm / 倍率(公称値): 81000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー


































 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)