+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of the methanogenic Na+ translocating N5-methyl-H4MPT:CoM methyltransferase complex | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | methanogenesis / tetrahydromethanopterin / coenzyme M / vitamin B12 / Na+ transport / TRANSFERASE | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報tetrahydromethanopterin S-methyltransferase / tetrahydromethanopterin S-methyltransferase activity / methyltransferase complex / methanogenesis, from carbon dioxide / vesicle membrane / cobalt ion binding / one-carbon metabolic process / methylation / plasma membrane / cytoplasm 類似検索 - 分子機能 | |||||||||
| 生物種 |   Methanothermobacter marburgensis (古細菌) Methanothermobacter marburgensis (古細菌) | |||||||||
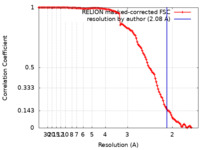
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.08 Å | |||||||||
 データ登録者 データ登録者 | Aziz I / Vonck J / Ermler U | |||||||||
| 資金援助 |  ドイツ, 1件 ドイツ, 1件
| |||||||||
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2024 ジャーナル: Proc Natl Acad Sci U S A / 年: 2024タイトル: Structural and mechanistic basis of the central energy-converting methyltransferase complex of methanogenesis. 著者: Iram Aziz / Kanwal Kayastha / Susann Kaltwasser / Janet Vonck / Sonja Welsch / Bonnie J Murphy / Jörg Kahnt / Di Wu / Tristan Wagner / Seigo Shima / Ulrich Ermler /  要旨: Methanogenic archaea inhabiting anaerobic environments play a crucial role in the global biogeochemical material cycle. The most universal electrogenic reaction of their methane-producing energy ...Methanogenic archaea inhabiting anaerobic environments play a crucial role in the global biogeochemical material cycle. The most universal electrogenic reaction of their methane-producing energy metabolism is catalyzed by -methyl-tetrahydromethanopterin: coenzyme M methyltransferase (MtrABCDEFGH), which couples the vectorial Na transport with a methyl transfer between the one-carbon carriers tetrahydromethanopterin and coenzyme M via a vitamin B derivative (cobamide) as prosthetic group. We present the 2.08 Å cryo-EM structure of Mtr(ABCDEFG) composed of the central Mtr(ABFG) stalk symmetrically flanked by three membrane-spanning MtrCDE globes. Tetraether glycolipids visible in the map fill gaps inside the multisubunit complex. Putative coenzyme M and Na were identified inside or in a side-pocket of a cytoplasmic cavity formed within MtrCDE. Its bottom marks the gate of the transmembrane pore occluded in the cryo-EM map. By integrating Alphafold2 information, functionally competent MtrA-MtrH and MtrA-MtrCDE subcomplexes could be modeled and thus the methyl-tetrahydromethanopterin demethylation and coenzyme M methylation half-reactions structurally described. Methyl-transfer-driven Na transport is proposed to be based on a strong and weak complex between MtrCDE and MtrA carrying vitamin B, the latter being placed at the entrance of the cytoplasmic MtrCDE cavity. Hypothetically, strongly attached methyl-cob(III)amide (His-on) carrying MtrA induces an inward-facing conformation, Na flux into the membrane protein center and finally coenzyme M methylation while the generated loosely attached (or detached) MtrA carrying cob(I)amide (His-off) induces an outward-facing conformation and an extracellular Na outflux. Methyl-cob(III)amide (His-on) is regenerated in the distant active site of the methyl-tetrahydromethanopterin binding MtrH implicating a large-scale shuttling movement of the vitamin B-carrying domain. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_18135.map.gz emd_18135.map.gz | 98.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-18135-v30.xml emd-18135-v30.xml emd-18135.xml emd-18135.xml | 21.9 KB 21.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_18135_fsc.xml emd_18135_fsc.xml | 11.3 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_18135.png emd_18135.png | 119.9 KB | ||
| マスクデータ |  emd_18135_msk_1.map emd_18135_msk_1.map | 125 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-18135.cif.gz emd-18135.cif.gz | 6.6 KB | ||
| その他 |  emd_18135_half_map_1.map.gz emd_18135_half_map_1.map.gz emd_18135_half_map_2.map.gz emd_18135_half_map_2.map.gz | 98.4 MB 98.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-18135 http://ftp.pdbj.org/pub/emdb/structures/EMD-18135 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-18135 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-18135 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_18135_validation.pdf.gz emd_18135_validation.pdf.gz | 955.7 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_18135_full_validation.pdf.gz emd_18135_full_validation.pdf.gz | 955.2 KB | 表示 | |
| XML形式データ |  emd_18135_validation.xml.gz emd_18135_validation.xml.gz | 18.6 KB | 表示 | |
| CIF形式データ |  emd_18135_validation.cif.gz emd_18135_validation.cif.gz | 24.6 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18135 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18135 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18135 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18135 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8q3vMC  8q54C M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_18135.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_18135.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.837 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_18135_msk_1.map emd_18135_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_18135_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_18135_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
+全体 : Methyl-H4MPT:CoM methyltransferase
+超分子 #1: Methyl-H4MPT:CoM methyltransferase
+分子 #1: Tetrahydromethanopterin S-methyltransferase subunit A 1
+分子 #2: Tetrahydromethanopterin S-methyltransferase subunit B
+分子 #3: Tetrahydromethanopterin S-methyltransferase subunit C
+分子 #4: Tetrahydromethanopterin S-methyltransferase subunit D
+分子 #5: Tetrahydromethanopterin S-methyltransferase subunit E
+分子 #6: Tetrahydromethanopterin S-methyltransferase subunit F
+分子 #7: Tetrahydromethanopterin S-methyltransferase subunit G
+分子 #8: MAGNESIUM ION
+分子 #9: [(2~{S},7~{R},11~{R},15~{S},19~{S},22~{S},26~{S},30~{R},34~{R},39...
+分子 #10: SODIUM ION
+分子 #11: water
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 73.9 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.1 µm / 最小 デフォーカス(公称値): 1.2 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー





 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)