登録情報 データベース : EMDB / ID : EMD-18062タイトル III2-IV1 respiratory supercomplex from S. pombe 複合体 : III2-IV1 respiratory supercomplexリガンド : x 13種 / / 機能・相同性 分子機能 ドメイン・相同性 構成要素
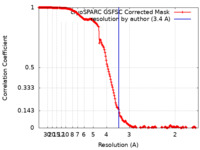
/ / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / 生物種 Schizosaccharomyces pombe (分裂酵母)手法 / / 解像度 : 3.4 Å Moe A / Brzezinski P 資金援助 Organization Grant number 国 Swedish Research Council
ジャーナル : Proc Natl Acad Sci U S A / 年 : 2023タイトル : Structure and function of the III-IV-cyt supercomplex.著者 : Agnes Moe / Anna-Roza Dimogkioka / Doron Rapaport / Linda Näsvik Öjemyr / Peter Brzezinski / 要旨 : The respiratory chain in aerobic organisms is composed of a number of membrane-bound protein complexes that link electron transfer to proton translocation across the membrane. In mitochondria, the ... The respiratory chain in aerobic organisms is composed of a number of membrane-bound protein complexes that link electron transfer to proton translocation across the membrane. In mitochondria, the final electron acceptor, complex IV (CIV), receives electrons from dimeric complex III (CIII), via a mobile electron carrier, cytochrome . In the present study, we isolated the CIIICIV supercomplex from the fission yeast and determined its structure with bound cyt. using single-particle electron cryomicroscopy. A respiratory supercomplex factor 2 was found to be bound at CIV distally positioned in the supercomplex. In addition to the redox-active metal sites, we found a metal ion, presumably Zn, coordinated in the CIII subunit Cor1, which is encoded by the same gene () as the mitochondrial-processing peptidase subunit β. Our data show that the isolated CIIICIV supercomplex displays proteolytic activity suggesting a dual role of CIII in . As in the supercomplex from , subunit Cox5 of CIV faces towards one CIII monomer, but in the two complexes are rotated relative to each other by ~45°. This orientation yields equal distances between the cyt. binding sites at CIV and at each of the two CIII monomers. The structure shows cyt. bound at four positions, but only along one of the two symmetrical branches. Overall, this combined structural and functional study reveals the integration of peptidase activity with the CIII respiratory system and indicates a two-dimensional cyt. diffusion mechanism within the CIII-CIV supercomplex. 履歴 登録 2023年7月31日 - ヘッダ(付随情報) 公開 2023年11月15日 - マップ公開 2023年11月15日 - 更新 2025年10月1日 - 現状 2025年10月1日 処理サイト : PDBe / 状態 : 公開
すべて表示 表示を減らす
 データを開く
データを開く 基本情報
基本情報
 マップデータ
マップデータ 試料
試料 キーワード
キーワード 機能・相同性情報
機能・相同性情報
 データ登録者
データ登録者 スウェーデン, 1件
スウェーデン, 1件  引用
引用 ジャーナル: Proc Natl Acad Sci U S A / 年: 2023
ジャーナル: Proc Natl Acad Sci U S A / 年: 2023

 構造の表示
構造の表示 ダウンロードとリンク
ダウンロードとリンク emd_18062.map.gz
emd_18062.map.gz EMDBマップデータ形式
EMDBマップデータ形式 emd-18062-v30.xml
emd-18062-v30.xml emd-18062.xml
emd-18062.xml EMDBヘッダ
EMDBヘッダ emd_18062_fsc.xml
emd_18062_fsc.xml FSCデータファイル
FSCデータファイル emd_18062.png
emd_18062.png emd-18062.cif.gz
emd-18062.cif.gz emd_18062_additional_1.map.gz
emd_18062_additional_1.map.gz emd_18062_additional_2.map.gz
emd_18062_additional_2.map.gz emd_18062_additional_3.map.gz
emd_18062_additional_3.map.gz emd_18062_additional_4.map.gz
emd_18062_additional_4.map.gz emd_18062_half_map_1.map.gz
emd_18062_half_map_1.map.gz emd_18062_half_map_2.map.gz
emd_18062_half_map_2.map.gz http://ftp.pdbj.org/pub/emdb/structures/EMD-18062
http://ftp.pdbj.org/pub/emdb/structures/EMD-18062 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-18062
ftp://ftp.pdbj.org/pub/emdb/structures/EMD-18062 emd_18062_validation.pdf.gz
emd_18062_validation.pdf.gz EMDB検証レポート
EMDB検証レポート emd_18062_full_validation.pdf.gz
emd_18062_full_validation.pdf.gz emd_18062_validation.xml.gz
emd_18062_validation.xml.gz emd_18062_validation.cif.gz
emd_18062_validation.cif.gz https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18062
https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18062 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18062
ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18062
 F&H 検索
F&H 検索 リンク
リンク EMDB (EBI/PDBe) /
EMDB (EBI/PDBe) /  EMDataResource
EMDataResource マップ
マップ ダウンロード / ファイル: emd_18062.map.gz / 形式: CCP4 / 大きさ: 512 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES)
ダウンロード / ファイル: emd_18062.map.gz / 形式: CCP4 / 大きさ: 512 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) 試料の構成要素
試料の構成要素 解析
解析 試料調製
試料調製 電子顕微鏡法
電子顕微鏡法 FIELD EMISSION GUN
FIELD EMISSION GUN
 ムービー
ムービー コントローラー
コントローラー













 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)