+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | 4.0 angstrom map of outward-facing MFS transporter MHAS2168, a homologue of M. tuberculosis Rv1410, in complex with a megabody | ||||||||||||
 マップデータ マップデータ | Membrane transporter MHAS2168, a homologue of M. tuberculosis Rv1410, in complex with megabody H2 consisting of nanobody H2 grafted to HopQ domain | ||||||||||||
 試料 試料 |
| ||||||||||||
 キーワード キーワード | Major Facilitator Superfamily transporter / megabody / outward-open conformation / triacylglyceride extraction / MEMBRANE PROTEIN | ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 | ||||||||||||
| 生物種 |  Mycolicibacterium hassiacum DSM 44199 (バクテリア) / Mycolicibacterium hassiacum DSM 44199 (バクテリア) /  | ||||||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.99 Å | ||||||||||||
 データ登録者 データ登録者 | Remm S / Gonda I / Seeger MA | ||||||||||||
| 資金援助 |  スイス, European Union, 3件 スイス, European Union, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2023 ジャーナル: Nat Commun / 年: 2023タイトル: Structural basis for triacylglyceride extraction from mycobacterial inner membrane by MFS transporter Rv1410. 著者: Sille Remm / Dario De Vecchis / Jendrik Schöppe / Cedric A J Hutter / Imre Gonda / Michael Hohl / Simon Newstead / Lars V Schäfer / Markus A Seeger /     要旨: Mycobacterium tuberculosis is protected from antibiotic therapy by a multi-layered hydrophobic cell envelope. Major facilitator superfamily (MFS) transporter Rv1410 and the periplasmic lipoprotein ...Mycobacterium tuberculosis is protected from antibiotic therapy by a multi-layered hydrophobic cell envelope. Major facilitator superfamily (MFS) transporter Rv1410 and the periplasmic lipoprotein LprG are involved in transport of triacylglycerides (TAGs) that seal the mycomembrane. Here, we report a 2.7 Å structure of a mycobacterial Rv1410 homologue, which adopts an outward-facing conformation and exhibits unusual transmembrane helix 11 and 12 extensions that protrude ~20 Å into the periplasm. A small, very hydrophobic cavity suitable for lipid transport is constricted by a functionally important ion-lock likely involved in proton coupling. Combining mutational analyses and MD simulations, we propose that TAGs are extracted from the core of the inner membrane into the central cavity via lateral clefts present in the inward-facing conformation. The functional role of the periplasmic helix extensions is to channel the extracted TAG into the lipid binding pocket of LprG. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_17787.map.gz emd_17787.map.gz | 327.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-17787-v30.xml emd-17787-v30.xml emd-17787.xml emd-17787.xml | 19.9 KB 19.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_17787.png emd_17787.png | 60.2 KB | ||
| マスクデータ |  emd_17787_msk_1.map emd_17787_msk_1.map | 347.6 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-17787.cif.gz emd-17787.cif.gz | 6.4 KB | ||
| その他 |  emd_17787_half_map_1.map.gz emd_17787_half_map_1.map.gz emd_17787_half_map_2.map.gz emd_17787_half_map_2.map.gz | 323.1 MB 323.1 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-17787 http://ftp.pdbj.org/pub/emdb/structures/EMD-17787 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-17787 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-17787 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_17787_validation.pdf.gz emd_17787_validation.pdf.gz | 857.5 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_17787_full_validation.pdf.gz emd_17787_full_validation.pdf.gz | 857.1 KB | 表示 | |
| XML形式データ |  emd_17787_validation.xml.gz emd_17787_validation.xml.gz | 17.1 KB | 表示 | |
| CIF形式データ |  emd_17787_validation.cif.gz emd_17787_validation.cif.gz | 20.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-17787 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-17787 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-17787 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-17787 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8pnlC C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_17787.map.gz / 形式: CCP4 / 大きさ: 347.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_17787.map.gz / 形式: CCP4 / 大きさ: 347.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Membrane transporter MHAS2168, a homologue of M. tuberculosis Rv1410, in complex with megabody H2 consisting of nanobody H2 grafted to HopQ domain | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.65 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_17787_msk_1.map emd_17787_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
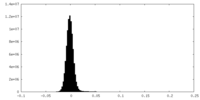
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_17787_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
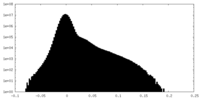
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_17787_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : MFS transporter MHAS2168 in outward-open conformation in complex ...
| 全体 | 名称: MFS transporter MHAS2168 in outward-open conformation in complex with Megabody_H2 |
|---|---|
| 要素 |
|
-超分子 #1: MFS transporter MHAS2168 in outward-open conformation in complex ...
| 超分子 | 名称: MFS transporter MHAS2168 in outward-open conformation in complex with Megabody_H2 タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Mycolicibacterium hassiacum DSM 44199 (バクテリア) Mycolicibacterium hassiacum DSM 44199 (バクテリア) |
-分子 #1: MFS transporter MHAS2168, a homologue of M. tuberculosis Rv1410c,...
| 分子 | 名称: MFS transporter MHAS2168, a homologue of M. tuberculosis Rv1410c, in outward-open conformation タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Mycolicibacterium hassiacum DSM 44199 (バクテリア) Mycolicibacterium hassiacum DSM 44199 (バクテリア) |
| 組換発現 | 生物種:  Mycolicibacterium smegmatis MC2 155 (バクテリア) Mycolicibacterium smegmatis MC2 155 (バクテリア) |
| 配列 | 文字列: MSAFPQTPNR LIRPRRTSRG IAISAGGLAV LLGALDTYVV VSIVTDIMRD VGIAVNQIQR VTPIITGYLL GYIAAMPLLG RASDRFGRKL LIQISLAGFA LGSVITALAT NLDVLVAGRV IQGAASGALL PVTLALAADL WATHKRAAVL GGVGAAQELG AVLGPIYGIF ...文字列: MSAFPQTPNR LIRPRRTSRG IAISAGGLAV LLGALDTYVV VSIVTDIMRD VGIAVNQIQR VTPIITGYLL GYIAAMPLLG RASDRFGRKL LIQISLAGFA LGSVITALAT NLDVLVAGRV IQGAASGALL PVTLALAADL WATHKRAAVL GGVGAAQELG AVLGPIYGIF VVWLFHHWQA VFWVNVPLAL IAMVLIHISL PPRVRTEEPQ RVDVTGGLLL ALALGLATIG LYNAEPDGKQ VLPEYGPPLI IGAVIAAVAF LVWERFARTR LLDPAGVRFR PFLIALLVSL VTGGALMVTL VNVELFGQGV LGLDQDEAVF LLARFLIALP VGALLGGWIA TRVGDRAVTA VGLLIAAGGF YLIAQWPADV LESRHDLGFV SLPTLDTDLA IAGFGLGLVI APLTSAALRV VPAAQHGIAS AAVVVARMIG MLIGIAALSA WGLYRFNQYL KEQLAALPPA PADFPGGQMA GQMMRLRTAT VQAYVLQYGE IFAITAGLCV FGAVLGLFIA GRREHAEESA DAVDGVSNAR DRAPSAALEV LFQ UniProtKB: Triacylglyceride transporter MHAS_02168/C731_2106 |
-分子 #2: Megabody_H2, derived from grafting alpaca-produced Nanobody_H2 on...
| 分子 | 名称: Megabody_H2, derived from grafting alpaca-produced Nanobody_H2 onto the HopQ adhesin domain of Helicobacter pylori タイプ: protein_or_peptide / ID: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  |
| 配列 | 文字列: QVQLQESGGG LVQTKTTTSV IDTTNDAQNL LTQAQTIVNT LKDYCPILIA KSSSSNGGTN NANTPSWQTA GGGKNSCATF GAEFSAASDM INNAQKIVQE TQQLSANQPK NITQPHNLNL NSPSSLTALA QKMLKNAQSQ AEILKLANQV ESDFNKLSSG HLKDYIGKCD ...文字列: QVQLQESGGG LVQTKTTTSV IDTTNDAQNL LTQAQTIVNT LKDYCPILIA KSSSSNGGTN NANTPSWQTA GGGKNSCATF GAEFSAASDM INNAQKIVQE TQQLSANQPK NITQPHNLNL NSPSSLTALA QKMLKNAQSQ AEILKLANQV ESDFNKLSSG HLKDYIGKCD ASAISSANMT MQNQKNNWGN GCAGVEETQS LLKTSAADFN NQTPQINQAQ NLANTLIQEL GNNPFRGGGK LSDTYEQLSR LLTNDNGTNS KTSAQAINQA VNNLNERAKT LAGGTTNSPA YQATLLALRS VLGLWNSMGY AVICGGYTKS PGENNQKDFH YTDENGNGTT INCGGSTNSN GTHSYNGTNT LKADKNVSLS IEQYEKIHEA YQILSKALKQ AGLAPLNSKG EKLEAHVTTS KYGSPGGSLR LSCADAGSIF NKFPMAWYRQ APGKERELVA RISSGGSTNY ADFVKGRFTI SRDNAKSTLY LQMNSLKPED TAMYYCARII NSASNIAYWG QGTRVTVSAA LEVLFQ |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 9.4 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.4 構成要素:
詳細: 20 mM Tris/HCl pH 7.4, 150 mM NaCl, 0.03% (w/v) beta-DDM | ||||||||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: GOLD / メッシュ: 200 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 45 sec. | ||||||||||||
| 凍結 | 凍結剤: ETHANE-PROPANE / チャンバー内湿度: 95 % / チャンバー内温度: 283.15 K / 装置: LEICA PLUNGER | ||||||||||||
| 詳細 | This sample was monodisperse |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 撮影したグリッド数: 3 / 実像数: 11713 / 平均電子線量: 66.54 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 130000 |
| 試料ステージ | ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー







 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)