+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | The ERAD misfolded glycoprotein checkpoint complex from Chaetomium thermophilum (EDEM:PDI heterodimer). | ||||||||||||
 マップデータ マップデータ | Cryosparc class, 150,000 particles, iteration 4 | ||||||||||||
 試料 試料 |
| ||||||||||||
 キーワード キーワード | mannosidase / disulfide isomerase / glycoprotein degradation / erad / misfolding / OXIDOREDUCTASE | ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報mannosyl-oligosaccharide 1,2-alpha-mannosidase activity / endoplasmic reticulum mannose trimming / protein disulfide-isomerase / endoplasmic reticulum quality control compartment / 加水分解酵素; 糖加水分解酵素; 配糖体結合加水分解酵素または糖加水分解酵素 / : / protein disulfide isomerase activity / protein-disulfide reductase activity / ERAD pathway / response to endoplasmic reticulum stress ...mannosyl-oligosaccharide 1,2-alpha-mannosidase activity / endoplasmic reticulum mannose trimming / protein disulfide-isomerase / endoplasmic reticulum quality control compartment / 加水分解酵素; 糖加水分解酵素; 配糖体結合加水分解酵素または糖加水分解酵素 / : / protein disulfide isomerase activity / protein-disulfide reductase activity / ERAD pathway / response to endoplasmic reticulum stress / bioluminescence / generation of precursor metabolites and energy / unfolded protein binding / protein folding / carbohydrate metabolic process / endoplasmic reticulum lumen / calcium ion binding / membrane 類似検索 - 分子機能 | ||||||||||||
| 生物種 |  Thermochaetoides thermophila (菌類) Thermochaetoides thermophila (菌類) | ||||||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.6 Å | ||||||||||||
 データ登録者 データ登録者 | Roversi P / Hitchman CJ / Lia A / Bayo Y / Gooptu B | ||||||||||||
| 資金援助 |  英国, 英国,  イタリア, 3件 イタリア, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: To Be Published ジャーナル: To Be Publishedタイトル: The ERAD misfolded glycoprotein checkpoint complex from Chaetomium thermophilum (EDEM:PDI heterodimer). 著者: Roversi P / Hitchman CJ / Lia A / Bayo Y | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_17749.map.gz emd_17749.map.gz | 51.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-17749-v30.xml emd-17749-v30.xml emd-17749.xml emd-17749.xml | 29 KB 29 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_17749.png emd_17749.png | 115.6 KB | ||
| Filedesc metadata |  emd-17749.cif.gz emd-17749.cif.gz | 9 KB | ||
| その他 |  emd_17749_half_map_1.map.gz emd_17749_half_map_1.map.gz emd_17749_half_map_2.map.gz emd_17749_half_map_2.map.gz | 95.6 MB 95.6 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-17749 http://ftp.pdbj.org/pub/emdb/structures/EMD-17749 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-17749 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-17749 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_17749_validation.pdf.gz emd_17749_validation.pdf.gz | 1.1 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_17749_full_validation.pdf.gz emd_17749_full_validation.pdf.gz | 1.1 MB | 表示 | |
| XML形式データ |  emd_17749_validation.xml.gz emd_17749_validation.xml.gz | 13.3 KB | 表示 | |
| CIF形式データ |  emd_17749_validation.cif.gz emd_17749_validation.cif.gz | 15.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-17749 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-17749 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-17749 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-17749 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8pkoMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_17749.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_17749.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Cryosparc class, 150,000 particles, iteration 4 | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.835 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: Cryosparc class, 150,000 particles, iteration 3
| ファイル | emd_17749_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Cryosparc class, 150,000 particles, iteration 3 | ||||||||||||
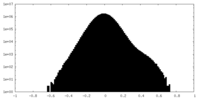
| 投影像・断面図 |
| ||||||||||||
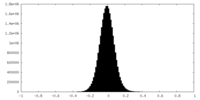
| 密度ヒストグラム |
-ハーフマップ: Cryosparc class, 150,000 particles, iteration 3
| ファイル | emd_17749_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Cryosparc class, 150,000 particles, iteration 3 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
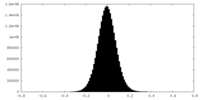
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
+全体 : Complex of Chaetomium thermophilum Endoplasmic reticulum degradat...
+超分子 #1: Complex of Chaetomium thermophilum Endoplasmic reticulum degradat...
+超分子 #2: Chaetomium thermophilum Endoplasmic reticulum degradation enhanci...
+超分子 #3: Endoplasmic reticulum degradation enhancing protein disulfide iso...
+分子 #1: Green fluorescent protein,alpha-1,2-Mannosidase
+分子 #2: Protein disulfide-isomerase
+分子 #5: CALCIUM ION
+分子 #6: THIOSULFATE
+分子 #7: 2-acetamido-2-deoxy-beta-D-glucopyranose
+分子 #8: alpha-D-mannopyranose
+分子 #9: water
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.02 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7 構成要素:
詳細: Micro SEC buffer: 150 mM NaCl, 20 mM MES pH 7.0, 1 mM CaCl2 | ||||||||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: GOLD / メッシュ: 300 / 支持フィルム - 材質: GRAPHENE OXIDE / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 180 sec. / 前処理 - 雰囲気: AIR / 前処理 - 気圧: 0.2 kPa 詳細: The Quantifoil Au300 R1.2/1.3 Holey Carbon grids were glow discharged using the EMS GloQube. For grids to be coated in GO, the Graphene Oxide setting was used (0.2m Bar, 40 mA for 180 seconds) ...詳細: The Quantifoil Au300 R1.2/1.3 Holey Carbon grids were glow discharged using the EMS GloQube. For grids to be coated in GO, the Graphene Oxide setting was used (0.2m Bar, 40 mA for 180 seconds). For GO coating, graphene oxide solution (Sigma) at 0.2 mg/ml was spun at 300 g for 30 s to remove large aggregates. 3 uL was added to the grids for 1 minute, before blotting with Whatman No1 filter paper and washing three times with 20 uL drops of ultrapure water. | ||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV 詳細: Blotting time 3 s, waiting time 30 s Sample volume 3 uL, blot force 10. | ||||||||||||
| 詳細 | 40 uL of a protein sample with an OD_280 of 0.88 were injected onto a Cytiva Superdex 200 Increase 3.2/300 size-exclusion chromatography column, equilibrated in 150 mM NaCl, 20 mM MES pH 7.0, 1 mM CaCl2, collecting 50 uL fractions. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 1.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 105000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー









 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)
































 Homo sapiens (ヒト)
Homo sapiens (ヒト)