+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Neisseria meningitidis Type IV pilus SB-DATDH variant bound to the C24 nanobody | |||||||||
 マップデータ マップデータ | Raw map of the SB-DATDH variant of T4P bund to the C24 nanobody | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Pilin / Extracellular / Adhesion / Aggregation / PROTEIN FIBRIL | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  Neisseria meningitidis 8013 (髄膜炎菌) / Neisseria meningitidis 8013 (髄膜炎菌) /  | |||||||||
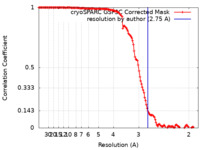
| 手法 | らせん対称体再構成法 / クライオ電子顕微鏡法 / 解像度: 2.75 Å | |||||||||
 データ登録者 データ登録者 | Fernandez-Martinez D / Dumenil G | |||||||||
| 資金援助 |  フランス, 1件 フランス, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2024 ジャーナル: Nat Commun / 年: 2024タイトル: Cryo-EM structures of type IV pili complexed with nanobodies reveal immune escape mechanisms. 著者: David Fernandez-Martinez / Youxin Kong / Sylvie Goussard / Agustin Zavala / Pauline Gastineau / Martial Rey / Gabriel Ayme / Julia Chamot-Rooke / Pierre Lafaye / Matthijn Vos / Ariel Mechaly ...著者: David Fernandez-Martinez / Youxin Kong / Sylvie Goussard / Agustin Zavala / Pauline Gastineau / Martial Rey / Gabriel Ayme / Julia Chamot-Rooke / Pierre Lafaye / Matthijn Vos / Ariel Mechaly / Guillaume Duménil /  要旨: Type IV pili (T4P) are prevalent, polymeric surface structures in pathogenic bacteria, making them ideal targets for effective vaccines. However, bacteria have evolved efficient strategies to evade ...Type IV pili (T4P) are prevalent, polymeric surface structures in pathogenic bacteria, making them ideal targets for effective vaccines. However, bacteria have evolved efficient strategies to evade type IV pili-directed antibody responses. Neisseria meningitidis are prototypical type IV pili-expressing Gram-negative bacteria responsible for life threatening sepsis and meningitis. This species has evolved several genetic strategies to modify the surface of its type IV pili, changing pilin subunit amino acid sequence, nature of glycosylation and phosphoforms, but how these modifications affect antibody binding at the structural level is still unknown. Here, to explore this question, we determine cryo-electron microscopy (cryo-EM) structures of pili of different sequence types with sufficiently high resolution to visualize posttranslational modifications. We then generate nanobodies directed against type IV pili which alter pilus function in vitro and in vivo. Cyro-EM in combination with molecular dynamics simulation of the nanobody-pilus complexes reveals how the different types of pili surface modifications alter nanobody binding. Our findings shed light on the impressive complementarity between the different strategies used by bacteria to avoid antibody binding. Importantly, we also show that structural information can be used to make informed modifications in nanobodies as countermeasures to these immune evasion mechanisms. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_17695.map.gz emd_17695.map.gz | 122.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-17695-v30.xml emd-17695-v30.xml emd-17695.xml emd-17695.xml | 18.3 KB 18.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_17695_fsc.xml emd_17695_fsc.xml | 13.2 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_17695.png emd_17695.png | 37.2 KB | ||
| マスクデータ |  emd_17695_msk_1.map emd_17695_msk_1.map | 244.1 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-17695.cif.gz emd-17695.cif.gz | 5.9 KB | ||
| その他 |  emd_17695_additional_1.map.gz emd_17695_additional_1.map.gz emd_17695_half_map_1.map.gz emd_17695_half_map_1.map.gz emd_17695_half_map_2.map.gz emd_17695_half_map_2.map.gz | 214.8 MB 226.3 MB 226.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-17695 http://ftp.pdbj.org/pub/emdb/structures/EMD-17695 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-17695 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-17695 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_17695_validation.pdf.gz emd_17695_validation.pdf.gz | 1.1 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_17695_full_validation.pdf.gz emd_17695_full_validation.pdf.gz | 1.1 MB | 表示 | |
| XML形式データ |  emd_17695_validation.xml.gz emd_17695_validation.xml.gz | 22.3 KB | 表示 | |
| CIF形式データ |  emd_17695_validation.cif.gz emd_17695_validation.cif.gz | 28.8 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-17695 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-17695 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-17695 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-17695 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_17695.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_17695.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Raw map of the SB-DATDH variant of T4P bund to the C24 nanobody | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.93 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_17695_msk_1.map emd_17695_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: DL sharpened map of the SB-DATDH variant of...
| ファイル | emd_17695_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | DL sharpened map of the SB-DATDH variant of T4P bund to the C24 nanobody | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map A
| ファイル | emd_17695_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map A | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map B
| ファイル | emd_17695_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map B | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Neisseria meningitidis PilE, SB-DATDH variant, bound to the C24 n...
| 全体 | 名称: Neisseria meningitidis PilE, SB-DATDH variant, bound to the C24 nanobody |
|---|---|
| 要素 |
|
-超分子 #1: Neisseria meningitidis PilE, SB-DATDH variant, bound to the C24 n...
| 超分子 | 名称: Neisseria meningitidis PilE, SB-DATDH variant, bound to the C24 nanobody タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#2 |
|---|---|
| 由来(天然) | 生物種:  Neisseria meningitidis 8013 (髄膜炎菌) Neisseria meningitidis 8013 (髄膜炎菌) |
-分子 #1: Pilin
| 分子 | 名称: Pilin / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Neisseria meningitidis 8013 (髄膜炎菌) Neisseria meningitidis 8013 (髄膜炎菌) |
| 分子量 | 理論値: 17.067182 KDa |
| 配列 | 文字列: FTLIELMIVI AIVGILAAVA LPAYQDYTAR AQVSEAILLA EGQKSAVTEY YLNHGEWPGD NSSAGVATSA DIKGKYVQSV TVANGVITA QMASSNVNNE IKSKKLSLWA KRQNGSVKWF CGQPVTRTTA TATDVAAANG KTDDKINTKH LPSTCRDDSS A S UniProtKB: Pilin |
-分子 #2: C24 nanobody
| 分子 | 名称: C24 nanobody / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 16.288223 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MAQLQLVESG GGLVQPGGSL RLSCASSGFR SDYYAIVWFR QAPGKEREGV SCISTSGKTT IYADSVKGRF TISRDNAKNT VYLQMNSLK PEDTAVYYCA ADFRGSRLSD VCSYSSMDYW GKGTLATVSS EPKTPKPQPA AALEHHHHHH |
-分子 #3: SN-GLYCEROL-3-PHOSPHATE
| 分子 | 名称: SN-GLYCEROL-3-PHOSPHATE / タイプ: ligand / ID: 3 / コピー数: 1 / 式: G3P |
|---|---|
| 分子量 | 理論値: 172.074 Da |
| Chemical component information |  ChemComp-G3P: |
-分子 #4: 2,4-bisacetamido-2,4,6-trideoxy-beta-D-glucopyranose
| 分子 | 名称: 2,4-bisacetamido-2,4,6-trideoxy-beta-D-glucopyranose タイプ: ligand / ID: 4 / コピー数: 1 / 式: B6D |
|---|---|
| 分子量 | 理論値: 246.26 Da |
| Chemical component information |  ChemComp-B6D: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | らせん対称体再構成法 |
| 試料の集合状態 | filament |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON IV (4k x 4k) 平均電子線量: 40.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 0.6 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: FLEXIBLE FIT |
|---|---|
| 得られたモデル |  PDB-8piz: |
 ムービー
ムービー コントローラー
コントローラー















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)