+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
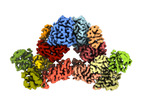
| タイトル | Cryo-EM structure of the c-di-GMP-free FleQ-FleN master regulator complex of P. aeruginosa | |||||||||
 マップデータ マップデータ | Sharpened cryo-EM density map of the c-di-GMP-free FleQ-FleN master regulator complex | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Pseudomonas aeruginosa / biofilm / c-di-GMP / transcription regulation / GENE REGULATION | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報positive regulation of cilium-dependent cell motility / regulation of bacterial-type flagellum-dependent cell motility / cyclic-di-GMP binding / negative regulation of extracellular matrix assembly / positive regulation of cell-substrate adhesion / DNA-binding transcription repressor activity / DNA-binding transcription activator activity / cis-regulatory region sequence-specific DNA binding / protein-DNA complex / cytoplasmic side of plasma membrane ...positive regulation of cilium-dependent cell motility / regulation of bacterial-type flagellum-dependent cell motility / cyclic-di-GMP binding / negative regulation of extracellular matrix assembly / positive regulation of cell-substrate adhesion / DNA-binding transcription repressor activity / DNA-binding transcription activator activity / cis-regulatory region sequence-specific DNA binding / protein-DNA complex / cytoplasmic side of plasma membrane / transcription cis-regulatory region binding / DNA-templated transcription / regulation of DNA-templated transcription / positive regulation of DNA-templated transcription / ATP hydrolysis activity / ATP binding / cytosol 類似検索 - 分子機能 | |||||||||
| 生物種 |  Pseudomonas aeruginosa PAO1 (緑膿菌) Pseudomonas aeruginosa PAO1 (緑膿菌) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.7 Å | |||||||||
 データ登録者 データ登録者 | Torres-Sanchez L / Krasteva PV | |||||||||
| 資金援助 | European Union, 1件
| |||||||||
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2023 ジャーナル: Proc Natl Acad Sci U S A / 年: 2023タイトル: Structures of the FleQ-FleN master regulators reveal large-scale conformational switching in motility and biofilm control. 著者: Lucía Torres-Sánchez / Thibault Géry Sana / Marion Decossas / Yaser Hashem / Petya Violinova Krasteva /  要旨: can cause a wide array of chronic and acute infections associated with its ability to rapidly switch between planktonic, biofilm, and dispersed lifestyles, each with a specific arsenal for bacterial ... can cause a wide array of chronic and acute infections associated with its ability to rapidly switch between planktonic, biofilm, and dispersed lifestyles, each with a specific arsenal for bacterial survival and virulence. At the cellular level, many of the physiological transitions are orchestrated by the intracellular second messenger c-di-GMP and its receptor-effector FleQ. A bacterial enhancer binding protein, FleQ acts as a master regulator of both flagellar motility and adherence factor secretion and uses remarkably different transcription activation mechanisms depending on its dinucleotide loading state, adenosine triphosphatase (ATPase) activity, interactions with polymerase sigma (σ) factors, and complexation with a second ATPase, FleN. How the FleQ-FleN tandem can exert diverse effects through recognition of a conserved FleQ binding consensus has remained enigmatic. Here, we provide cryogenic electron microscopy (cryo-EM) structures of both c-di-GMP-bound and c-di-GMP-free FleQ-FleN complexes which deepen our understanding of the proteins' (di)nucleotide-dependent conformational switching and fine-tuned roles in gene expression regulation. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_17445.map.gz emd_17445.map.gz | 587.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-17445-v30.xml emd-17445-v30.xml emd-17445.xml emd-17445.xml | 22 KB 22 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_17445_fsc.xml emd_17445_fsc.xml | 18.5 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_17445.png emd_17445.png | 93.9 KB | ||
| Filedesc metadata |  emd-17445.cif.gz emd-17445.cif.gz | 6.8 KB | ||
| その他 |  emd_17445_additional_1.map.gz emd_17445_additional_1.map.gz emd_17445_half_map_1.map.gz emd_17445_half_map_1.map.gz emd_17445_half_map_2.map.gz emd_17445_half_map_2.map.gz | 328.7 MB 621 MB 621 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-17445 http://ftp.pdbj.org/pub/emdb/structures/EMD-17445 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-17445 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-17445 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_17445_validation.pdf.gz emd_17445_validation.pdf.gz | 661.2 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_17445_full_validation.pdf.gz emd_17445_full_validation.pdf.gz | 660.8 KB | 表示 | |
| XML形式データ |  emd_17445_validation.xml.gz emd_17445_validation.xml.gz | 28.4 KB | 表示 | |
| CIF形式データ |  emd_17445_validation.cif.gz emd_17445_validation.cif.gz | 37.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-17445 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-17445 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-17445 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-17445 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8p53MC  8pb9C M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_17445.map.gz / 形式: CCP4 / 大きさ: 669.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_17445.map.gz / 形式: CCP4 / 大きさ: 669.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Sharpened cryo-EM density map of the c-di-GMP-free FleQ-FleN master regulator complex | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.66 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: Unsharpened cryo-EM density map of the c-di-GMP-free FleQ-FleN...
| ファイル | emd_17445_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Unsharpened cryo-EM density map of the c-di-GMP-free FleQ-FleN master regulator complex | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
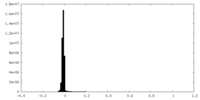
| 密度ヒストグラム |
-ハーフマップ: Half-map of the c-di-GMP-free FleQ-FleN master regulator complex
| ファイル | emd_17445_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half-map of the c-di-GMP-free FleQ-FleN master regulator complex | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
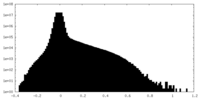
| 密度ヒストグラム |
-ハーフマップ: Half-map of the c-di-GMP-free FleQ-FleN master regulator complex
| ファイル | emd_17445_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half-map of the c-di-GMP-free FleQ-FleN master regulator complex | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
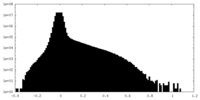
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : C-di-GMP-free FleQ-FleN complex
| 全体 | 名称: C-di-GMP-free FleQ-FleN complex |
|---|---|
| 要素 |
|
-超分子 #1: C-di-GMP-free FleQ-FleN complex
| 超分子 | 名称: C-di-GMP-free FleQ-FleN complex / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#2 詳細: Full-length FleQ co-purified with His-tagged FleN D48A |
|---|---|
| 由来(天然) | 生物種:  Pseudomonas aeruginosa PAO1 (緑膿菌) Pseudomonas aeruginosa PAO1 (緑膿菌) |
| 分子量 | 理論値: 288 KDa |
-分子 #1: Antiactivator FleN
| 分子 | 名称: Antiactivator FleN / タイプ: protein_or_peptide / ID: 1 / 詳細: P. aeruginosa FleN D48A / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Pseudomonas aeruginosa PAO1 (緑膿菌) / 株: PAO1 Pseudomonas aeruginosa PAO1 (緑膿菌) / 株: PAO1 |
| 分子量 | 理論値: 33.236332 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MSYYHHHHHH DYDIPTTLEV LFQGPMGSKQ MGSMHPVQVI AVTGGKGGVG KTNVSVNLAL ALADLGRRVM LLDAALGLAN VDVLLGLTP KRTLADVIEG RCELRDVLLL GPGGVRIVPA ASGTQSMVHL SPMQHAGLIQ AFSDISDNLD VLVVDTAAGI G DSVVSFVR ...文字列: MSYYHHHHHH DYDIPTTLEV LFQGPMGSKQ MGSMHPVQVI AVTGGKGGVG KTNVSVNLAL ALADLGRRVM LLDAALGLAN VDVLLGLTP KRTLADVIEG RCELRDVLLL GPGGVRIVPA ASGTQSMVHL SPMQHAGLIQ AFSDISDNLD VLVVDTAAGI G DSVVSFVR AAQEVLLVVC DEPTSITDAY ALIKLLNRDH GMTRFRVLAN MAHSPQEGRN LFAKLTKVTD RFLDVALQYV GV IPYDESV RKAVQKQRAV YEAFPRSKAS LAFKAVAQKV DSWPLPANPR GHLEFFVERL VQHPATGSAV UniProtKB: Antiactivator FleN |
-分子 #2: Transcriptional regulator FleQ
| 分子 | 名称: Transcriptional regulator FleQ / タイプ: protein_or_peptide / ID: 2 詳細: ...詳細: MGSWRETKLLLIDDNLDRSRDLAVILNFLGEDQLTCNSEDWREVAAGLSNSREALCVLLGSVESKGGAVELLKQLASWDEYLPILLIGEPAPADWPEELRRRVLASLEMPPSYNKLLDSLHRAQVYREMYDQARERGRSREPNLFRSLVGTSRAIQQVRQMMQQVADTDASVLILGESGTGKEVVARNLHYHSKRREGPFVPVNCGAIPAELLESELFGHEKGAFTGAITSRAGRFELANGGTLFLDEIGDMPLPMQVKLLRVLQERTFERVGSNKTQNVDVRIIAATHKNLEKMIEDGTFREDLYYRLNVFPIEMAPLRERVEDIALLLNELISRMEHEKRGSIRFNSAAIMSLCRHDWPGNVRELANLVERLAIMHPYGVIGVGELPKKFRHVDDEDEQLASSLREELEERAAINAGLPGMDAPAMLPAEGLDLKDYLANLEQGLIQQALDDAGGVVARAAERLRIRRTTLVEKMRKYGMSRRDDDLSDD コピー数: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Pseudomonas aeruginosa PAO1 (緑膿菌) Pseudomonas aeruginosa PAO1 (緑膿菌) |
| 分子量 | 理論値: 55.494125 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MGSWRETKLL LIDDNLDRSR DLAVILNFLG EDQLTCNSED WREVAAGLSN SREALCVLLG SVESKGGAVE LLKQLASWDE YLPILLIGE PAPADWPEEL RRRVLASLEM PPSYNKLLDS LHRAQVYREM YDQARERGRS REPNLFRSLV GTSRAIQQVR Q MMQQVADT ...文字列: MGSWRETKLL LIDDNLDRSR DLAVILNFLG EDQLTCNSED WREVAAGLSN SREALCVLLG SVESKGGAVE LLKQLASWDE YLPILLIGE PAPADWPEEL RRRVLASLEM PPSYNKLLDS LHRAQVYREM YDQARERGRS REPNLFRSLV GTSRAIQQVR Q MMQQVADT DASVLILGES GTGKEVVARN LHYHSKRREG PFVPVNCGAI PAELLESELF GHEKGAFTGA ITSRAGRFEL AN GGTLFLD EIGDMPLPMQ VKLLRVLQER TFERVGSNKT QNVDVRIIAA THKNLEKMIE DGTFREDLYY RLNVFPIEMA PLR ERVEDI ALLLNELISR MEHEKRGSIR FNSAAIMSLC RHDWPGNVRE LANLVERLAI MHPYGVIGVG ELPKKFRHVD DEDE QLASS LREELEERAA INAGLPGMDA PAMLPAEGLD LKDYLANLEQ GLIQQALDDA GGVVARAAER LRIRRTTLVE KMRKY GMSR RDDDLSDD UniProtKB: Transcriptional regulator FleQ |
-分子 #3: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 3 / コピー数: 2 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-分子 #4: PHOSPHOMETHYLPHOSPHONIC ACID ADENYLATE ESTER
| 分子 | 名称: PHOSPHOMETHYLPHOSPHONIC ACID ADENYLATE ESTER / タイプ: ligand / ID: 4 / コピー数: 2 / 式: ACP |
|---|---|
| 分子量 | 理論値: 505.208 Da |
| Chemical component information |  ChemComp-ACP: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 2 mg/mL |
|---|---|
| 緩衝液 | pH: 8 詳細: 20 mM HEPES pH 8.0, 250mM NaCl, 2mM MgCl2, and 2% glycerol |
| グリッド | モデル: UltrAuFoil R1.2/1.3 / 材質: GOLD / 支持フィルム - 材質: GOLD / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 60 sec. / 前処理 - 雰囲気: AIR |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 4 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Quantum LS |
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / デジタル化 - サイズ - 横: 5760 pixel / デジタル化 - サイズ - 縦: 4092 pixel / 撮影したグリッド数: 1 / 実像数: 21233 / 平均電子線量: 51.5 e/Å2 / 詳細: 17099 micrographs retained for processing |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 0.3 µm |
| 試料ステージ | ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: AB INITIO MODEL |
|---|---|
| 得られたモデル |  PDB-8p53: |
 ムービー
ムービー コントローラー
コントローラー






 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)