+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of Phthaloyl-CoA decarboxylase (Pcd) bound with product, benzoyl-CoA | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | prFMN / plastic degradation / Phthalates / anaerobic / light sensitive / Co-enzyme A / decarboxylase / benzoyl CoA / product bound / FLAVOPROTEIN | |||||||||
| 生物種 |  Thauera chlorobenzoica (バクテリア) Thauera chlorobenzoica (バクテリア) | |||||||||
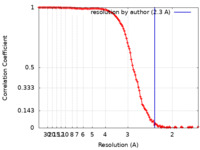
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.3 Å | |||||||||
 データ登録者 データ登録者 | Kayastha K / Ermler U | |||||||||
| 資金援助 |  ドイツ, 1件 ドイツ, 1件
| |||||||||
 引用 引用 |  ジャーナル: To Be Published ジャーナル: To Be Publishedタイトル: Cryo-EM structure of Phthaloyl-CoA decarboxylase (Pcd) bound with product, benzoyl-CoA 著者: Kayastha K / Ermler U | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_17324.map.gz emd_17324.map.gz | 97.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-17324-v30.xml emd-17324-v30.xml emd-17324.xml emd-17324.xml | 18.5 KB 18.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_17324_fsc.xml emd_17324_fsc.xml | 14.5 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_17324.png emd_17324.png | 172.6 KB | ||
| Filedesc metadata |  emd-17324.cif.gz emd-17324.cif.gz | 6.1 KB | ||
| その他 |  emd_17324_half_map_1.map.gz emd_17324_half_map_1.map.gz emd_17324_half_map_2.map.gz emd_17324_half_map_2.map.gz | 98.3 MB 98.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-17324 http://ftp.pdbj.org/pub/emdb/structures/EMD-17324 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-17324 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-17324 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_17324_validation.pdf.gz emd_17324_validation.pdf.gz | 740.5 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_17324_full_validation.pdf.gz emd_17324_full_validation.pdf.gz | 740.1 KB | 表示 | |
| XML形式データ |  emd_17324_validation.xml.gz emd_17324_validation.xml.gz | 18.4 KB | 表示 | |
| CIF形式データ |  emd_17324_validation.cif.gz emd_17324_validation.cif.gz | 24.5 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-17324 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-17324 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-17324 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-17324 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_17324.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_17324.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.837 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #1
| ファイル | emd_17324_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
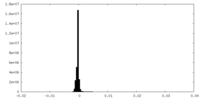
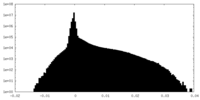
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_17324_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
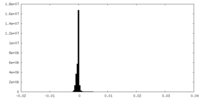
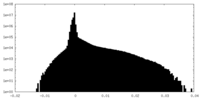
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Phthaloyl-CoA decarboxylase (Pcd) bound with benzoyl-CoA
| 全体 | 名称: Phthaloyl-CoA decarboxylase (Pcd) bound with benzoyl-CoA |
|---|---|
| 要素 |
|
-超分子 #1: Phthaloyl-CoA decarboxylase (Pcd) bound with benzoyl-CoA
| 超分子 | 名称: Phthaloyl-CoA decarboxylase (Pcd) bound with benzoyl-CoA タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  Thauera chlorobenzoica (バクテリア) Thauera chlorobenzoica (バクテリア) |
| 分子量 | 理論値: 360 KDa |
-分子 #1: Phthaloyl-CoA decarboxylase (Pcd)
| 分子 | 名称: Phthaloyl-CoA decarboxylase (Pcd) / タイプ: protein_or_peptide / ID: 1 詳細: Pcd monomers bound with co-factors prFMN, Fe, K and reaction product, Benzoyl-CoA. コピー数: 6 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Thauera chlorobenzoica (バクテリア) Thauera chlorobenzoica (バクテリア) |
| 分子量 | 理論値: 57.682492 KDa |
| 配列 | 文字列: ERVGEKDLRA ALEWFRSKGY LVETNKEVNP DLEITGLQKI FDGSLPMLFN NVKDMPHARA ITNLFGDIRV VEELFGWENS LDRVKKVAR AIDHPLKPVI IGQDEAPVQE EVLTTDLDVN KWLTAIRHTP LETEMTIGSG ISCVVGPYFD GGSHIGYNRM N FRWGNVGT ...文字列: ERVGEKDLRA ALEWFRSKGY LVETNKEVNP DLEITGLQKI FDGSLPMLFN NVKDMPHARA ITNLFGDIRV VEELFGWENS LDRVKKVAR AIDHPLKPVI IGQDEAPVQE EVLTTDLDVN KWLTAIRHTP LETEMTIGSG ISCVVGPYFD GGSHIGYNRM N FRWGNVGT FQISPGSHMW QVMTEHYKDD EPIPLTMCFG VPPSCTYVAG AGFDYAILPK GCDEIGIAGA IQGSPVRLVK CR TIDAYTL ADAEYVLEGY LHPRDKRYET AESEAADIQG RFHFHPEWAG YMGKAYKAPT FHVTAITMRR RESKPIIFPL GVH TADDAN IDTSVRESAI FALCERLQPG IVQNVHIPYC MTDWGGCIIQ VKKRNQIEEG WQRNFLAAIL ACSQGMRLAI AVSE DVDIY SMDDIMWCLT TRVNPQTDIL NPLPGGRGQT FMPAERMTSG DKQWTASNTQ FEGGMGIDAT VPYGYESDFH RPVYG VDLV KPENFFDAKD IDKMKSRMAG WVLSLARTGR |
-分子 #2: benzoyl coenzyme A
| 分子 | 名称: benzoyl coenzyme A / タイプ: ligand / ID: 2 / コピー数: 6 / 式: BYC |
|---|---|
| 分子量 | 理論値: 871.64 Da |
| Chemical component information | 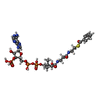 ChemComp-BYC: |
-分子 #3: hydroxylated prenyl-FMN
| 分子 | 名称: hydroxylated prenyl-FMN / タイプ: ligand / ID: 3 / コピー数: 6 / 式: BYN |
|---|---|
| 分子量 | 理論値: 542.476 Da |
| Chemical component information |  ChemComp-BYN: |
-分子 #4: FE (III) ION
| 分子 | 名称: FE (III) ION / タイプ: ligand / ID: 4 / コピー数: 12 / 式: FE |
|---|---|
| 分子量 | 理論値: 55.845 Da |
-分子 #5: CALCIUM ION
| 分子 | 名称: CALCIUM ION / タイプ: ligand / ID: 5 / コピー数: 6 / 式: CA |
|---|---|
| 分子量 | 理論値: 40.078 Da |
-分子 #6: POTASSIUM ION
| 分子 | 名称: POTASSIUM ION / タイプ: ligand / ID: 6 / コピー数: 6 / 式: K |
|---|---|
| 分子量 | 理論値: 39.098 Da |
-分子 #7: water
| 分子 | 名称: water / タイプ: ligand / ID: 7 / コピー数: 59 / 式: HOH |
|---|---|
| 分子量 | 理論値: 18.015 Da |
| Chemical component information |  ChemComp-HOH: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1.5 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 |
| グリッド | モデル: C-flat-1.2/1.3 / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 135 sec. |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277.15 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 30 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 撮影したグリッド数: 1 / 実像数: 6743 / 平均露光時間: 3.38 sec. / 平均電子線量: 52.27 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.1 µm / 最小 デフォーカス(公称値): 1.2 µm / 倍率(公称値): 105000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | Chain - Source name: Other / Chain - Initial model type: experimental model / 詳細: pdb from the first holo-complex was used |
|---|---|
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT |
| 得られたモデル |  PDB-8p02: |
 ムービー
ムービー コントローラー
コントローラー




 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)