+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-1688 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | CryoEM reconstruction of human parechovirus 1 complexed with integrin alphaV-beta3 | |||||||||
 マップデータ マップデータ | This is a cryoEM reconstruction of human parechovirus 1 complexed with integrin alphaV-beta3 at 15 angstrom resolution | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | picornavirus / parechovirus / icosahedral / integrin | |||||||||
| 生物種 |  Homo sapiens (ヒト) / Homo sapiens (ヒト) /  Human parechovirus 1 (ウイルス) Human parechovirus 1 (ウイルス) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / ネガティブ染色法 / 解像度: 15.0 Å | |||||||||
 データ登録者 データ登録者 | Seitsonen JJT / Susi P / Heikkila O / Laurinmaki P / Hyypia T / Butcher SJ | |||||||||
 引用 引用 |  ジャーナル: J Virol / 年: 2010 ジャーナル: J Virol / 年: 2010タイトル: Interaction of alphaVbeta3 and alphaVbeta6 integrins with human parechovirus 1. 著者: Jani Seitsonen / Petri Susi / Outi Heikkilä / Robert S Sinkovits / Pasi Laurinmäki / Timo Hyypiä / Sarah J Butcher /  要旨: Human parechovirus (HPEV) infections are very common in early childhood and can be severe in neonates. It has been shown that integrins are important for cellular infectivity of HPEV1 through ...Human parechovirus (HPEV) infections are very common in early childhood and can be severe in neonates. It has been shown that integrins are important for cellular infectivity of HPEV1 through experiments using peptide blocking assays and function-blocking antibodies to alpha(V) integrins. The interaction of HPEV1 with alpha(V) integrins is presumably mediated by a C-terminal RGD motif in the capsid protein VP1. We characterized the binding of integrins alpha(V)beta(3) and alpha(V)beta(6) to HPEV1 by biochemical and structural studies. We showed that although HPEV1 bound efficiently to immobilized integrins, alpha(V)beta(6) bound more efficiently than alpha(V)beta(3) to immobilized HPEV1. Moreover, soluble alpha(V)beta(6), but not alpha(V)beta(3), blocked HPEV1 cellular infectivity, indicating that it is a high-affinity receptor for HPEV1. We also showed that HPEV1 binding to integrins in vitro could be partially blocked by RGD peptides. Using electron cryo-microscopy and image reconstruction, we showed that HPEV1 has the typical T=1 (pseudo T=3) organization of a picornavirus. Complexes of HPEV1 and integrins indicated that both integrin footprints reside between the 5-fold and 3-fold symmetry axes. This result does not match the RGD position predicted from the coxsackievirus A9 X-ray structure but is consistent with the predicted location of this motif in the shorter C terminus found in HPEV1. This first structural characterization of a parechovirus indicates that the differences in receptor binding are due to the amino acid differences in the integrins rather than to significantly different viral footprints. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_1688.map.gz emd_1688.map.gz | 40.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-1688-v30.xml emd-1688-v30.xml emd-1688.xml emd-1688.xml | 10.4 KB 10.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  EMD-1688_HPEV1avb3.tif EMD-1688_HPEV1avb3.tif | 732.6 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-1688 http://ftp.pdbj.org/pub/emdb/structures/EMD-1688 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1688 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1688 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_1688_validation.pdf.gz emd_1688_validation.pdf.gz | 216.3 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_1688_full_validation.pdf.gz emd_1688_full_validation.pdf.gz | 215.4 KB | 表示 | |
| XML形式データ |  emd_1688_validation.xml.gz emd_1688_validation.xml.gz | 6.6 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1688 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1688 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1688 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1688 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_1688.map.gz / 形式: CCP4 / 大きさ: 120.1 MB / タイプ: IMAGE STORED AS SIGNED INTEGER (2 BYTES) ダウンロード / ファイル: emd_1688.map.gz / 形式: CCP4 / 大きさ: 120.1 MB / タイプ: IMAGE STORED AS SIGNED INTEGER (2 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | This is a cryoEM reconstruction of human parechovirus 1 complexed with integrin alphaV-beta3 at 15 angstrom resolution | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.13 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
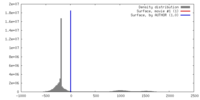
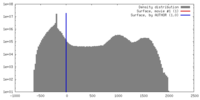
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Purified human parechovirus 1 in 1 mM MgCl2 and integrin alphaV-b...
| 全体 | 名称: Purified human parechovirus 1 in 1 mM MgCl2 and integrin alphaV-beta6 in PBS buffer |
|---|---|
| 要素 |
|
-超分子 #1000: Purified human parechovirus 1 in 1 mM MgCl2 and integrin alphaV-b...
| 超分子 | 名称: Purified human parechovirus 1 in 1 mM MgCl2 and integrin alphaV-beta6 in PBS buffer タイプ: sample / ID: 1000 集合状態: Complete particle (60-mer) with the RNA genome inside with few integrin molecules attached Number unique components: 2 |
|---|
-超分子 #1: Human parechovirus 1
| 超分子 | 名称: Human parechovirus 1 / タイプ: virus / ID: 1 / Name.synonym: HPEV1 / NCBI-ID: 12063 / 生物種: Human parechovirus 1 / ウイルスタイプ: VIRION / ウイルス・単離状態: STRAIN / ウイルス・エンベロープ: No / ウイルス・中空状態: No / Syn species name: HPEV1 |
|---|---|
| 宿主 | 生物種:  Homo sapiens (ヒト) / 別称: VERTEBRATES Homo sapiens (ヒト) / 別称: VERTEBRATES |
| 分子量 | 理論値: 5.1 MDa |
| ウイルス殻 | Shell ID: 1 / 名称: native / 直径: 280 Å / T番号(三角分割数): 1 |
-分子 #1: Integrin alphaV-beta3
| 分子 | 名称: Integrin alphaV-beta3 / タイプ: protein_or_peptide / ID: 1 / Name.synonym: Integrin alphaV-beta3 / 詳細: Product of BioMarket Ltd., Finland / コピー数: 1 / 集合状態: Heterodimer / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) / 別称: Human / 細胞中の位置: Plasma membrane Homo sapiens (ヒト) / 別称: Human / 細胞中の位置: Plasma membrane |
| 分子量 | 理論値: 203 KDa |
-実験情報
-構造解析
| 手法 | ネガティブ染色法, クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1.24 mg/mL |
|---|---|
| 緩衝液 | pH: 7.2 / 詳細: 1 mM MgCl2 in PBS |
| 染色 | タイプ: NEGATIVE / 詳細: Cryo preparation |
| グリッド | 詳細: 400 mesh carbon coated copper |
| 凍結 | 凍結剤: ETHANE / 装置: HOMEMADE PLUNGER / 詳細: Vitrification instrument: Guillotine / 手法: Blot for 1-2 seconds before plunging |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI F20 |
|---|---|
| 温度 | 平均: 91 K |
| アライメント法 | Legacy - 非点収差: objective lens astigmatism was corrected at 62,000 times magnification |
| 撮影 | カテゴリ: FILM / フィルム・検出器のモデル: KODAK SO-163 FILM / デジタル化 - スキャナー: OTHER / デジタル化 - サンプリング間隔: 7 µm / 実像数: 16 / 平均電子線量: 18 e/Å2 / 詳細: Scanned with Zeiss Photoscan TD scanner |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 60500 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.0 mm / 最大 デフォーカス(公称値): 2.922 µm / 最小 デフォーカス(公称値): 1.486 µm / 倍率(公称値): 62000 |
| 試料ステージ | 試料ホルダー: Gatan 626 / 試料ホルダーモデル: GATAN LIQUID NITROGEN |
| 実験機器 |  モデル: Tecnai F20 / 画像提供: FEI Company |
- 画像解析
画像解析
| CTF補正 | 詳細: Each micrograph |
|---|---|
| 最終 再構成 | 想定した対称性 - 点群: I (正20面体型対称) / アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 15.0 Å / 解像度の算出法: FSC 0.5 CUT-OFF / ソフトウェア - 名称: AUTO3DEM / 使用した粒子像数: 431 |
 ムービー
ムービー コントローラー
コントローラー












 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)