+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of the Nucleosome Core Particle from Trypanosoma brucei | |||||||||
 マップデータ マップデータ | Masked sharpened map of Nucleosome core particle from Trypanosoma brucei | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Nucleosome Chromatin Parasite Trypanosome Kinetoplast / DNA BINDING PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報ciliary transition zone / ciliary plasm / nuclear lumen / phosphate ion binding / chromosome organization / structural constituent of chromatin / nucleosome / heterochromatin formation / nucleosome assembly / protein heterodimerization activity ...ciliary transition zone / ciliary plasm / nuclear lumen / phosphate ion binding / chromosome organization / structural constituent of chromatin / nucleosome / heterochromatin formation / nucleosome assembly / protein heterodimerization activity / DNA binding / nucleoplasm / nucleus / cytoplasm 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.3 Å | |||||||||
 データ登録者 データ登録者 | Sandoval G / Deak G / Tuijtel MW / Wilson MD | |||||||||
| 資金援助 |  英国, 2件 英国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nucleic Acids Res / 年: 2023 ジャーナル: Nucleic Acids Res / 年: 2023タイトル: Histone divergence in trypanosomes results in unique alterations to nucleosome structure. 著者: Gauri Deák / Hannah Wapenaar / Gorka Sandoval / Ruofan Chen / Mark R D Taylor / Hayden Burdett / James A Watson / Maarten W Tuijtel / Shaun Webb / Marcus D Wilson /   要旨: Eukaryotes have a multitude of diverse mechanisms for organising and using their genomes, but the histones that make up chromatin are highly conserved. Unusually, histones from kinetoplastids are ...Eukaryotes have a multitude of diverse mechanisms for organising and using their genomes, but the histones that make up chromatin are highly conserved. Unusually, histones from kinetoplastids are highly divergent. The structural and functional consequences of this variation are unknown. Here, we have biochemically and structurally characterised nucleosome core particles (NCPs) from the kinetoplastid parasite Trypanosoma brucei. A structure of the T. brucei NCP reveals that global histone architecture is conserved, but specific sequence alterations lead to distinct DNA and protein interaction interfaces. The T. brucei NCP is unstable and has weakened overall DNA binding. However, dramatic changes at the H2A-H2B interface introduce local reinforcement of DNA contacts. The T. brucei acidic patch has altered topology and is refractory to known binders, indicating that the nature of chromatin interactions in T. brucei may be unique. Overall, our results provide a detailed molecular basis for understanding evolutionary divergence in chromatin structure. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_16777.map.gz emd_16777.map.gz | 113.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-16777-v30.xml emd-16777-v30.xml emd-16777.xml emd-16777.xml | 27.3 KB 27.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_16777.png emd_16777.png | 64.1 KB | ||
| マスクデータ |  emd_16777_msk_1.map emd_16777_msk_1.map | 120.4 MB |  マスクマップ マスクマップ | |
| その他 |  emd_16777_additional_1.map.gz emd_16777_additional_1.map.gz emd_16777_half_map_1.map.gz emd_16777_half_map_1.map.gz emd_16777_half_map_2.map.gz emd_16777_half_map_2.map.gz | 59.9 MB 111.6 MB 111.6 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-16777 http://ftp.pdbj.org/pub/emdb/structures/EMD-16777 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-16777 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-16777 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_16777_validation.pdf.gz emd_16777_validation.pdf.gz | 1 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_16777_full_validation.pdf.gz emd_16777_full_validation.pdf.gz | 1 MB | 表示 | |
| XML形式データ |  emd_16777_validation.xml.gz emd_16777_validation.xml.gz | 14 KB | 表示 | |
| CIF形式データ |  emd_16777_validation.cif.gz emd_16777_validation.cif.gz | 16.5 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16777 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16777 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16777 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16777 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8comMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_16777.map.gz / 形式: CCP4 / 大きさ: 120.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_16777.map.gz / 形式: CCP4 / 大きさ: 120.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Masked sharpened map of Nucleosome core particle from Trypanosoma brucei | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.829 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_16777_msk_1.map emd_16777_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
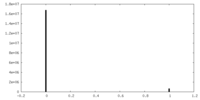
| 密度ヒストグラム |
-追加マップ: unsharpened map of Nucleosome core particle from Trypanosoma brucei
| ファイル | emd_16777_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | unsharpened map of Nucleosome core particle from Trypanosoma brucei | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
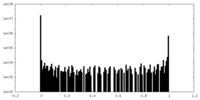
| 密度ヒストグラム |
-ハーフマップ: half map 2 of Nucleosome core particle from Trypanosoma brucei
| ファイル | emd_16777_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map 2 of Nucleosome core particle from Trypanosoma brucei | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
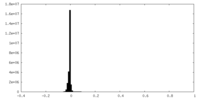
| 密度ヒストグラム |
-ハーフマップ: half map 1 of Nucleosome core particle from Trypanosoma brucei
| ファイル | emd_16777_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map 1 of Nucleosome core particle from Trypanosoma brucei | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
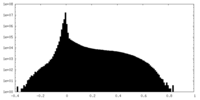
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Nucleosome core particle
| 全体 | 名称: Nucleosome core particle |
|---|---|
| 要素 |
|
-超分子 #1: Nucleosome core particle
| 超分子 | 名称: Nucleosome core particle / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all 詳細: Nucleosome core particle with histones from Trypanosoma brucei and Widom 601 145 bp DNA |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 89.6 KDa |
-超分子 #2: Histone octamer
| 超分子 | 名称: Histone octamer / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1-#4 詳細: Trypanosoma brucei histone octamer composed of an H4-H3-H3-H4 tetramer and two H2A-H2B dimers |
|---|---|
| 由来(天然) | 生物種:  |
-超分子 #3: Widom 601 145 bp DNA
| 超分子 | 名称: Widom 601 145 bp DNA / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #5-#6 |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
-分子 #1: Histone H3, putative
| 分子 | 名称: Histone H3, putative / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  株: TREU927 |
| 分子量 | 理論値: 14.647858 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: SRTKETARTK KTITSKKSKK ASKGSDAASG VKTAQRRWRP GTVALREIRQ FQRSTDLLLQ KAPFQRLVRE VSGAQKEGLR FQSSAILAA QEATESYIVS LLADTNRACI HSGRVTIQPK DIHLALCLRG ERA UniProtKB: Histone H3, putative |
-分子 #2: Histone H4
| 分子 | 名称: Histone H4 / タイプ: protein_or_peptide / ID: 2 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  株: TREU927 |
| 分子量 | 理論値: 11.037914 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: AKGKKSGEAK GSQKRQKKVL RENVRGITRG SIRRLARRGG VKRISGVIYD EVRGVLKSFV EGVVRDATAY TEYSRKKTVT AVDVVNALR KRGKILYGYA UniProtKB: Histone H4 |
-分子 #3: Histone H2A
| 分子 | 名称: Histone H2A / タイプ: protein_or_peptide / ID: 3 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  株: TREU927 |
| 分子量 | 理論値: 14.108614 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: ATPKQAVKKA SKGGSSRSVK AGLIFPVGRV GTLLRRGQYA RRIGASGAVY MAAVLEYLTA ELLELSVKAA AQQTKKTKRL TPRTVTLAV RHDDDLGALL RNVTMSRGGV MPSLNKALAK KQKSGKHAKA TPSV UniProtKB: Histone H2A |
-分子 #4: Histone H2B
| 分子 | 名称: Histone H2B / タイプ: protein_or_peptide / ID: 4 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  株: TREU927 |
| 分子量 | 理論値: 12.464503 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: ATPKSTPAKT RKEAKKTRRQ RKRTWNVYVS RSLRSINSQM SMTSRTMKIV NSFVNDLFER IAAEAATIVR VNRKRTLGAR ELQTAVRLV LPADLAKHAM AEGTKAVSHA SS UniProtKB: Histone H2B |
-分子 #5: Widom 601 145 bp DNA (127-mer ordered and built)
| 分子 | 名称: Widom 601 145 bp DNA (127-mer ordered and built) / タイプ: dna / ID: 5 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 44.99166 KDa |
| 配列 | 文字列: (DA)(DT)(DC)(DG)(DA)(DT)(DG)(DT)(DA)(DT) (DA)(DT)(DA)(DT)(DC)(DT)(DG)(DA)(DC)(DA) (DC)(DG)(DT)(DG)(DC)(DC)(DT)(DG)(DG) (DA)(DG)(DA)(DC)(DT)(DA)(DG)(DG)(DG)(DA) (DG) (DT)(DA)(DA)(DT)(DC) ...文字列: (DA)(DT)(DC)(DG)(DA)(DT)(DG)(DT)(DA)(DT) (DA)(DT)(DA)(DT)(DC)(DT)(DG)(DA)(DC)(DA) (DC)(DG)(DT)(DG)(DC)(DC)(DT)(DG)(DG) (DA)(DG)(DA)(DC)(DT)(DA)(DG)(DG)(DG)(DA) (DG) (DT)(DA)(DA)(DT)(DC)(DC)(DC)(DC) (DT)(DT)(DG)(DG)(DC)(DG)(DG)(DT)(DT)(DA) (DA)(DA) (DA)(DC)(DG)(DC)(DG)(DG)(DG) (DG)(DG)(DA)(DC)(DA)(DG)(DC)(DG)(DC)(DG) (DT)(DA)(DC) (DG)(DT)(DG)(DC)(DG)(DT) (DT)(DT)(DA)(DA)(DG)(DC)(DG)(DG)(DT)(DG) (DC)(DT)(DA)(DG) (DA)(DG)(DC)(DT)(DG) (DT)(DC)(DT)(DA)(DC)(DG)(DA)(DC)(DC)(DA) (DA)(DT)(DT)(DG)(DA) (DG)(DC)(DG)(DG) (DC)(DC)(DT)(DC)(DG)(DG)(DC)(DA)(DC)(DC) (DG)(DG)(DG)(DA)(DT)(DT) (DC)(DT)(DG) (DA)(DT) |
-分子 #6: Widom 601 145 bp DNA (127-mer ordered and built)
| 分子 | 名称: Widom 601 145 bp DNA (127-mer ordered and built) / タイプ: dna / ID: 6 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 44.520383 KDa |
| 配列 | 文字列: (DA)(DT)(DC)(DA)(DG)(DA)(DA)(DT)(DC)(DC) (DC)(DG)(DG)(DT)(DG)(DC)(DC)(DG)(DA)(DG) (DG)(DC)(DC)(DG)(DC)(DT)(DC)(DA)(DA) (DT)(DT)(DG)(DG)(DT)(DC)(DG)(DT)(DA)(DG) (DA) (DC)(DA)(DG)(DC)(DT) ...文字列: (DA)(DT)(DC)(DA)(DG)(DA)(DA)(DT)(DC)(DC) (DC)(DG)(DG)(DT)(DG)(DC)(DC)(DG)(DA)(DG) (DG)(DC)(DC)(DG)(DC)(DT)(DC)(DA)(DA) (DT)(DT)(DG)(DG)(DT)(DC)(DG)(DT)(DA)(DG) (DA) (DC)(DA)(DG)(DC)(DT)(DC)(DT)(DA) (DG)(DC)(DA)(DC)(DC)(DG)(DC)(DT)(DT)(DA) (DA)(DA) (DC)(DG)(DC)(DA)(DC)(DG)(DT) (DA)(DC)(DG)(DC)(DG)(DC)(DT)(DG)(DT)(DC) (DC)(DC)(DC) (DC)(DG)(DC)(DG)(DT)(DT) (DT)(DT)(DA)(DA)(DC)(DC)(DG)(DC)(DC)(DA) (DA)(DG)(DG)(DG) (DG)(DA)(DT)(DT)(DA) (DC)(DT)(DC)(DC)(DC)(DT)(DA)(DG)(DT)(DC) (DT)(DC)(DC)(DA)(DG) (DG)(DC)(DA)(DC) (DG)(DT)(DG)(DT)(DC)(DA)(DG)(DA)(DT)(DA) (DT)(DA)(DT)(DA)(DC)(DA) (DT)(DC)(DG) (DA)(DT) |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.5 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 / 詳細: 20 mM HEPES pH 7.5, 150 mM NaCl, 1 mM DTT |
| グリッド | モデル: Quantifoil R2/2 / 材質: COPPER / メッシュ: 300 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 120 sec. / 前処理 - 雰囲気: AIR / 前処理 - 気圧: 3e-05 kPa / 詳細: PELCO easiglow |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
| 詳細 | Glutaraldehyde fixation, S200 purification |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 撮影したグリッド数: 1 / 実像数: 4913 / 平均露光時間: 7.0 sec. / 平均電子線量: 45.7 e/Å2 詳細: correlated double sampling used, super-resolution, binning at motion correction stage |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 75.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.2 µm / 最小 デフォーカス(公称値): 1.5 µm / 倍率(公称値): 105000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー






 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)