+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | HIV-1 mature capsid hexamer from CA-IP6 CLPs | ||||||||||||||||||
 マップデータ マップデータ | |||||||||||||||||||
 試料 試料 |
| ||||||||||||||||||
 キーワード キーワード | Capsid / Hexamer / HIV-1 / Mature / VIRUS LIKE PARTICLE | ||||||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報viral nucleocapsid / host cell cytoplasm / viral translational frameshifting / host cell nucleus / virion membrane / structural molecule activity / RNA binding / zinc ion binding / ATP binding 類似検索 - 分子機能 | ||||||||||||||||||
| 生物種 |   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) | ||||||||||||||||||
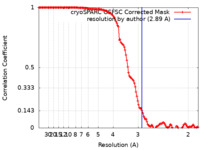
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.89 Å | ||||||||||||||||||
 データ登録者 データ登録者 | Stacey JCV / Briggs JAG | ||||||||||||||||||
| 資金援助 |  ドイツ, ドイツ,  米国, 米国,  英国, 5件 英国, 5件
| ||||||||||||||||||
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2023 ジャーナル: Proc Natl Acad Sci U S A / 年: 2023タイトル: Two structural switches in HIV-1 capsid regulate capsid curvature and host factor binding. 著者: James C V Stacey / Aaron Tan / John M Lu / Leo C James / Robert A Dick / John A G Briggs /    要旨: The mature HIV-1 capsid protects the viral genome and interacts with host proteins to travel from the cell periphery into the nucleus. To achieve this, the capsid protein, CA, constructs conical ...The mature HIV-1 capsid protects the viral genome and interacts with host proteins to travel from the cell periphery into the nucleus. To achieve this, the capsid protein, CA, constructs conical capsids from a lattice of hexamers and pentamers, and engages in and then relinquishes multiple interactions with cellular proteins in an orchestrated fashion. Cellular host factors including Nup153, CPSF6, and Sec24C engage the same pocket within CA hexamers. How CA assembles pentamers and hexamers of different curvatures, how CA oligomerization states or curvature might modulate host-protein interactions, and how binding of multiple cofactors to a single site is coordinated, all remain to be elucidated. Here, using single-particle cryoEM, we have determined the structure of the mature HIV-1 CA pentamer and hexamer from conical CA-IP polyhedra to ~3 Å resolution. We also determined structures of hexamers in the context of multiple lattice curvatures and number of pentamer contacts. Comparison of these structures, bound or not to host protein peptides, revealed two structural switches within HIV-1 CA that modulate peptide binding according to CA lattice curvature and whether CA is hexameric or pentameric. These observations suggest that the conical HIV-1 capsid has different host-protein binding properties at different positions on its surface, which may facilitate cell entry and represent an evolutionary advantage of conical morphology. | ||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_16703.map.gz emd_16703.map.gz | 202.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-16703-v30.xml emd-16703-v30.xml emd-16703.xml emd-16703.xml | 19.8 KB 19.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_16703_fsc.xml emd_16703_fsc.xml | 14.3 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_16703.png emd_16703.png | 114.3 KB | ||
| マスクデータ |  emd_16703_msk_1.map emd_16703_msk_1.map | 216 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-16703.cif.gz emd-16703.cif.gz | 6.2 KB | ||
| その他 |  emd_16703_half_map_1.map.gz emd_16703_half_map_1.map.gz emd_16703_half_map_2.map.gz emd_16703_half_map_2.map.gz | 195.9 MB 195.9 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-16703 http://ftp.pdbj.org/pub/emdb/structures/EMD-16703 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-16703 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-16703 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8ckvMC  8ckwC  8ckxC  8ckyC  8ckzC  8cl0C  8cl1C  8cl2C  8cl3C  8cl4C M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_16703.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_16703.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.93 Å | ||||||||||||||||||||||||||||||||||||
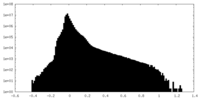
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_16703_msk_1.map emd_16703_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
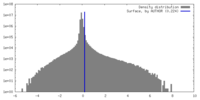
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_16703_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
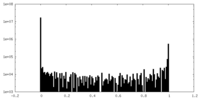
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_16703_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
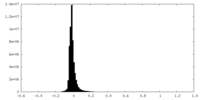
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : HIV-1 Mature Capsid
| 全体 | 名称: HIV-1 Mature Capsid |
|---|---|
| 要素 |
|
-超分子 #1: HIV-1 Mature Capsid
| 超分子 | 名称: HIV-1 Mature Capsid / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all / 詳細: Hexamer |
|---|---|
| 由来(天然) | 生物種:   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) |
| 分子量 | 理論値: 26 KDa |
-分子 #1: Gag polyprotein
| 分子 | 名称: Gag polyprotein / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) |
| 分子量 | 理論値: 25.761623 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MPIVQNLQGQ MVHQAISPRT LNAWVKVVEE KAFSPEVIPM FSALSEGATP QDLNTMLNTV GGHQAAMQML KETINEEAAE WDRLHPVHA GPIAPGQMRE PRGSDIAGTT STLQEQIGWM THNPPIPVGE IYKRWIILGL NKIVRMYSPT SILDIRQGPK E PFRDYVDR ...文字列: MPIVQNLQGQ MVHQAISPRT LNAWVKVVEE KAFSPEVIPM FSALSEGATP QDLNTMLNTV GGHQAAMQML KETINEEAAE WDRLHPVHA GPIAPGQMRE PRGSDIAGTT STLQEQIGWM THNPPIPVGE IYKRWIILGL NKIVRMYSPT SILDIRQGPK E PFRDYVDR FYKTLRAEQA SQEVKNWMTE TLLVQNANPD CKTILKALGP GATLEEMMTA CQGVGGPGHK ARVL UniProtKB: Gag polyprotein |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.5 mg/mL | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 6.1 構成要素:
| |||||||||||||||
| グリッド | モデル: C-flat-2/2 / 材質: COPPER / メッシュ: 200 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 支持フィルム - Film thickness: 2 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 30 sec. / 前処理 - 雰囲気: AIR | |||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 291 K / 装置: FEI VITROBOT MARK IV | |||||||||||||||
| 詳細 | VLP assembly. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: TFS Selectris / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: FEI FALCON IV (4k x 4k) デジタル化 - サイズ - 横: 4096 pixel / デジタル化 - サイズ - 縦: 4096 pixel / 撮影したグリッド数: 1 / 実像数: 27252 / 平均電子線量: 40.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 0.6 µm / 倍率(公称値): 130000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 詳細 | Initial model:4XFX |
|---|---|
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT / 当てはまり具合の基準: Cross-correlation |
| 得られたモデル |  PDB-8ckv: |
 ムービー
ムービー コントローラー
コントローラー














 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)