+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of the SNV L protein bound to 5' RNA | ||||||||||||||||||
 マップデータ マップデータ | |||||||||||||||||||
 試料 試料 |
| ||||||||||||||||||
 キーワード キーワード | Negative-strand viruses / Bunyaviruses / Hantaviruses / RNA-dependent RNA polymerase / viral genome replication and transcription / VIRUS PROTEIN / VIRAL PROTEIN | ||||||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報cap snatching / endonuclease activity / host cell perinuclear region of cytoplasm / RNA-directed RNA polymerase / viral RNA genome replication / RNA-directed RNA polymerase activity / DNA-templated transcription 類似検索 - 分子機能 | ||||||||||||||||||
| 生物種 |  Sin Nombre orthohantavirus (ウイルス) Sin Nombre orthohantavirus (ウイルス) | ||||||||||||||||||
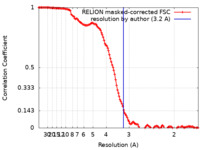
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.2 Å | ||||||||||||||||||
 データ登録者 データ登録者 | Meier K / Thorkelsson SR / Durieux Trouilleton Q / Vogel D / Yu D / Kosinski J / Cusack S / Malet H / Grunewald K / Quemin ERJ / Rosenthal M | ||||||||||||||||||
| 資金援助 |  ドイツ, ドイツ,  フランス, 5件 フランス, 5件
| ||||||||||||||||||
 引用 引用 |  ジャーナル: PLoS Pathog / 年: 2023 ジャーナル: PLoS Pathog / 年: 2023タイトル: Structural and functional characterization of the Sin Nombre virus L protein. 著者: Kristina Meier / Sigurdur R Thorkelsson / Quentin Durieux Trouilleton / Dominik Vogel / Dingquan Yu / Jan Kosinski / Stephen Cusack / Hélène Malet / Kay Grünewald / Emmanuelle R J Quemin / Maria Rosenthal /   要旨: The Bunyavirales order is a large and diverse group of segmented negative-strand RNA viruses. Several virus families within this order contain important human pathogens, including Sin Nombre virus ...The Bunyavirales order is a large and diverse group of segmented negative-strand RNA viruses. Several virus families within this order contain important human pathogens, including Sin Nombre virus (SNV) of the Hantaviridae. Despite the high epidemic potential of bunyaviruses, specific medical countermeasures such as vaccines or antivirals are missing. The multifunctional ~250 kDa L protein of hantaviruses, amongst other functional domains, harbors the RNA-dependent RNA polymerase (RdRp) and an endonuclease and catalyzes transcription as well as replication of the viral RNA genome, making it a promising therapeutic target. The development of inhibitors targeting these key processes requires a profound understanding of the catalytic mechanisms. Here, we established expression and purification protocols of the full-length SNV L protein bearing the endonuclease mutation K124A. We applied different biochemical in vitro assays to provide an extensive characterization of the different enzymatic functions as well as the capacity of the hantavirus L protein to interact with the viral RNA. By using single-particle cryo-EM, we obtained a 3D model including the L protein core region containing the RdRp, in complex with the 5' promoter RNA. This first high-resolution model of a New World hantavirus L protein shows striking similarity to related bunyavirus L proteins. The interaction of the L protein with the 5' RNA observed in the structural model confirms our hypothesis of protein-RNA binding based on our biochemical data. Taken together, this study provides an excellent basis for future structural and functional studies on the hantavirus L protein and for the development of antiviral compounds. | ||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_16670.map.gz emd_16670.map.gz | 11.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-16670-v30.xml emd-16670-v30.xml emd-16670.xml emd-16670.xml | 17.3 KB 17.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_16670_fsc.xml emd_16670_fsc.xml | 12.8 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_16670.png emd_16670.png | 78.1 KB | ||
| その他 |  emd_16670_half_map_1.map.gz emd_16670_half_map_1.map.gz emd_16670_half_map_2.map.gz emd_16670_half_map_2.map.gz | 140.7 MB 140.6 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-16670 http://ftp.pdbj.org/pub/emdb/structures/EMD-16670 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-16670 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-16670 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_16670_validation.pdf.gz emd_16670_validation.pdf.gz | 769.1 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_16670_full_validation.pdf.gz emd_16670_full_validation.pdf.gz | 768.6 KB | 表示 | |
| XML形式データ |  emd_16670_validation.xml.gz emd_16670_validation.xml.gz | 20.1 KB | 表示 | |
| CIF形式データ |  emd_16670_validation.cif.gz emd_16670_validation.cif.gz | 26.6 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16670 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16670 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16670 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16670 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8ci5MC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_16670.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_16670.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.85 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #2
| ファイル | emd_16670_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_16670_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Structure of Sin nombre virus L protein bound to 5' RNA
| 全体 | 名称: Structure of Sin nombre virus L protein bound to 5' RNA |
|---|---|
| 要素 |
|
-超分子 #1: Structure of Sin nombre virus L protein bound to 5' RNA
| 超分子 | 名称: Structure of Sin nombre virus L protein bound to 5' RNA タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Sin Nombre orthohantavirus (ウイルス) Sin Nombre orthohantavirus (ウイルス) |
| 分子量 | 理論値: 254 KDa |
-分子 #1: RNA-directed RNA polymerase L
| 分子 | 名称: RNA-directed RNA polymerase L / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Sin Nombre orthohantavirus (ウイルス) Sin Nombre orthohantavirus (ウイルス) |
| 分子量 | 理論値: 246.846797 KDa |
| 組換発現 | 生物種:  Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 配列 | 文字列: MEKYREIHQR VKEIPPGGAS ALECLDLLDR LYAVRHDVVD QMIKHDWSDN KDMERPIGQV LLMAGVPNDI IQGMEKKVIP TSPSGQILK SFFRMTPDNY KITGALIEFI EVTVTADVAK GIREAKLKYE SGLQFIESLL SQEHKKGNIN QVYKITFDVV A VKTDGSNI ...文字列: MEKYREIHQR VKEIPPGGAS ALECLDLLDR LYAVRHDVVD QMIKHDWSDN KDMERPIGQV LLMAGVPNDI IQGMEKKVIP TSPSGQILK SFFRMTPDNY KITGALIEFI EVTVTADVAK GIREAKLKYE SGLQFIESLL SQEHKKGNIN QVYKITFDVV A VKTDGSNI STQWPSRRND GVVQHMRLVQ ADINYVREHL IKPDERASLE AMFNLKFHVG GPKLRYFNIP DYKPQSLCQP EI TNLIQYC KHWLTEDHDF VFKEVTGNNV MSSFENNEDV YMSRYKESRK PRNFLLIQGS IQGPYLPSTI SSDQCDTRIG CLE VLKVHP ETPVQAIAVD MAYKYMELNR DEIINYYNPR VHFQATQSVK EPGTFKLGLS QLNPMSKSIL DQVGKHKSEK GLFG EPLES INISSQIQQN ECSRIIESIL SNLEINVGEV TMNLANPRKT TGVDELLGKF YENELSKYLI SILRKTAAWH IGHLI RDIT ESLIAHAGLK RSKYWSIHAY DHGGVILFIL PSKSLEVVGS YIRYFTVFKD GIGLIDEENL DSKVDIDGVQ WCFSKV MSI DLNRLLALNI AFEKALLATA TWFQYYTEDQ GHFPLQHALR SVFSFHFLLC VSQKMKICAI FDNLRYLIPA VTSLYSG YE LLIEKFFERP FKSALEVYLY NIIKALLISL AQNNKVRFYS KVRLLGLTVD HSTVGASGVY PSLMSRVVYK HYRSLISE A TTCFFLFEKG LHGNLNEEAK IHLETVEWAR KFEAKERKYG DILMREGYTI DAIRVGDVQV EQQLFCQEVV ELSAEELNK YLQAKSQVLS SNIMNKHWDK PYFSQTRNIS LKGMSGALQE DGHLAASVTL IEAIRFLNRS QTNPNVIDMY EQTKQHKAQA RIVRKYQRT EADRGFFITT LPTRVRLEII EDYYDAIARV VPEEYISYGG DKKILNIQTA LEKALRWASG SSEITTSTGN V IKFKRRLM YVSADATKWS PGDNSAKFKR FTQALYDGLS DEKLKCCVVD ALRHVYETEF FMSRKLHRYI DSMDEHSEAV QD FLDFFKG GVSATVKGNW LQGNLNKCSS LFGAAVSLLF RRIWAELFPE LECFFEFAHH SDDALFIYGY LEPEDDGTDW FLY VSQQIQ AGNYHWHAVN QEMWKSMFNL HEHLLLMGSI KVSPKKTTVS PTNAEFLSTF FEGCAVSIPF IKILLGSLSD LPGL GFFDD LAAAQSRCVK AMDLGASPQL AQLAVVICTS KVERLYGTAD GMVNSPVAFL KVTKAHVPIP LGGDGSMSIM ELATA GIGM ADKNILKQAF YSYKHTRRDG DRYVLGLFKF LMSLSEDVFQ HDRLGEFSFV GKVQWKVFTP KNEFEFYDQF SQSYLK SWT NQHPVYDYII PRGRDNLLVY LVRKLNDPSI VTAMTMQSPL QLRFRMQAKQ HMKVCKLDGE WVTFREVLAA ADSFATK YN PTEKDLDLFN TLVSCTFSKE YAWKDFLNEV RCEVVPTKHV HRSKIARTFT VREKDQAIQN PITAVIGYKY ASTVDEIS D VLDSSFFPDS LSADLQVMKE GVYRELGLDI GLPEVLKRIA PLLYKAGRSR VVIVEGNVEG TAESICSYWL RSMSLVKTI KVRPKKEVLR AVSLYSTKEN IGLQDDVAAT RLCIEVWRWC KANDQNVNDW LNALYFEKQT LMDWVERFRR KGVVPVDPEI QCIALLLYD VLGYKSVLQM QANRRAYSGK QYDAYCVQTY NEETKLYEGD LRVTFNFGLD CARLEIFWDK KEYILETSIT Q RHVLKLMM EEVTQELLRC GMRFKTEQVS HTKSLVLFKT ESGFEWGKPN VPCIVFKHCA LRTGLRTKQA INKEFMINVQ AD GFRAIAQ MDVESPRFLL AHAYHTLRDV RYQAVQAVGN VWFQTNQHKL FINPIISSGL LENFMKGLPA AIPPAAYSLI MNK AKISVD LFMFNELLAL VNPRNVLNLD GIEETSEGYS TVTSISSRQW SEEVSLMADD NIDDEEEFTI ALDDIDFEQI NLDE DIQHF LQDESAYTGD LTIQTEEVEV KRIRGVTRVL EPVKLIKSWV SKGLAIDKVY NPIGIVLMAR YMSKNYDFSK IPLAL LNPY DLTEFESVVK GWGETVNDRF LEVDNDAQRL IREKNILPED ILPDSLFSFR HVDVLLKRLF PRDPVSSFY UniProtKB: RNA-directed RNA polymerase L |
-分子 #2: RNA (5'-R(P*AP*GP*UP*AP*GP*UP*AP*GP*AP*CP*U)-3')
| 分子 | 名称: RNA (5'-R(P*AP*GP*UP*AP*GP*UP*AP*GP*AP*CP*U)-3') / タイプ: rna / ID: 2 / コピー数: 1 |
|---|---|
| 由来(天然) | 生物種:  Sin Nombre orthohantavirus (ウイルス) Sin Nombre orthohantavirus (ウイルス) |
| 分子量 | 理論値: 5.796515 KDa |
| 配列 | 文字列: UAGUAGUAGA CUCCGAGA |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.2 µm / 最小 デフォーカス(公称値): 0.8 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー




 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)