+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | The organise full-length structure of the fungal non-reducing polyketide synthase (NR-PKS) PksA | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Fungal non-reducing polyketide synthase (NR-PKS) / BIOSYNTHETIC PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報noranthrone synthase / aflatoxin biosynthetic process / norsolorinate anthrone synthase activity / fatty acid synthase activity / phosphopantetheine binding / fatty acid biosynthetic process / identical protein binding 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.6 Å | |||||||||
 データ登録者 データ登録者 | Munoz-Hernandez H / Maier T | |||||||||
| 資金援助 | European Union,  スイス, 2件 スイス, 2件
| |||||||||
 引用 引用 |  ジャーナル: To Be Published ジャーナル: To Be Publishedタイトル: CryoEM structure of the Aspergilus sp. fungal non-reducing polyketide synthase (NR-PKS) PksA at 2.6 Angstroms resolution 著者: Munoz-Hernandez H / Maier T | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_16630.map.gz emd_16630.map.gz | 107.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-16630-v30.xml emd-16630-v30.xml emd-16630.xml emd-16630.xml | 20.4 KB 20.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_16630.png emd_16630.png | 78.2 KB | ||
| Filedesc metadata |  emd-16630.cif.gz emd-16630.cif.gz | 7.5 KB | ||
| その他 |  emd_16630_half_map_1.map.gz emd_16630_half_map_1.map.gz emd_16630_half_map_2.map.gz emd_16630_half_map_2.map.gz | 200.5 MB 200.5 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-16630 http://ftp.pdbj.org/pub/emdb/structures/EMD-16630 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-16630 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-16630 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_16630_validation.pdf.gz emd_16630_validation.pdf.gz | 909.5 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_16630_full_validation.pdf.gz emd_16630_full_validation.pdf.gz | 909 KB | 表示 | |
| XML形式データ |  emd_16630_validation.xml.gz emd_16630_validation.xml.gz | 15.7 KB | 表示 | |
| CIF形式データ |  emd_16630_validation.cif.gz emd_16630_validation.cif.gz | 18.7 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16630 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16630 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16630 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16630 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8cg4MC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_16630.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_16630.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.82 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #1
| ファイル | emd_16630_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
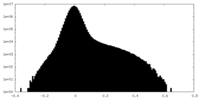
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_16630_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : The full-length Non-reducing polyketide synthase PksA
| 全体 | 名称: The full-length Non-reducing polyketide synthase PksA |
|---|---|
| 要素 |
|
-超分子 #1: The full-length Non-reducing polyketide synthase PksA
| 超分子 | 名称: The full-length Non-reducing polyketide synthase PksA タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 450 KDa |
-分子 #1: Norsolorinic acid synthase
| 分子 | 名称: Norsolorinic acid synthase / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO / EC番号: noranthrone synthase |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 237.74125 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MAHHHHHHHH HHGSTSGSGE QKLISEEDLG STSGSGDYKD DDDKLTSLYK KAGLENLYFQ GMAQSRQLFL FGDQTADFVP KLRSLLSVQ DSPILAAFLD QSHYVVRAQM LQSMNTVDHK LARTADLRQM VQKYVDGKLT PAFRTALVCL CQLGCFIREY E ESGNMYPQ ...文字列: MAHHHHHHHH HHGSTSGSGE QKLISEEDLG STSGSGDYKD DDDKLTSLYK KAGLENLYFQ GMAQSRQLFL FGDQTADFVP KLRSLLSVQ DSPILAAFLD QSHYVVRAQM LQSMNTVDHK LARTADLRQM VQKYVDGKLT PAFRTALVCL CQLGCFIREY E ESGNMYPQ PSDSYVLGFC MGSLAAVAVS CSRSLSELLP IAVQTVLIAF RLGLCALEMR DRVDGCSDDR GDPWSTIVWG LD PQQARDQ IEVFCRTTNV PQTRRPWISC ISKNAITLSG SPSTLRAFCA MPQMAQHRTA PIPICLPAHN GALFTQADIT TIL DTTPTT PWEQLPGQIP YISHVTGNVV QTSNYRDLIE VALSETLLEQ VRLDLVETGL PRLLQSRQVK SVTIVPFLTR MNET MSNIL PDSFISTETR TDTGRAIPAS GRPGAGKCKL AIVSMSGRFP ESPTTESFWD LLYKGLDVCK EVPRRRWDIN THVDP SGKA RNKGATKWGC WLDFSGDFDP RFFGISPKEA PQMDPAQRMA LMSTYEAMER AGLVPDTTPS TQRDRIGVFH GVTSND WME TNTAQNIDTY FITGGNRGFI PGRINFCFEF AGPSYTNDTA CSSSLAAIHL ACNSLWRGDC DTAVAGGTNM IYTPDGH TG LDKGFFLSRT GNCKPYDDKA DGYCRAEGVG TVFIKRLEDA LADNDPILGV ILDAKTNHSA MSESMTRPHV GAQIDNMT A ALNTTGLHPN DFSYIEMHGT GTQVGDAVEM ESVLSVFAPS ETARKADQPL FVGSAKANVG HGEGVSGVTS LIKVLMMMQ HDTIPPHCGI KPGSKINRNF PDLGARNVHI AFEPKPWPRT HTPRRVLINN FSAAGGNTAL IVEDAPERHW PTEKDPRSSH IVALSAHVG ASMKTNLERL HQYLLKNPHT DLAQLSYTTT ARRWHYLHRV SVTGASVEEV TRKLEMAIQN GDGVSRPKSK P KILFAFTG QGSQYATMGK QVYDAYPSFR EDLEKFDRLA QSHGFPSFLH VCTSPKGDVE EMAPVVVQLA ITCLQMALTN LM TSFGIRP DVTVGHSLGE FAALYAAGVL SASDVVYLVG QRAELLQERC QRGTHAMLAV KATPEALSQW IQDHDCEVAC ING PEDTVL SGTTKNVAEV QRAMTDNGIK CTLLKLPFAF HSAQVQPILD DFEALAQGAT FAKPQLLILS PLLRTEIHEQ GVVT PSYVA QHCRHTVDMA QALRSAREKG LIDDKTLVIE LGPKPLISGM VKMTLGDKIS TLPTLAPNKA IWPSLQKILT SVYTG GWDI NWKKYHAPFA SSQKVVDLPS YGWDLKDYYI PYQGDWCLHR HQQDCKCAAP GHEIKTADYQ VPPESTPHRP SKLDPS KEA FPEIKTTTTL HRVVEETTKP LGATLVVETD ISRKDVNGLA RGHLVDGIPL CTPSFYADIA MQVGQYSMQR LRAGHPG AG AIDGLVDVSD MVVDKALVPH GKGPQLLRTT LTMEWPPKAA ATTRSAKVKF ATYFADGKLD TEHASCTVRF TSDAQLKS L RRSVSEYKTH IRQLHDGHAK GQFMRYNRKT GYKLMSSMAR FNPDYMLLDY LVLNEAENEA ASGVDFSLGS SEGTFAAHP AHVDAITQVA GFAMNANDNV DIEKQVYVNH GWDSFQIYQP LDNSKSYQVY TKMGQAKEND LVHGDVVVLD GEQIVAFFRG LTLRSVPRG ALRVVLQTTV KKADRQLGFK TMPSPPPPTT TMPISPYKPA NTQVSSQAIP AEATHSHTPP QPKHSPVPET A GSAPAAKG VGVSNEKLDA VMRVVSEESG IALEELTDDS NFADMGIDSL SSMVIGSRFR EDLGLDLGPE FSLFIDCTTV RA LKDFMLG SGDAGSGSNV EDPPPSATPG INPETDWSSS ASDSIFASED HGHSSESGAD TGSPPALDLK PYCRPSTSVV LQG LPMVAR KTLFMLPDGG GSAFSYASLP RLKSDTAVVG LNCPYARDPE NMNCTHGAMI ESFCNEIRRR QPRGPYHLGG WSSG GAFAY VVAEALVNQG EEVHSLIIID APIPQAMEQL PRAFYEHCNS IGLFATQPGA SPDGSTEPPS YLIPHFTAVV DVMLD YKLA PLHARRMPKV GIVWAADTVM DERDAPKMKG MHFMIQKRTE FGPDGWDTIM PGASFDIVRA DGANHFTLMQ KEHVSI ISD LIDRVMA UniProtKB: Norsolorinic acid synthase |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.8 |
|---|---|
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 300 / 前処理 - タイプ: GLOW DISCHARGE |
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K2 QUANTUM (4k x 4k) 撮影したグリッド数: 1 / 実像数: 6479 / 平均電子線量: 60.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 最大 デフォーカス(補正後): 3.2 µm / 最小 デフォーカス(補正後): 0.9 µm / 照射モード: SPOT SCAN / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 0.6 µm / 倍率(公称値): 165000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT |
|---|---|
| 得られたモデル |  PDB-8cg4: |
 ムービー
ムービー コントローラー
コントローラー






 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)