+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | HK97 Portal Protein In situ (prohead II) | |||||||||
 マップデータ マップデータ | HK97 bacteriophage portal protein in prohead II as part of packaging complex. Symmetry = C12. | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | portal / HK97 / bacteriophage / packaging / VIRAL PROTEIN | |||||||||
| 機能・相同性 | Phage portal protein, HK97 / Bacteriophage/Gene transfer agent portal protein / Phage portal protein / viral capsid / Portal protein 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  Hendrixvirus (ウイルス) Hendrixvirus (ウイルス) | |||||||||
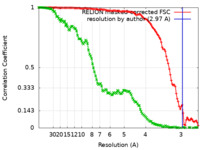
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.97 Å | |||||||||
 データ登録者 データ登録者 | Hawkins DEDP / Antson AA | |||||||||
| 資金援助 |  英国, 1件 英国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nucleic Acids Res / 年: 2023 ジャーナル: Nucleic Acids Res / 年: 2023タイトル: Insights into a viral motor: the structure of the HK97 packaging termination assembly. 著者: Dorothy E D P Hawkins / Oliver W Bayfield / Herman K H Fung / Daniel N Grba / Alexis Huet / James F Conway / Alfred A Antson /    要旨: Double-stranded DNA viruses utilise machinery, made of terminase proteins, to package viral DNA into the capsid. For cos bacteriophage, a defined signal, recognised by small terminase, flanks each ...Double-stranded DNA viruses utilise machinery, made of terminase proteins, to package viral DNA into the capsid. For cos bacteriophage, a defined signal, recognised by small terminase, flanks each genome unit. Here we present the first structural data for a cos virus DNA packaging motor, assembled from the bacteriophage HK97 terminase proteins, procapsids encompassing the portal protein, and DNA containing a cos site. The cryo-EM structure is consistent with the packaging termination state adopted after DNA cleavage, with DNA density within the large terminase assembly ending abruptly at the portal protein entrance. Retention of the large terminase complex after cleavage of the short DNA substrate suggests that motor dissociation from the capsid requires headful pressure, in common with pac viruses. Interestingly, the clip domain of the 12-subunit portal protein does not adhere to C12 symmetry, indicating asymmetry induced by binding of the large terminase/DNA. The motor assembly is also highly asymmetric, showing a ring of 5 large terminase monomers, tilted against the portal. Variable degrees of extension between N- and C-terminal domains of individual subunits suggest a mechanism of DNA translocation driven by inter-domain contraction and relaxation. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_16614.map.gz emd_16614.map.gz | 116.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-16614-v30.xml emd-16614-v30.xml emd-16614.xml emd-16614.xml | 16.1 KB 16.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_16614_fsc.xml emd_16614_fsc.xml emd_16614_fsc_2.xml emd_16614_fsc_2.xml | 11.3 KB 14.6 KB | 表示 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_16614.png emd_16614.png | 68.6 KB | ||
| マスクデータ |  emd_16614_msk_1.map emd_16614_msk_1.map | 125 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-16614.cif.gz emd-16614.cif.gz | 6.1 KB | ||
| その他 |  emd_16614_half_map_1.map.gz emd_16614_half_map_1.map.gz emd_16614_half_map_2.map.gz emd_16614_half_map_2.map.gz | 98.1 MB 98.1 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-16614 http://ftp.pdbj.org/pub/emdb/structures/EMD-16614 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-16614 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-16614 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8cezMC  8cfaC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_16614.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_16614.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | HK97 bacteriophage portal protein in prohead II as part of packaging complex. Symmetry = C12. | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.34 Å | ||||||||||||||||||||||||||||||||||||
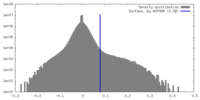
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_16614_msk_1.map emd_16614_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
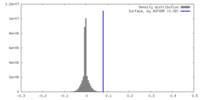
| 密度ヒストグラム |
-ハーフマップ: HK97 bacteriophage portal protein in prohead II as...
| ファイル | emd_16614_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | HK97 bacteriophage portal protein in prohead II as part of packaging complex. Symmetry = C12. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: HK97 bacteriophage portal protein in prohead II as...
| ファイル | emd_16614_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | HK97 bacteriophage portal protein in prohead II as part of packaging complex. Symmetry = C12. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Hendrixvirus
| 全体 | 名称:  Hendrixvirus (ウイルス) Hendrixvirus (ウイルス) |
|---|---|
| 要素 |
|
-超分子 #1: Hendrixvirus
| 超分子 | 名称: Hendrixvirus / タイプ: virus / ID: 1 / 親要素: 0 / 含まれる分子: all 詳細: Proheads were produced by infection of E. coli 594 cells with HK97 amber mutant amC2, propagated using Escherichia LE392 cells. NCBI-ID: 2169654 / 生物種: Hendrixvirus / Sci species strain: HK97 / ウイルスタイプ: VIRION / ウイルス・単離状態: STRAIN / ウイルス・エンベロープ: No / ウイルス・中空状態: No |
|---|---|
| 宿主 | 生物種:  Escherichia phage EcSzw-2 (ファージ) / 株: 594 Escherichia phage EcSzw-2 (ファージ) / 株: 594 |
| 分子量 | 理論値: 560 KDa |
-分子 #1: Portal protein
| 分子 | 名称: Portal protein / タイプ: protein_or_peptide / ID: 1 / コピー数: 12 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Hendrixvirus (ウイルス) Hendrixvirus (ウイルス) |
| 分子量 | 理論値: 47.318488 KDa |
| 組換発現 | 生物種:  Escherichia phage EcSzw-2 (ファージ) Escherichia phage EcSzw-2 (ファージ) |
| 配列 | 文字列: MEEPKYTIDL RTNNGWWARL KSWFVGGRLV TPNQGSQTGP VSAHGYLGDS SINDERILQI STVWRCVSLI STLTACLPLD VFETDQNDN RKKVDLSNPL ARLLRYSPNQ YMTAQEFREA MTMQLCFYGN AYALVDRNSA GDVISLLPLQ SANMDVKLVG K KVVYRYQR ...文字列: MEEPKYTIDL RTNNGWWARL KSWFVGGRLV TPNQGSQTGP VSAHGYLGDS SINDERILQI STVWRCVSLI STLTACLPLD VFETDQNDN RKKVDLSNPL ARLLRYSPNQ YMTAQEFREA MTMQLCFYGN AYALVDRNSA GDVISLLPLQ SANMDVKLVG K KVVYRYQR DSEYADFSQK EIFHLKGFGF TGLVGLSPIA FACKSAGVAV AMEDQQRDFF ANGAKSPQIL STGEKVLTEQ QR SQVEENF KEIAGGPVKK RLWILEAGFS TSAIGVTPQD AEMMASRKFQ VSELARFFGV PPHLVGDVEK STSWGSGIEQ QNL GFLQYT LQPYISRWEN SIQRWLIPSK DVGRLHAEHN LDGLLRGDSA SRAAFMKAMG ESGLRTINEM RRTDNMPPLP GGDV AMRQA QYVPITDLGT NKEPRNNGA UniProtKB: Portal protein |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 詳細: final buffer = 20 mM Tris pH 7.5, 10 mM MgCl2, 50 mM KGlu, 5 mM ATP-gamma-S |
|---|---|
| グリッド | モデル: Quantifoil / 材質: COPPER / メッシュ: 200 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: CONTINUOUS / 支持フィルム - Film thickness: 0.2 / 前処理 - タイプ: PLASMA CLEANING / 前処理 - 時間: 60 sec. |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 90 % / 装置: FEI VITROBOT MARK IV |
| 詳細 | Packaging reactions 30 nM hk97 proheads, 2.5 micromolar large terminase, 5 micromolar small terminase and 30 nM DNA, 75 micromolar ATP were incubated for 2 mins then 5 micromolar ATP-gamma-S was added. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 撮影したグリッド数: 1 / 平均電子線量: 40.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 0.5 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー








 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)