+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | 21S ribosomal precursors induced by heat shock. | |||||||||
 マップデータ マップデータ | 21S cryo-EM map, postprocessed and masked | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | 21S / DnaK / Heat shock / Ribosome assembly / RIBOSOME | |||||||||
| 生物種 |  | |||||||||
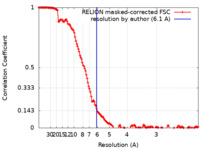
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 6.1 Å | |||||||||
 データ登録者 データ登録者 | Giudice E / Georgeault S / Lavigne R / Pineau C / Trautwetter A / Ermel G / Blanco C / Gillet R | |||||||||
| 資金援助 |  フランス, 2件 フランス, 2件
| |||||||||
 引用 引用 |  ジャーナル: Int J Mol Sci / 年: 2023 ジャーナル: Int J Mol Sci / 年: 2023タイトル: Purification and Characterization of Authentic 30S Ribosomal Precursors Induced by Heat Shock. 著者: Emmanuel Giudice / Sylvie Georgeault / Régis Lavigne / Charles Pineau / Annie Trautwetter / Gwennola Ermel / Carlos Blanco / Reynald Gillet /  要旨: Ribosome biogenesis is a complex and multistep process that depends on various assembly factors. To understand this process and identify the ribosome assembly intermediates, most studies have set out ...Ribosome biogenesis is a complex and multistep process that depends on various assembly factors. To understand this process and identify the ribosome assembly intermediates, most studies have set out to delete or deplete these assembly factors. Instead, we took advantage of the impact of heat stress (45 °C) on the late stages of the biogenesis of the 30S ribosomal subunit to explore authentic precursors. Under these conditions, reduced levels of the DnaK chaperone proteins devoted to ribosome assembly lead to the transient accumulation of 21S ribosomal particles, which are 30S precursors. We constructed strains with different affinity tags on one early and one late 30S ribosomal protein and purified the 21S particles that form under heat shock. A combination of relative quantification using mass spectrometry-based proteomics and cryo-electron microscopy (cryo-EM) was then used to determine their protein contents and structures. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_16556.map.gz emd_16556.map.gz | 17.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-16556-v30.xml emd-16556-v30.xml emd-16556.xml emd-16556.xml | 37.5 KB 37.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_16556_fsc.xml emd_16556_fsc.xml | 14.3 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_16556.png emd_16556.png | 61.4 KB | ||
| マスクデータ |  emd_16556_msk_1.map emd_16556_msk_1.map emd_16556_msk_2.map emd_16556_msk_2.map emd_16556_msk_3.map emd_16556_msk_3.map | 244.1 MB 244.1 MB 244.1 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-16556.cif.gz emd-16556.cif.gz | 5.5 KB | ||
| その他 |  emd_16556_additional_1.map.gz emd_16556_additional_1.map.gz emd_16556_additional_2.map.gz emd_16556_additional_2.map.gz emd_16556_additional_3.map.gz emd_16556_additional_3.map.gz emd_16556_additional_4.map.gz emd_16556_additional_4.map.gz emd_16556_additional_5.map.gz emd_16556_additional_5.map.gz emd_16556_additional_6.map.gz emd_16556_additional_6.map.gz emd_16556_additional_7.map.gz emd_16556_additional_7.map.gz emd_16556_additional_8.map.gz emd_16556_additional_8.map.gz emd_16556_additional_9.map.gz emd_16556_additional_9.map.gz emd_16556_half_map_1.map.gz emd_16556_half_map_1.map.gz emd_16556_half_map_2.map.gz emd_16556_half_map_2.map.gz | 193.7 MB 183.6 MB 14.9 MB 183 MB 183.7 MB 161.5 MB 11.9 MB 161.5 MB 161.5 MB 194.3 MB 194.6 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-16556 http://ftp.pdbj.org/pub/emdb/structures/EMD-16556 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-16556 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-16556 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_16556_validation.pdf.gz emd_16556_validation.pdf.gz | 776.8 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_16556_full_validation.pdf.gz emd_16556_full_validation.pdf.gz | 776.4 KB | 表示 | |
| XML形式データ |  emd_16556_validation.xml.gz emd_16556_validation.xml.gz | 21.6 KB | 表示 | |
| CIF形式データ |  emd_16556_validation.cif.gz emd_16556_validation.cif.gz | 28.3 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16556 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16556 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16556 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16556 | HTTPS FTP |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_16556.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_16556.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | 21S cryo-EM map, postprocessed and masked | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.11 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
+マスク #1
+マスク #2
+マスク #3
+追加マップ: 21S cryo-EM map (not postprocessed)
+追加マップ: multibody analysis : 21S Body first half map
+追加マップ: multibody analysis : 21S Body, postprocessed and masked
+追加マップ: multibody analysis : 21S Body (not postprocessed)
+追加マップ: multibody analysis : 21S Body second half map
+追加マップ: multibody analysis : 21S Head (not postprocessed)
+追加マップ: multibody analysis : 21S Head, postprocessed and masked
+追加マップ: multibody analysis : 21S Head first half map
+追加マップ: multibody analysis : 21S Head second half map
+ハーフマップ: 21S first half map
+ハーフマップ: 21S second half map
- 試料の構成要素
試料の構成要素
-全体 : 21S ribosomal precursors induced by heat shock.
| 全体 | 名称: 21S ribosomal precursors induced by heat shock. |
|---|---|
| 要素 |
|
-超分子 #1: 21S ribosomal precursors induced by heat shock.
| 超分子 | 名称: 21S ribosomal precursors induced by heat shock. / タイプ: complex / ID: 1 / 親要素: 0 |
|---|---|
| 由来(天然) | 生物種:  |
-超分子 #2: 16S ribosomal RNA
| 超分子 | 名称: 16S ribosomal RNA / タイプ: complex / ID: 2 / 親要素: 1 |
|---|---|
| 由来(天然) | 生物種:  |
-超分子 #3: 21S Ribosomal proteins
| 超分子 | 名称: 21S Ribosomal proteins / タイプ: complex / ID: 3 / 親要素: 1 詳細: uS3 uS4 uS5 bS6 uS7 uS8 uS9 uS10 uS11 uS12 uS13 uS14 uS15 bS16 uS17 bS18 uS19 bS20 |
|---|---|
| 由来(天然) | 生物種:  |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 構成要素:
| ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| グリッド | モデル: Quantifoil R2/2 / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 20 sec. / 前処理 - 雰囲気: AIR / 前処理 - 気圧: 0.04 kPa / 詳細: 30mA | ||||||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 295.15 K / 装置: FEI VITROBOT MARK III / 詳細: blot for 2s before plunging. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | 球面収差補正装置: Microscope was modified with a Cs corrector |
| 撮影 | フィルム・検出器のモデル: FEI FALCON II (4k x 4k) 検出モード: INTEGRATING / デジタル化 - サイズ - 横: 4096 pixel / デジタル化 - サイズ - 縦: 4096 pixel / 撮影したグリッド数: 1 / 実像数: 922 / 平均露光時間: 1.0 sec. / 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 100.0 µm / 最大 デフォーカス(補正後): 2.7 µm / 最小 デフォーカス(補正後): 0.4 µm / 倍率(補正後): 120443 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 0.0023 mm / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 1.5 µm / 倍率(公称値): 59000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | プロトコル: RIGID BODY FIT |
|---|
 ムービー
ムービー コントローラー
コントローラー




 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)