+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | cryo-EM structure of BG505 SOSIP.664 HIV-1 Env trimer in complex with bNAbs EPTC112 and 3BNC117 | ||||||||||||
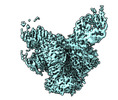
 マップデータ マップデータ | Overall cryo-EM map of BG505 SOSIP.664 HIV-1 Env trimer in complex with bNAbs EPTC112 and 3BNC117 | ||||||||||||
 試料 試料 |
| ||||||||||||
 キーワード キーワード | HIV / post-treatment controller / broadly neutralizing antibodies / V3-site / IMMUNE SYSTEM | ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報positive regulation of plasma membrane raft polarization / positive regulation of receptor clustering / host cell endosome membrane / clathrin-dependent endocytosis of virus by host cell / viral protein processing / fusion of virus membrane with host plasma membrane / fusion of virus membrane with host endosome membrane / viral envelope / virion attachment to host cell / host cell plasma membrane ...positive regulation of plasma membrane raft polarization / positive regulation of receptor clustering / host cell endosome membrane / clathrin-dependent endocytosis of virus by host cell / viral protein processing / fusion of virus membrane with host plasma membrane / fusion of virus membrane with host endosome membrane / viral envelope / virion attachment to host cell / host cell plasma membrane / virion membrane / structural molecule activity / membrane 類似検索 - 分子機能 | ||||||||||||
| 生物種 |  Homo sapiens (ヒト) / Homo sapiens (ヒト) /   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) | ||||||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.2 Å | ||||||||||||
 データ登録者 データ登録者 | Baquero E / Molinos-Albert L / Mouquet H | ||||||||||||
| 資金援助 |  フランス, フランス,  米国, 3件 米国, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Cell Host Microbe / 年: 2023 ジャーナル: Cell Host Microbe / 年: 2023タイトル: Anti-V1/V3-glycan broadly HIV-1 neutralizing antibodies in a post-treatment controller. 著者: Luis M Molinos-Albert / Eduard Baquero / Mélanie Bouvin-Pley / Valérie Lorin / Caroline Charre / Cyril Planchais / Jordan D Dimitrov / Valérie Monceaux / Matthijn Vos / / Laurent ...著者: Luis M Molinos-Albert / Eduard Baquero / Mélanie Bouvin-Pley / Valérie Lorin / Caroline Charre / Cyril Planchais / Jordan D Dimitrov / Valérie Monceaux / Matthijn Vos / / Laurent Hocqueloux / Jean-Luc Berger / Michael S Seaman / Martine Braibant / Véronique Avettand-Fenoël / Asier Sáez-Cirión / Hugo Mouquet /   要旨: HIV-1 broadly neutralizing antibodies (bNAbs) can decrease viremia but are usually unable to counteract autologous viruses escaping the antibody pressure. Nonetheless, bNAbs may contribute to natural ...HIV-1 broadly neutralizing antibodies (bNAbs) can decrease viremia but are usually unable to counteract autologous viruses escaping the antibody pressure. Nonetheless, bNAbs may contribute to natural HIV-1 control in individuals off antiretroviral therapy (ART). Here, we describe a bNAb B cell lineage elicited in a post-treatment controller (PTC) that exhibits broad seroneutralization and show that a representative antibody from this lineage, EPTC112, targets a quaternary epitope in the glycan-V3 loop supersite of the HIV-1 envelope glycoprotein. The cryo-EM structure of EPTC112 complexed with soluble BG505 SOSIP.664 envelope trimers revealed interactions with N301- and N156-branched N-glycans and the GDIR V3 loop motif. Although the sole contemporaneous virus circulating in this PTC was resistant to EPTC112, it was potently neutralized by autologous plasma IgG antibodies. Our findings illuminate how cross-neutralizing antibodies can alter the HIV-1 infection course in PTCs and may control viremia off-ART, supporting their role in functional HIV-1 cure strategies. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_16493.map.gz emd_16493.map.gz | 230.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-16493-v30.xml emd-16493-v30.xml emd-16493.xml emd-16493.xml | 25.7 KB 25.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_16493.png emd_16493.png | 135.1 KB | ||
| Filedesc metadata |  emd-16493.cif.gz emd-16493.cif.gz | 7.1 KB | ||
| その他 |  emd_16493_additional_1.map.gz emd_16493_additional_1.map.gz emd_16493_additional_2.map.gz emd_16493_additional_2.map.gz emd_16493_half_map_1.map.gz emd_16493_half_map_1.map.gz emd_16493_half_map_2.map.gz emd_16493_half_map_2.map.gz | 229.7 MB 229.7 MB 226.7 MB 226.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-16493 http://ftp.pdbj.org/pub/emdb/structures/EMD-16493 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-16493 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-16493 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_16493_validation.pdf.gz emd_16493_validation.pdf.gz | 1022.7 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_16493_full_validation.pdf.gz emd_16493_full_validation.pdf.gz | 1022.3 KB | 表示 | |
| XML形式データ |  emd_16493_validation.xml.gz emd_16493_validation.xml.gz | 15.9 KB | 表示 | |
| CIF形式データ |  emd_16493_validation.cif.gz emd_16493_validation.cif.gz | 18.8 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16493 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16493 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16493 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16493 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8c8tMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_16493.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_16493.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Overall cryo-EM map of BG505 SOSIP.664 HIV-1 Env trimer in complex with bNAbs EPTC112 and 3BNC117 | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.86 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: Local refined cryo-EM map of first bNAbs EPTC112...
| ファイル | emd_16493_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
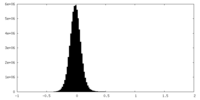
| 注釈 | Local refined cryo-EM map of first bNAbs EPTC112 bound to BG505 SOSIP.664 HIV-1 Env trimer | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
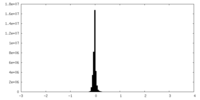
| 密度ヒストグラム |
-追加マップ: Local refined cryo-EM map of second bNAbs EPTC112...
| ファイル | emd_16493_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
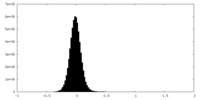
| 注釈 | Local refined cryo-EM map of second bNAbs EPTC112 bound to BG505 SOSIP.664 HIV-1 Env trimer | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
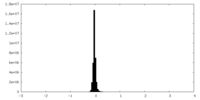
| 密度ヒストグラム |
-ハーフマップ: Half cryo-EM map A of BG505 SOSIP.664 HIV-1...
| ファイル | emd_16493_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
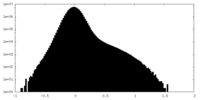
| 注釈 | Half cryo-EM map A of BG505 SOSIP.664 HIV-1 Env trimer in complex with bNAbs EPTC112 and 3BNC117 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
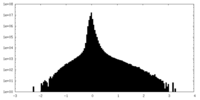
| 密度ヒストグラム |
-ハーフマップ: Half cryo-EM map B of BG505 SOSIP.664 HIV-1...
| ファイル | emd_16493_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
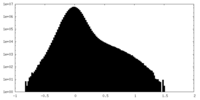
| 注釈 | Half cryo-EM map B of BG505 SOSIP.664 HIV-1 Env trimer in complex with bNAbs EPTC112 and 3BNC117 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
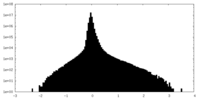
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Ternary complex of Bg505 Sosip.664 HIV-1 Env with bNAbs EPTC112 a...
| 全体 | 名称: Ternary complex of Bg505 Sosip.664 HIV-1 Env with bNAbs EPTC112 and 3BNC117 |
|---|---|
| 要素 |
|
-超分子 #1: Ternary complex of Bg505 Sosip.664 HIV-1 Env with bNAbs EPTC112 a...
| 超分子 | 名称: Ternary complex of Bg505 Sosip.664 HIV-1 Env with bNAbs EPTC112 and 3BNC117 タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#6 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: Envelope glycoprotein gp160
| 分子 | 名称: Envelope glycoprotein gp160 / タイプ: protein_or_peptide / ID: 1 / 詳細: Gp120 Bg505 Sosip.664 T332n / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) |
| 分子量 | 理論値: 52.5515 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: LWVTVYYGVP VWKDAETTLF CASDAKAYET EKHNVWATHA CVPTDPNPQE IHLENVTEEF NMWKNNMVEQ MHTDIISLWD QSLKPCVKL TPLCVTLQCT NVTNNITDDM RGELKNCSFN MTTELRDKKQ KVYSLFYRLD VVQINENQGN RSNNSNKEYR L INCNTSAI ...文字列: LWVTVYYGVP VWKDAETTLF CASDAKAYET EKHNVWATHA CVPTDPNPQE IHLENVTEEF NMWKNNMVEQ MHTDIISLWD QSLKPCVKL TPLCVTLQCT NVTNNITDDM RGELKNCSFN MTTELRDKKQ KVYSLFYRLD VVQINENQGN RSNNSNKEYR L INCNTSAI TQACPKVSFE PIPIHYCAPA GFAILKCKDK KFNGTGPCPS VSTVQCTHGI KPVVSTQLLL NGSLAEEEVM IR SENITNN AKNILVQFNT PVQINCTRPN NNTRKSIRIG PGQAFYATGD IIGDIRQAHC NVSKATWNET LGKVVKQLRK HFG NNTIIR FANSSGGDLE VTTHSFNCGG EFFYCNTSGL FNSTWISNTS VQGSNSTGSN DSITLPCRIK QIINMWQRIG QAMY APPIQ GVIRCVSNIT GLILTRDGGS TNSTTETFRP GGGDMRDNWR SELYKYKVVK IEPLGVAPTR CKRR UniProtKB: Envelope glycoprotein gp160 |
-分子 #2: Gp41 Bg505 T332n Sosip.664
| 分子 | 名称: Gp41 Bg505 T332n Sosip.664 / タイプ: protein_or_peptide / ID: 2 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) |
| 分子量 | 理論値: 16.431627 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: LGFLGAAGST MGAASMTLTV QARNLLSGIV QQQSNLLRAP EAQQHLLKLT VWGIKQLQAR VLAVERYLRD QQLLGIWGCS GKLICCTNV PWNSSWSNRN LSEIWDNMTW LQWDKEISNY TQIIYGLLEE SQNQQEKNEQ DLLALD |
-分子 #3: IgG EPTC112 Fab fragment heavy chain
| 分子 | 名称: IgG EPTC112 Fab fragment heavy chain / タイプ: protein_or_peptide / ID: 3 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 25.320389 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: QVQLLESGPG LVRPSETLTL TCSVFNSRVS GYYYSWIRQP PGRGLEWIAS THFSLRPSRN PSLLSRVTTS IDTERYQVFL NMRSVTAAD TAVYFCARGD ASGWRADYFP HWGQGTLVVV SSASTKGPSV FPLAPSSKST SGGTAALGCL VKDYFPEPVT V SWNSGALT ...文字列: QVQLLESGPG LVRPSETLTL TCSVFNSRVS GYYYSWIRQP PGRGLEWIAS THFSLRPSRN PSLLSRVTTS IDTERYQVFL NMRSVTAAD TAVYFCARGD ASGWRADYFP HWGQGTLVVV SSASTKGPSV FPLAPSSKST SGGTAALGCL VKDYFPEPVT V SWNSGALT SGVHTFPAVL QSSGLYSLSS VVTVPSSSLG TQTYICNVNH KPSNTKVDKR VEPKSCDKTH HHHHH |
-分子 #4: IgG 3BNC117 Fab heavy chain
| 分子 | 名称: IgG 3BNC117 Fab heavy chain / タイプ: protein_or_peptide / ID: 4 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 24.640484 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: QVQLLQSGAA VTKPGASVRV SCEASGYNIR DYFIHWWRQA PGQGLQWVGW INPKTGQPNN PRQFQGRVSL TRHASWDFDT FSFYMDLKA LRSDDTAVYF CARQRSDYWD FDVWGSGTQV TVSSASTKGP SVFPLAPSSK STSGGTAALG CLVKDYFPEP V TVSWNSGA ...文字列: QVQLLQSGAA VTKPGASVRV SCEASGYNIR DYFIHWWRQA PGQGLQWVGW INPKTGQPNN PRQFQGRVSL TRHASWDFDT FSFYMDLKA LRSDDTAVYF CARQRSDYWD FDVWGSGTQV TVSSASTKGP SVFPLAPSSK STSGGTAALG CLVKDYFPEP V TVSWNSGA LTSGVHTFPA VLQSSGLYSL SSVVTVPSSS LGTQTYICNV NHKPSNTKVD KKVEPKSC |
-分子 #5: IgG 3BNC117 Fab Light Chain
| 分子 | 名称: IgG 3BNC117 Fab Light Chain / タイプ: protein_or_peptide / ID: 5 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 23.022658 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: DIQMTQSPSS LSASVGDTVT ITCQANGYLN WYQQRRGKAP KLLIYDGSKL ERGVPSRFSG RRWGQEYNLT INNLQPEDIA TYFCQVYEF VVPGTRLDLK RTVAAPSVFI FPPSDEQLKS GTASVVCLLN NFYPREAKVQ WKVDNALQSG NSQESVTEQD S KDSTYSLS ...文字列: DIQMTQSPSS LSASVGDTVT ITCQANGYLN WYQQRRGKAP KLLIYDGSKL ERGVPSRFSG RRWGQEYNLT INNLQPEDIA TYFCQVYEF VVPGTRLDLK RTVAAPSVFI FPPSDEQLKS GTASVVCLLN NFYPREAKVQ WKVDNALQSG NSQESVTEQD S KDSTYSLS STLTLSKADY EKHKVYACEV THQGLSSPVT KSFNRGEC |
-分子 #6: IgG EPTC112 Fab Light Chain
| 分子 | 名称: IgG EPTC112 Fab Light Chain / タイプ: protein_or_peptide / ID: 6 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 22.680932 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: QSVLTQPPSA SGSPGQSVTI SCTGTSSDIG ASDYVSWYQQ YPGEAPKVII YDVTKRPSGV PDRFSGSKSG TTASLTVSGL QAEDEADYY CSSDAGRHTL LFGGGTKVTV LGQPKAAPSV TLFPPSSEEL QANKATLVCL ISDFYPGAVT VAWKADSSPV K AGVETTTP ...文字列: QSVLTQPPSA SGSPGQSVTI SCTGTSSDIG ASDYVSWYQQ YPGEAPKVII YDVTKRPSGV PDRFSGSKSG TTASLTVSGL QAEDEADYY CSSDAGRHTL LFGGGTKVTV LGQPKAAPSV TLFPPSSEEL QANKATLVCL ISDFYPGAVT VAWKADSSPV K AGVETTTP SKQSNNKYAA SSYLSLTPEQ WKSHRSYSCQ VTHEGSTVEK TVAPTECS |
-分子 #10: 2-acetamido-2-deoxy-beta-D-glucopyranose
| 分子 | 名称: 2-acetamido-2-deoxy-beta-D-glucopyranose / タイプ: ligand / ID: 10 / コピー数: 24 / 式: NAG |
|---|---|
| 分子量 | 理論値: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 / 詳細: PBS |
|---|---|
| グリッド | モデル: EMS Lacey Carbon / 材質: COPPER / 前処理 - タイプ: GLOW DISCHARGE |
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 1.0 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: PDB ENTRY PDBモデル - PDB ID: |
|---|---|
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 3.2 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 251591 |
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
 ムービー
ムービー コントローラー
コントローラー






 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)