+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of BoNT/Wo-NTNH complex | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | The complex of botulinum neurotoxin-like protein from Weissella oryzae and its non-toxic non-hemagglutinin partner. / TOXIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報bontoxilysin / metalloendopeptidase activity / proteolysis / zinc ion binding / metal ion binding 類似検索 - 分子機能 | |||||||||
| 生物種 |  Weissella oryzae (バクテリア) Weissella oryzae (バクテリア) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.98 Å | |||||||||
 データ登録者 データ登録者 | Kosenina S / Skerlova J / Stenmark P | |||||||||
| 資金援助 |  スウェーデン, 2件 スウェーデン, 2件
| |||||||||
 引用 引用 |  ジャーナル: FEBS J / 年: 2024 ジャーナル: FEBS J / 年: 2024タイトル: The cryo-EM structure of the BoNT/Wo-NTNH complex reveals two immunoglobulin-like domains. 著者: Sara Košenina / Jana Škerlová / Sicai Zhang / Min Dong / Pål Stenmark /    要旨: The botulinum neurotoxin-like toxin from Weissella oryzae (BoNT/Wo) is one of the BoNT-like toxins recently identified outside of the Clostridium genus. We show that, like the canonical BoNTs, ...The botulinum neurotoxin-like toxin from Weissella oryzae (BoNT/Wo) is one of the BoNT-like toxins recently identified outside of the Clostridium genus. We show that, like the canonical BoNTs, BoNT/Wo forms a complex with its non-toxic non-hemagglutinin (NTNH) partner, which in traditional BoNT serotypes protects the toxin from proteases and the acidic environment of the hosts' guts. We here report the cryo-EM structure of the 300 kDa BoNT/Wo-NTNH/Wo complex together with pH stability studies of the complex. The structure reveals molecular details of the toxin's interactions with its protective partner. The overall structural arrangement is similar to other reported BoNT-NTNH complexes, but NTNH/Wo uniquely contains two extra bacterial immunoglobulin-like (Big) domains on the C-terminus. Although the function of these Big domains is unknown, they are structurally most similar to bacterial proteins involved in adhesion to host cells. In addition, the BoNT/Wo protease domain contains an internal disulfide bond not seen in other BoNTs. Mass photometry analysis revealed that the BoNT/Wo-NTNH/Wo complex is stable under acidic conditions and may dissociate at neutral to basic pH. These findings established that BoNT/Wo-NTNH/Wo shares the general fold of canonical BoNT-NTNH complexes. The presence of unique structural features suggests that it may have an alternative mode of activation, translocation and recognition of host cells, raising interesting questions about the activity and the mechanism of action of BoNT/Wo as well as about its target environment, receptors and substrates. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_16475.map.gz emd_16475.map.gz | 118 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-16475-v30.xml emd-16475-v30.xml emd-16475.xml emd-16475.xml | 21 KB 21 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_16475_fsc.xml emd_16475_fsc.xml | 14.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_16475.png emd_16475.png | 90.4 KB | ||
| Filedesc metadata |  emd-16475.cif.gz emd-16475.cif.gz | 7.4 KB | ||
| その他 |  emd_16475_half_map_1.map.gz emd_16475_half_map_1.map.gz emd_16475_half_map_2.map.gz emd_16475_half_map_2.map.gz | 116.1 MB 116.1 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-16475 http://ftp.pdbj.org/pub/emdb/structures/EMD-16475 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-16475 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-16475 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_16475_validation.pdf.gz emd_16475_validation.pdf.gz | 1002.3 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_16475_full_validation.pdf.gz emd_16475_full_validation.pdf.gz | 1001.8 KB | 表示 | |
| XML形式データ |  emd_16475_validation.xml.gz emd_16475_validation.xml.gz | 18.7 KB | 表示 | |
| CIF形式データ |  emd_16475_validation.cif.gz emd_16475_validation.cif.gz | 24.7 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16475 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16475 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16475 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16475 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8c8gMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_16475.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_16475.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.82 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #1
| ファイル | emd_16475_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
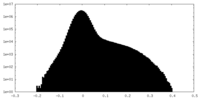
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_16475_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Botulinum neurotoxin-like protein from Weissella oryzae in comple...
| 全体 | 名称: Botulinum neurotoxin-like protein from Weissella oryzae in complex with its non-toxic non-hemagglutinin partner protein |
|---|---|
| 要素 |
|
-超分子 #1: Botulinum neurotoxin-like protein from Weissella oryzae in comple...
| 超分子 | 名称: Botulinum neurotoxin-like protein from Weissella oryzae in complex with its non-toxic non-hemagglutinin partner protein タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Weissella oryzae (バクテリア) Weissella oryzae (バクテリア) |
-分子 #1: Putative botulinum-like toxin Wo
| 分子 | 名称: Putative botulinum-like toxin Wo / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO / EC番号: bontoxilysin |
|---|---|
| 由来(天然) | 生物種:  Weissella oryzae (バクテリア) Weissella oryzae (バクテリア) |
| 分子量 | 理論値: 156.964484 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MDVLEMFDVN YESPILESFD STTQSLNDVH VFMSRIQMSA YDADGEGRIE YRNLKLYEIS SGIFISTDRL DTGASGVEDD HEMVDYYSS ARLTREFLGE SLDSQKSDYF EGIKKVFSFY KNKCNESRYI KEFFEEIQFR NICGFPKQAG TSSTDIFDQF N SVDVLLQD ...文字列: MDVLEMFDVN YESPILESFD STTQSLNDVH VFMSRIQMSA YDADGEGRIE YRNLKLYEIS SGIFISTDRL DTGASGVEDD HEMVDYYSS ARLTREFLGE SLDSQKSDYF EGIKKVFSFY KNKCNESRYI KEFFEEIQFR NICGFPKQAG TSSTDIFDQF N SVDVLLQD PVTSVWNKKV GSKKANIVII PPATNLPITE ACATAGFQPE GFPKLGSGSF FTVQFDPFFS TRFKAHETDD VA LLDPTLT LLHEMTHGLH FQKGIANPVN RSGETPAWAT TWGRVTGDND AFKETPMEEL LTFNKHTIDD DIEISDHLKS TYI GFLYNG RNEDDPTESV DGVYQNVSSF LNQYRGFEIS SDFQHFIESC YGVKYNQESK KFIVNPRNIK RYVQDGFFID EAKF ARILN IKTRSYYTLM PDNLGVWSYR VDILNRLRET FDEDRGLLSQ ELDFHTALTP VVSENPALEL EVAGMQRMVS LPKIK ASYL PSDIKIKNFT GQKISHDTIL DTNISGIIIS KIKYKSDFVV DESMPRSSLN TTNYNLSPIK GTKFETDIRD KTSVKV TVS EITAPMINHV MKLDNSKVLT ERPSLNEDLE ETFKNTKDVY IPKTTAMMKL KEGADQTLGA VGFAVWSGQI LEDLYNL AQ KKEVSIDQIK DDLMSILPFY CAYKNLSAEK YEQAFANATL DAFLIFATDG GGFAGLGITV GAIAINSMYA KAETMEAY D SMFGKYVDQY QNDIKNFTLN AYVQWENNIL SRLWNESRLA ITGFRNMLKT VKTVMEFDAT NQAYSEEDRK IIKAKCEEI FSEFPMLMQT FAKNSMTANL ENASKIFNDI VWQKIKEELD QYVIDSKKYF LDSLEEAYNN GSISAESYYK YQTEAREKFV SPREVIDLY IAAHDTVVKR KRYIRRYSRK YDLATDFKGN TVHLNGLGEG TQDIQDLYGN YSVYADKKTV STQEGHFDQT I KIAKDTNT INKVVLAVSS NNGKEYALNK DEQYTISFWL RMPVPSSSEE RRIFSYSAVS GVNKEVEELI LQVKNNEFVL AT ANLLRNS EFVIEPRIAL NRWVKITIVN ENTRIKVYQN DNLLGLIKDS SRKKPIAQRG TFKFYNYNVD YQLDDISYYN GTI SQRDIK YTFKEDHGQF VYDHWGERLQ YNKAYYLLSD DNKSAFETVY ETKRLKLKSV PGVDIKYLGM NDRVYGYYGG LQFK LVPLD SKNMNNYVRW GDKFTMQSIE TTNLSLAIIQ DNAYFAPTQL KLISNEGKSE EEIFTFDRNI KLQNAAILVG TGNSK QGPI SAYKRGYSGD LWINGARLDG YVTVVNKSNY SNDEIQEKFK WIFVPKDANW VEGSYPYDVP DYALESGKET AAAKFE RQH MDSSTSAA UniProtKB: Putative botulinum-like toxin Wo |
-分子 #2: Structural protein
| 分子 | 名称: Structural protein / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Weissella oryzae (バクテリア) Weissella oryzae (バクテリア) |
| 分子量 | 理論値: 166.969734 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MGSDYKDDDD KSGMDNKLKT ENIRYFRTAA GSEDVLEVKA YEVYPNVWAI PSRYMMEPLQ DLDEVTNPEQ FSIYDKKYLA DIQEQDEFL KSIQAAIEDI KKRTFGLELL TAVSGAVPLP KDTGATNTTL QCIDENGKHT HDVVANVVLW GPGNNLNSNR L ISKSDDDS ...文字列: MGSDYKDDDD KSGMDNKLKT ENIRYFRTAA GSEDVLEVKA YEVYPNVWAI PSRYMMEPLQ DLDEVTNPEQ FSIYDKKYLA DIQEQDEFL KSIQAAIEDI KKRTFGLELL TAVSGAVPLP KDTGATNTTL QCIDENGKHT HDVVANVVLW GPGNNLNSNR L ISKSDDDS NGIGSMVELI WNPQILIKNI GTNRIKPATD ELVGLLTKAL FRLYGLGLNK IRYPFYQLDD KKYYSLTAED LI SYGGFSA NVVNLQPYYF LEDQFTKVKE KYESAKKRID DIKVNDEYSQ MLTLKYQFDL YSLFHISTSY IVSTVIPAND KYG GLVSYY TGPNALIDSK TDEKLTSMVK IPLKKIKYSK NQSREYDEYD LTNGEDSTQY FENFTFPKSK HVFVETQPTP ENVF VNLPS EEITKIILPV IPAESDLIKI PFQPATPKSI TTELITTDVP TLGLIFPAVK SKQNLSDIKM TSKLSDALDS DKQTF AFDN TLVDKLSELT SVSDAELFGI IRLIKNELLS VIDNFTTFGD NWSCPRWIDY CFQQVFGSDL KNLIVQGDFE KVFNIS DTL ILPKQLPEDI LQLKPYLFYQ WYAKRYTRIL RLESLFYQIL NEHITLIRSL VSSNNKGQYL QGFMNDLDKI AYNAQYM LS DWTIQLGYYD FKNQVTQVIK TSSMTSEFNI DDLLYDYDTF KLTISQFGAD SINNFTPSQD LKLALNDNNS PILLLGND E IKSNGSITQT DDSLDDETSL LLSKNTSFEG NFSAKYLLSS VGVNFTFKSI ENLNFSVDFM NINIAFSNNF FEITQTGQE TKKYSIAKLF GWNSLVYLIK HSSVEIWDIH SNILLVSHDL TAPQNNIVKA PIKLTNLDNE LILKSFEVFE QDEEANYNDI EQGFKNGII YTAKKMPIIV GEKYALKSSI LDDMGILTSD ENKKYPVFST DVEVESSLNI ILESTTGDKI SVDAGVNIRT I NSNGEENY LGIEDNHLIF VPKEEAELFY LKKAVVEDTI DIFYVVKTLG NMFINVERIS DNIYRLNFKA GILYSTMESD ML VLPAEEA NTAFYIQPIG LASLEVKDSV LGEGNPWLKE DNFLDATDDY GNQIDLSDNR ISVTGSVDTD KVGTYSVVYS YTG IDKTNT EKATITVKLD KSSIKTQDST LQNGKEWVRA DNLVEVIDED GNKVDYSDDR IIQEGDVDIN KAGVYDITFR YRGK FKIIS SSFKVTVIND IWYDSIKNAC KTYLIDYGER INDVKGITFQ NILEATRGKL YGYRVVYDNP HDVINQNPPK DFHFD LIKP FDVKNPSRVH LADYSGYLRL FIISTGKINT DIKVKIYAVL ENKDEIEIFD NHQNDKRHEE IAEIYKSNFD DNNYSA DGK YFISVLFKND VQAVVKDEIY GYEIFYSYFT KFRKDTAFQT DGSKRIFFHD YFNFEVPLDY KDSTFINVIL KNGEKIR IY KFAGYYYYSG HHHHHH UniProtKB: Ig-like domain-containing protein |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製 #1
試料調製 #1
| Preparation ID | 1 | ||||||
|---|---|---|---|---|---|---|---|
| 濃度 | 0.7 mg/mL | ||||||
| 緩衝液 | pH: 5.5 / 構成要素:
| ||||||
| グリッド | モデル: Quantifoil R2/2 / 材質: COPPER / メッシュ: 300 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 60 sec. | ||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
- 試料調製 #2
試料調製 #2
| Preparation ID | 2 | ||||||
|---|---|---|---|---|---|---|---|
| 濃度 | 0.2 mg/mL | ||||||
| 緩衝液 | pH: 5.5 / 構成要素:
| ||||||
| グリッド | モデル: Quantifoil R2/2 / 材質: COPPER / メッシュ: 300 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 60 sec. | ||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / 撮影したグリッド数: 2 / 実像数: 5946 / 平均電子線量: 40.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: OTHER / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 3.2 µm / 最小 デフォーカス(公称値): 1.6 µm / 倍率(公称値): 165000 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: AB INITIO MODEL |
|---|---|
| 得られたモデル |  PDB-8c8g: |
 ムービー
ムービー コントローラー
コントローラー




 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)