+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Hantaan virus polymerase in replication pre-initiation state | |||||||||
 マップデータ マップデータ | Hantaan virus polymerase cryo-EM map in replication pre-initiation state Local resolution filtered map | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Polymerase / replication / bunyavirus / VIRAL PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報RNA-templated viral transcription / negative stranded viral RNA replication / cap snatching / endonuclease activity / 加水分解酵素; エステル加水分解酵素 / host cell perinuclear region of cytoplasm / RNA-directed RNA polymerase / nucleotide binding / RNA-directed RNA polymerase activity / DNA-templated transcription / metal ion binding 類似検索 - 分子機能 | |||||||||
| 生物種 |  Hantaan virus 76-118 (ウイルス) Hantaan virus 76-118 (ウイルス) | |||||||||
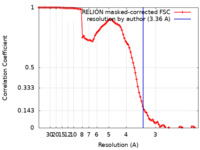
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.36 Å | |||||||||
 データ登録者 データ登録者 | Durieux trouilleton Q / Arragain B / Malet H | |||||||||
| 資金援助 |  フランス, 1件 フランス, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2023 ジャーナル: Nat Commun / 年: 2023タイトル: Structures of active Hantaan virus polymerase uncover the mechanisms of Hantaviridae genome replication. 著者: Quentin Durieux Trouilleton / Sergio Barata-García / Benoît Arragain / Juan Reguera / Hélène Malet /  要旨: Hantaviruses are causing life-threatening zoonotic infections in humans. Their tripartite negative-stranded RNA genome is replicated by the multi-functional viral RNA-dependent RNA-polymerase. Here ...Hantaviruses are causing life-threatening zoonotic infections in humans. Their tripartite negative-stranded RNA genome is replicated by the multi-functional viral RNA-dependent RNA-polymerase. Here we describe the structure of the Hantaan virus polymerase core and establish conditions for in vitro replication activity. The apo structure adopts an inactive conformation that involves substantial folding rearrangement of polymerase motifs. Binding of the 5' viral RNA promoter triggers Hantaan virus polymerase reorganization and activation. It induces the recruitment of the 3' viral RNA towards the polymerase active site for prime-and-realign initiation. The elongation structure reveals the formation of a template/product duplex in the active site cavity concomitant with polymerase core widening and the opening of a 3' viral RNA secondary binding site. Altogether, these elements reveal the molecular specificities of Hantaviridae polymerase structure and uncover the mechanisms underlying replication. They provide a solid framework for future development of antivirals against this group of emerging pathogens. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_16429.map.gz emd_16429.map.gz | 42.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-16429-v30.xml emd-16429-v30.xml emd-16429.xml emd-16429.xml | 25.7 KB 25.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_16429_fsc.xml emd_16429_fsc.xml | 9.2 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_16429.png emd_16429.png | 78.9 KB | ||
| Filedesc metadata |  emd-16429.cif.gz emd-16429.cif.gz | 8.1 KB | ||
| その他 |  emd_16429_additional_1.map.gz emd_16429_additional_1.map.gz emd_16429_half_map_1.map.gz emd_16429_half_map_1.map.gz emd_16429_half_map_2.map.gz emd_16429_half_map_2.map.gz | 4.4 MB 52 MB 52 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-16429 http://ftp.pdbj.org/pub/emdb/structures/EMD-16429 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-16429 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-16429 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_16429_validation.pdf.gz emd_16429_validation.pdf.gz | 790.6 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_16429_full_validation.pdf.gz emd_16429_full_validation.pdf.gz | 790.2 KB | 表示 | |
| XML形式データ |  emd_16429_validation.xml.gz emd_16429_validation.xml.gz | 16.2 KB | 表示 | |
| CIF形式データ |  emd_16429_validation.cif.gz emd_16429_validation.cif.gz | 21 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16429 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16429 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16429 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16429 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_16429.map.gz / 形式: CCP4 / 大きさ: 67 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_16429.map.gz / 形式: CCP4 / 大きさ: 67 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Hantaan virus polymerase cryo-EM map in replication pre-initiation state Local resolution filtered map | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.1 Å | ||||||||||||||||||||||||||||||||||||
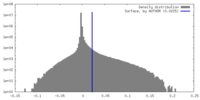
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: Hantaan virus polymerase cryo-EM map in replication pre-initiation...
| ファイル | emd_16429_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Hantaan virus polymerase cryo-EM map in replication pre-initiation state Post-processed map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
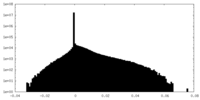
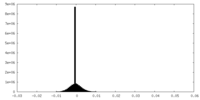
| 密度ヒストグラム |
-ハーフマップ: Hantaan virus polymerase cryo-EM map in replication pre-initiation...
| ファイル | emd_16429_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Hantaan virus polymerase cryo-EM map in replication pre-initiation state Half-map 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
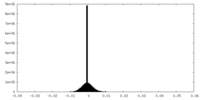
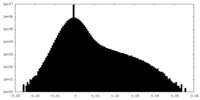
| 密度ヒストグラム |
-ハーフマップ: Hantaan virus polymerase cryo-EM map in replication pre-initiation...
| ファイル | emd_16429_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Hantaan virus polymerase cryo-EM map in replication pre-initiation state Half map 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
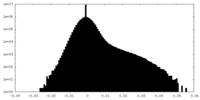
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Hantaan virus polymerase in replication pre-initiation state
| 全体 | 名称: Hantaan virus polymerase in replication pre-initiation state |
|---|---|
| 要素 |
|
-超分子 #1: Hantaan virus polymerase in replication pre-initiation state
| 超分子 | 名称: Hantaan virus polymerase in replication pre-initiation state タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#3 |
|---|---|
| 由来(天然) | 生物種:  Hantaan virus 76-118 (ウイルス) / 株: 76-118 Hantaan virus 76-118 (ウイルス) / 株: 76-118 |
| 分子量 | 理論値: 246 KDa |
-分子 #1: RNA-directed RNA polymerase L
| 分子 | 名称: RNA-directed RNA polymerase L / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO / EC番号: RNA-directed RNA polymerase |
|---|---|
| 由来(天然) | 生物種:  Hantaan virus 76-118 (ウイルス) / 株: 76-118 Hantaan virus 76-118 (ウイルス) / 株: 76-118 |
| 分子量 | 理論値: 249.432484 KDa |
| 組換発現 | 生物種:  Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 配列 | 文字列: MGHHHHHHDY DIPTTENLYF QGMDKYREIH NKLKEFSPGT LTAVECIDYL DRLYAVRHDI VDQMIKHDWS DNKDSEEAIG KVLLFAGVP SNIITALEKK IIPNHPTGKS LKAFFKMTPA NYKISGTTIE FVEVTVTADV DKGIREKKLK YEAGLTYIEQ E LHKFFLKG ...文字列: MGHHHHHHDY DIPTTENLYF QGMDKYREIH NKLKEFSPGT LTAVECIDYL DRLYAVRHDI VDQMIKHDWS DNKDSEEAIG KVLLFAGVP SNIITALEKK IIPNHPTGKS LKAFFKMTPA NYKISGTTIE FVEVTVTADV DKGIREKKLK YEAGLTYIEQ E LHKFFLKG EIPQPYKITF NVVAVRTDGS NITTQWPSRR NDGVVQYMRL VQAEISYVRE HLIKTEERAA LEAMFNLKFN IS THKSQPY YIPDYKGMEP IGANIEDLVD YSKDWLSRAR NFSFFEVKGT AVFECFNSNE ANHCQRYPMS RKPRNFLLIQ CSL ITSYKP ATTLSDQIDS RRACSYILNL IPDTPASYLI HDMAYRYINL TREDMINYYA PRIQFKQTQN VREPGTFKLT SSML RAESK AMLDLLNNHK SGEKHGAQIE SLNIASHIVQ SESVSLITKI LSDLELNITE PSTQEYSTTK HTYVDTVLDK FFQNE TQKY LIDVLKKTTA WHIGHLIRDI TESLIAHSGL KRSKYWSLHS YNNGNVILFI LPSKSLEVAG SFIRFITVFR IGPGLV DKD NLDTILIDGD SQWGVSKVMS IDLNRLLALN IAFEKALIAT ATWFQYYTED QGQFPLQYAI RSVFANHFLL AICQKMK LC AIFDNLRYLI PAVTSLYSGF PSLIEKLFER PFKSSLEVYI YYNIKSLLVA LAQNNKARFY SKVKLLGLTV DQSTVGAS G VYPSFMSRIV YKHYRSLISE VTTCFFLFEK GLHGNMNEEA KIHLETVEWA LKFREKEEKY GESLVENGYM MWELRANAE LAEQQLYCQD AIELAAIELN KVLATKSSVV ANSILSKNWE EPYFSQTRNI SLKGMSGQVQ EDGHLSSSVT IIEAIRYLSN SRHNPSLLK LYEETREQKA MARIVRKYQR TEADRGFFIT TLPTRCRLEI IEDYYDAIAK NISEEYISYG GEKKILAIQG A LEKALRWA SGESFIELSN HKFIRMKRKL MYVSADATKW SPGDNSAKFR RFTSMLHNGL PNNKLKNCVI DALKQVYKTD FF MSRKLRN YIDSMESLDP HIKQFLDFFP DGHHGEVKGN WLQGNLNKCS SLFGVAMSLL FKQVWTNLFP ELDCFFEFAH HSD DALFIY GYLEPVDDGT DWFLFVSQQI QAGHLHWFSV NTEMWKSMFN LHEHILLLGS IKISPKKTTV SPTNAEFLST FFEG CAVSI PFVKILLGSL SDLPGLGYFD DLAAAQSRCV KALDLGASPQ VAQLAVALCT SKVERLYGTA PGMVNHPAAY LQVKH TDTP IPLGGNGAMS IMELATAGIG MSDKNLLKRA LLGYSHKRQK SMLYILGLFK FLMKLSDETF QHERLGQFSF IGKVQW KIF TPKSEFEFAD MYTSKFLELW SSQHVTYDYI IPKGRDNLLI YLVRKLNDPS IVTAMTMQSP LQLRFRMQAK QHMKVCR LD GEWVTFREVL AAANSFAENY SATSQDMDLF QTLTSCTFSK EYAWKDFLNG IHCDVIPTKQ VQRAKVARTF TVREKDQI I QNSIPAVIGY KFAVTVEEMS DVLDTAKFPD SLSVDLKTMK DGVYRELGLD ISLPDVMKRI APMLYKSSKS RVVIVQGNV EGTAEAICRY WLKSMSLVKT IRVKPHKEVL QAVSIFNRKE DIGQQKDLAA LKLCIEVWRW CKANSAPYRD WFQALWFEDK TFSEWLDRF CRVGVPPIDP EIQCAALMIA DIKGDYSVLQ LQANRRAYSG KQYDAYCVQT YNEVTKLYEG DLRVTFNFGL D CARLEIFW DKKAYILETS ITQKHVLKIM MDEVSKELIK CGMRFNTEQV QGVRHMVLFK TESGFEWGKP NIPCIVYKNC VL RTSLRTT QAINHKFMIT IKDDGLRAIA QHDEDSPRFL LAHAFHTIRD IRYQAVDAVS NVWFIHKGVK LYLNPIISSG LLE NFMKNL PAAIPPAAYS LIMNRAKISV DLFMFNDLLK LINPRNTLDL SGLETTGDEF STVSSMSSRL WSEEMSLVDD DEEL DDEFT IDLQDVDFEN IDIEADIEHF LQDESSYTGD LLISTEETES KKMRGIVKIL EPVRLIKSWV SRGLSIEKVY SPVNI ILMS RYISKTFNLS TKQVSLLDPY DLTELESIVR GWGECVIDQF ESLDREAQNM VVNKGICPED VIPDSLFSFR HTMVLL RRL FPQDSISSFY UniProtKB: RNA-directed RNA polymerase L |
-分子 #2: RNA (5'-R(P*UP*AP*GP*GP*AP*GP*UP*AP*UP*CP*CP*AP*CP*CP*GP*CP*AP*AP...
| 分子 | 名称: RNA (5'-R(P*UP*AP*GP*GP*AP*GP*UP*AP*UP*CP*CP*AP*CP*CP*GP*CP*AP*AP*GP*A)-3') タイプ: rna / ID: 2 / コピー数: 1 |
|---|---|
| 由来(天然) | 生物種:  Hantaan virus 76-118 (ウイルス) Hantaan virus 76-118 (ウイルス) |
| 分子量 | 理論値: 6.429919 KDa |
| 配列 | 文字列: UAGGAGUAUC CACCGCAAGA |
-分子 #3: RNA (5'-R(P*UP*UP*UP*UP*GP*CP*GP*GP*AP*GP*UP*AP*CP*UP*AP*CP*UP*A)-3')
| 分子 | 名称: RNA (5'-R(P*UP*UP*UP*UP*GP*CP*GP*GP*AP*GP*UP*AP*CP*UP*AP*CP*UP*A)-3') タイプ: rna / ID: 3 / コピー数: 1 |
|---|---|
| 由来(天然) | 生物種:  Hantaan virus 76-118 (ウイルス) Hantaan virus 76-118 (ウイルス) |
| 分子量 | 理論値: 7.851604 KDa |
| 配列 | 文字列: CUUUCUUUUG CGGAGUCUAC UACUA |
-分子 #4: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 4 / コピー数: 1 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.3 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 8 構成要素:
| ||||||||||||
| グリッド | モデル: UltrAuFoil R1.2/1.3 / 材質: GOLD / メッシュ: 300 / 支持フィルム - 材質: GOLD / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 30 sec. / 詳細: 25 mA | ||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 276 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS GLACIOS |
|---|---|
| 温度 | 最低: 77.0 K / 最高: 77.0 K |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / デジタル化 - サイズ - 横: 3840 pixel / デジタル化 - サイズ - 縦: 3712 pixel / デジタル化 - 画像ごとのフレーム数: 1-50 / 撮影したグリッド数: 1 / 実像数: 3260 / 平均露光時間: 5.5 sec. / 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 最大 デフォーカス(補正後): 2.2 µm / 最小 デフォーカス(補正後): 2.2 µm / 倍率(補正後): 45454 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 0.8 µm / 最小 デフォーカス(公称値): 0.8 µm / 倍率(公称値): 36000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: AB INITIO MODEL / 温度因子: 68.69 |
|---|---|
| 得られたモデル |  PDB-8c4u: |
 ムービー
ムービー コントローラー
コントローラー










 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)