+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | SARS-CoV2 Omicron BA.1 RBD in complex with CAB-A17 antibody | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Antibody / Spike / Complex / VIRAL PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報Maturation of spike protein / viral translation / Translation of Structural Proteins / Virion Assembly and Release / host cell surface / host extracellular space / symbiont-mediated-mediated suppression of host tetherin activity / Induction of Cell-Cell Fusion / structural constituent of virion / entry receptor-mediated virion attachment to host cell ...Maturation of spike protein / viral translation / Translation of Structural Proteins / Virion Assembly and Release / host cell surface / host extracellular space / symbiont-mediated-mediated suppression of host tetherin activity / Induction of Cell-Cell Fusion / structural constituent of virion / entry receptor-mediated virion attachment to host cell / membrane fusion / Attachment and Entry / host cell endoplasmic reticulum-Golgi intermediate compartment membrane / positive regulation of viral entry into host cell / receptor-mediated virion attachment to host cell / host cell surface receptor binding / symbiont-mediated suppression of host innate immune response / receptor ligand activity / endocytosis involved in viral entry into host cell / fusion of virus membrane with host plasma membrane / fusion of virus membrane with host endosome membrane / viral envelope / virion attachment to host cell / SARS-CoV-2 activates/modulates innate and adaptive immune responses / host cell plasma membrane / virion membrane / identical protein binding / membrane / plasma membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |   Enterobacteria phage T4 (ファージ) / Enterobacteria phage T4 (ファージ) /  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
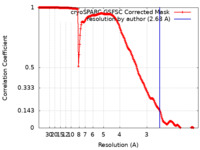
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.68 Å | |||||||||
 データ登録者 データ登録者 | Das H / Hallberg BM | |||||||||
| 資金援助 | 1件
| |||||||||
 引用 引用 |  ジャーナル: Cell Rep Med / 年: 2024 ジャーナル: Cell Rep Med / 年: 2024タイトル: Structural basis of broad SARS-CoV-2 cross-neutralization by affinity-matured public antibodies. 著者: Daniel J Sheward / Pradeepa Pushparaj / Hrishikesh Das / Allison J Greaney / Changil Kim / Sungyong Kim / Leo Hanke / Erik Hyllner / Robert Dyrdak / Jimin Lee / Xaquin Castro Dopico / Pia ...著者: Daniel J Sheward / Pradeepa Pushparaj / Hrishikesh Das / Allison J Greaney / Changil Kim / Sungyong Kim / Leo Hanke / Erik Hyllner / Robert Dyrdak / Jimin Lee / Xaquin Castro Dopico / Pia Dosenovic / Thomas P Peacock / Gerald M McInerney / Jan Albert / Martin Corcoran / Jesse D Bloom / Ben Murrell / Gunilla B Karlsson Hedestam / B Martin Hällberg /      要旨: Descendants of the severe acute respiratory syndrome coronavirus 2 (SARS-CoV-2) Omicron variant now account for almost all SARS-CoV-2 infections. The Omicron variant and its sublineages have spike ...Descendants of the severe acute respiratory syndrome coronavirus 2 (SARS-CoV-2) Omicron variant now account for almost all SARS-CoV-2 infections. The Omicron variant and its sublineages have spike glycoproteins that are highly diverged from the pandemic founder and first-generation vaccine strain, resulting in significant evasion from monoclonal antibody therapeutics and vaccines. Understanding how commonly elicited antibodies can broaden to cross-neutralize escape variants is crucial. We isolate IGHV3-53, using "public" monoclonal antibodies (mAbs) from an individual 7 months post infection with the ancestral virus and identify antibodies that exhibit potent and broad cross-neutralization, extending to the BA.1, BA.2, and BA.4/BA.5 sublineages of Omicron. Deep mutational scanning reveals these mAbs' high resistance to viral escape. Structural analysis via cryoelectron microscopy of a representative broadly neutralizing antibody, CAB-A17, in complex with the Omicron BA.1 spike highlights the structural underpinnings of this broad neutralization. By reintroducing somatic hypermutations into a germline-reverted CAB-A17, we delineate the role of affinity maturation in the development of cross-neutralization by a public class of antibodies. #1:  ジャーナル: Biochem Biophys Res Commun / 年: 1975 ジャーナル: Biochem Biophys Res Commun / 年: 1975タイトル: Delineation of the intimate details of the backbone conformation of pyridine nucleotide coenzymes in aqueous solution. 著者: Bose KS / Sarma RH | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_16375.map.gz emd_16375.map.gz | 412.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-16375-v30.xml emd-16375-v30.xml emd-16375.xml emd-16375.xml | 20.9 KB 20.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_16375_fsc.xml emd_16375_fsc.xml | 19.8 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_16375.png emd_16375.png | 15.6 KB | ||
| Filedesc metadata |  emd-16375.cif.gz emd-16375.cif.gz | 6.3 KB | ||
| その他 |  emd_16375_half_map_1.map.gz emd_16375_half_map_1.map.gz emd_16375_half_map_2.map.gz emd_16375_half_map_2.map.gz | 764.7 MB 764.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-16375 http://ftp.pdbj.org/pub/emdb/structures/EMD-16375 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-16375 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-16375 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_16375_validation.pdf.gz emd_16375_validation.pdf.gz | 855.1 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_16375_full_validation.pdf.gz emd_16375_full_validation.pdf.gz | 854.6 KB | 表示 | |
| XML形式データ |  emd_16375_validation.xml.gz emd_16375_validation.xml.gz | 29.1 KB | 表示 | |
| CIF形式データ |  emd_16375_validation.cif.gz emd_16375_validation.cif.gz | 38.6 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16375 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16375 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16375 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16375 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_16375.map.gz / 形式: CCP4 / 大きさ: 824 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_16375.map.gz / 形式: CCP4 / 大きさ: 824 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.01 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #1
| ファイル | emd_16375_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
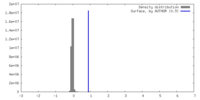
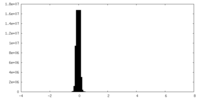
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_16375_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
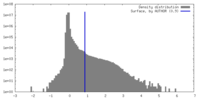
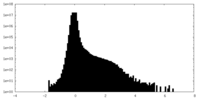
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : SARS-CoV2 Omicron BA.1 Spike in complex with CAB-A17 antibody
| 全体 | 名称: SARS-CoV2 Omicron BA.1 Spike in complex with CAB-A17 antibody |
|---|---|
| 要素 |
|
-超分子 #1: SARS-CoV2 Omicron BA.1 Spike in complex with CAB-A17 antibody
| 超分子 | 名称: SARS-CoV2 Omicron BA.1 Spike in complex with CAB-A17 antibody タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 分子量 | 理論値: 95.7 KDa |
-超分子 #2: SARS-CoV-2 Omicron RBD
| 超分子 | 名称: SARS-CoV-2 Omicron RBD / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  |
-超分子 #3: CAB-A17 Antibody (Fab)
| 超分子 | 名称: CAB-A17 Antibody (Fab) / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #2-#3 |
|---|---|
| 由来(天然) | 生物種:  Enterobacteria phage T4 (ファージ) Enterobacteria phage T4 (ファージ) |
-分子 #1: Spike protein S2'
| 分子 | 名称: Spike protein S2' / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 21.942738 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: NLCPFDEVFN ATRFASVYAW NRKRISNCVA DYSVLYNSAP FFTFKCYGVS PTKLNDLCFT NVYADSFVIR GDEVRQIAPG QTGNIADYN YKLPDDFTGC VIAWNSNKLD SKVSGNYNYL YRLFRKSNLK PFERDISTEI YQAGNKPCNG VAGFNCYFPL R SYSFRPTY ...文字列: NLCPFDEVFN ATRFASVYAW NRKRISNCVA DYSVLYNSAP FFTFKCYGVS PTKLNDLCFT NVYADSFVIR GDEVRQIAPG QTGNIADYN YKLPDDFTGC VIAWNSNKLD SKVSGNYNYL YRLFRKSNLK PFERDISTEI YQAGNKPCNG VAGFNCYFPL R SYSFRPTY GVGHQPYRVV VLSFELLHAP ATVCG UniProtKB: Spike glycoprotein |
-分子 #2: CAB-A17 antibody (variable)
| 分子 | 名称: CAB-A17 antibody (variable) / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 11.395632 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: EIVLTQSPGT LSLSPGERAS LSCRASQSLS TYLAWYQQKP GQAPRLLIFG ASSRASGIPD RFSGGGSGTD FTLTISRLEP EDFAVYYCQ QYGSSPRTFG QGTKVEI |
-分子 #3: CAB-A17 antibody (variable)
| 分子 | 名称: CAB-A17 antibody (variable) / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 12.839197 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: DVHLVESGGG LIQPGGSLRL SCAASEFIVS ANYMSWVRQA PGEGLQWVSV IYPGGSTFYA ESVKGRFTIS RDNSRNTLYL QMNSLRAED TGVYYCARDY GDFYFDYWGQ GTLVTVS |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1.5 mg/mL |
|---|---|
| 緩衝液 | pH: 7.4 |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 10 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 実像数: 25652 / 平均露光時間: 1.5 sec. / 平均電子線量: 54.6 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 20.0 µm / 最小 デフォーカス(補正後): 0.3 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 0.3 µm / 倍率(公称値): 165000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー













 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)