+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Enp1TAP_B, multibody refinement | |||||||||
 マップデータ マップデータ | Enp1TAP_B, multibody refinement | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |  | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.4 Å | |||||||||
 データ登録者 データ登録者 | Milkereit P / Poell G | |||||||||
| 資金援助 |  ドイツ, 1件 ドイツ, 1件
| |||||||||
 引用 引用 |  ジャーナル: PLoS One / 年: 2023 ジャーナル: PLoS One / 年: 2023タイトル: Impact of the yeast S0/uS2-cluster ribosomal protein rpS21/eS21 on rRNA folding and the architecture of small ribosomal subunit precursors. 著者: Gisela Pöll / Joachim Griesenbeck / Herbert Tschochner / Philipp Milkereit /  要旨: RpS0/uS2, rpS2/uS5, and rpS21/eS21 form a cluster of ribosomal proteins (S0-cluster) at the head-body junction near the central pseudoknot of eukaryotic small ribosomal subunits (SSU). Previous work ...RpS0/uS2, rpS2/uS5, and rpS21/eS21 form a cluster of ribosomal proteins (S0-cluster) at the head-body junction near the central pseudoknot of eukaryotic small ribosomal subunits (SSU). Previous work in yeast indicated that S0-cluster assembly is required for the stabilisation and maturation of SSU precursors at specific post-nucleolar stages. Here, we analysed the role of S0-cluster formation for rRNA folding. Structures of SSU precursors isolated from yeast S0-cluster expression mutants or control strains were analysed by cryogenic electron microscopy. The obtained resolution was sufficient to detect individual 2'-O-methyl RNA modifications using an unbiased scoring approach. The data show how S0-cluster formation enables the initial recruitment of the pre-rRNA processing factor Nob1 in yeast. Furthermore, they reveal hierarchical effects on the pre-rRNA folding pathway, including the final maturation of the central pseudoknot. Based on these structural insights we discuss how formation of the S0-cluster determines at this early cytoplasmic assembly checkpoint if SSU precursors further mature or are degraded. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_16350.map.gz emd_16350.map.gz | 14.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-16350-v30.xml emd-16350-v30.xml emd-16350.xml emd-16350.xml | 15.9 KB 15.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_16350.png emd_16350.png | 162 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-16350 http://ftp.pdbj.org/pub/emdb/structures/EMD-16350 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-16350 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-16350 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_16350_validation.pdf.gz emd_16350_validation.pdf.gz | 406.5 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_16350_full_validation.pdf.gz emd_16350_full_validation.pdf.gz | 406.1 KB | 表示 | |
| XML形式データ |  emd_16350_validation.xml.gz emd_16350_validation.xml.gz | 7.1 KB | 表示 | |
| CIF形式データ |  emd_16350_validation.cif.gz emd_16350_validation.cif.gz | 8.1 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16350 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16350 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16350 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16350 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_16350.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_16350.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Enp1TAP_B, multibody refinement | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.968 Å | ||||||||||||||||||||||||||||||||||||
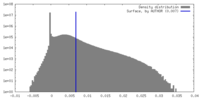
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Enp1-TAP associated immature ribosomal particles from S. cerevisiae
| 全体 | 名称: Enp1-TAP associated immature ribosomal particles from S. cerevisiae |
|---|---|
| 要素 |
|
-超分子 #1: Enp1-TAP associated immature ribosomal particles from S. cerevisiae
| 超分子 | 名称: Enp1-TAP associated immature ribosomal particles from S. cerevisiae タイプ: complex / ID: 1 / キメラ: Yes / 親要素: 0 / 含まれる分子: #1-#31 |
|---|---|
| 由来(天然) | 生物種:  |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 構成要素:
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY | ||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | JEOL CRYO ARM 200 |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / 撮影したグリッド数: 1 / 実像数: 8575 / 平均露光時間: 4.4 sec. / 平均電子線量: 40.0 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.2 µm / 最小 デフォーカス(公称値): 0.8 µm / 倍率(公称値): 50000 |
| 試料ステージ | 試料ホルダーモデル: JEOL CRYOSPECPORTER / ホルダー冷却材: NITROGEN |
- 画像解析
画像解析
| 最終 再構成 | 使用したクラス数: 1 / 想定した対称性 - 点群: C1 (非対称) / 解像度のタイプ: BY AUTHOR / 解像度: 3.4 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: RELION (ver. 4.0) / 使用した粒子像数: 24592 |
|---|---|
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD / ソフトウェア - 名称: RELION (ver. 4.0) |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD / ソフトウェア - 名称: RELION (ver. 4.0) |
| 最終 3次元分類 | ソフトウェア - 名称: RELION (ver. 4.0) |
-原子モデル構築 1
| 精密化 | プロトコル: FLEXIBLE FIT |
|---|
 ムービー
ムービー コントローラー
コントローラー


















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)