+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | In situ subtomogram average of GEM2 particles in human cells | |||||||||||||||||||||
 マップデータ マップデータ | FSC-weighted non-sharpened map | |||||||||||||||||||||
 試料 試料 |
| |||||||||||||||||||||
 キーワード キーワード | Encapsulin / Protein Engineering / CYTOSOLIC PROTEIN | |||||||||||||||||||||
| 機能・相同性 | : / Type 2A encapsulin shell protein SrpI-like / Type 2A encapsulin shell protein SrpI-like / encapsulin nanocompartment / Type 2A encapsulin shell protein SrpI 機能・相同性情報 機能・相同性情報 | |||||||||||||||||||||
| 生物種 |  Synechococcus elongatus PCC 7942 = FACHB-805 (バクテリア) / Synechococcus elongatus PCC 7942 = FACHB-805 (バクテリア) /  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||||||||||||||
| 手法 | サブトモグラム平均法 / クライオ電子顕微鏡法 / 解像度: 31.3 Å | |||||||||||||||||||||
 データ登録者 データ登録者 | Fung HKH / Hayashi Y / Salo VT / Babenko A / Zagoriy I / Brunner A / Ellenberg J / Mueller CW / Cuylen-Haering S / Mahamid J | |||||||||||||||||||||
| 資金援助 |  ドイツ, European Union, ドイツ, European Union,  フランス, 6件 フランス, 6件
| |||||||||||||||||||||
 引用 引用 |  ジャーナル: Nat Methods / 年: 2023 ジャーナル: Nat Methods / 年: 2023タイトル: Genetically encoded multimeric tags for subcellular protein localization in cryo-EM. 著者: Herman K H Fung / Yuki Hayashi / Veijo T Salo / Anastasiia Babenko / Ievgeniia Zagoriy / Andreas Brunner / Jan Ellenberg / Christoph W Müller / Sara Cuylen-Haering / Julia Mahamid /  要旨: Cryo-electron tomography (cryo-ET) allows for label-free high-resolution imaging of macromolecular assemblies in their native cellular context. However, the localization of macromolecules of interest ...Cryo-electron tomography (cryo-ET) allows for label-free high-resolution imaging of macromolecular assemblies in their native cellular context. However, the localization of macromolecules of interest in tomographic volumes can be challenging. Here we present a ligand-inducible labeling strategy for intracellular proteins based on fluorescent, 25-nm-sized, genetically encoded multimeric particles (GEMs). The particles exhibit recognizable structural signatures, enabling their automated detection in cryo-ET data by convolutional neural networks. The coupling of GEMs to green fluorescent protein-tagged macromolecules of interest is triggered by addition of a small-molecule ligand, allowing for time-controlled labeling to minimize disturbance to native protein function. We demonstrate the applicability of GEMs for subcellular-level localization of endogenous and overexpressed proteins across different organelles in human cells using cryo-correlative fluorescence and cryo-ET imaging. We describe means for quantifying labeling specificity and efficiency, and for systematic optimization for rare and abundant protein targets, with emphasis on assessing the potential effects of labeling on protein function. | |||||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_16303.map.gz emd_16303.map.gz | 7.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-16303-v30.xml emd-16303-v30.xml emd-16303.xml emd-16303.xml | 16.7 KB 16.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_16303_fsc.xml emd_16303_fsc.xml | 4.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_16303.png emd_16303.png | 51 KB | ||
| Filedesc metadata |  emd-16303.cif.gz emd-16303.cif.gz | 5.6 KB | ||
| その他 |  emd_16303_half_map_1.map.gz emd_16303_half_map_1.map.gz emd_16303_half_map_2.map.gz emd_16303_half_map_2.map.gz | 6 MB 6 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-16303 http://ftp.pdbj.org/pub/emdb/structures/EMD-16303 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-16303 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-16303 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_16303_validation.pdf.gz emd_16303_validation.pdf.gz | 485.2 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_16303_full_validation.pdf.gz emd_16303_full_validation.pdf.gz | 484.7 KB | 表示 | |
| XML形式データ |  emd_16303_validation.xml.gz emd_16303_validation.xml.gz | 10.3 KB | 表示 | |
| CIF形式データ |  emd_16303_validation.cif.gz emd_16303_validation.cif.gz | 13.8 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16303 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16303 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16303 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16303 | HTTPS FTP |
-関連構造データ
| 関連構造データ | C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_16303.map.gz / 形式: CCP4 / 大きさ: 8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_16303.map.gz / 形式: CCP4 / 大きさ: 8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | FSC-weighted non-sharpened map | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 6.85 Å | ||||||||||||||||||||||||||||||||||||
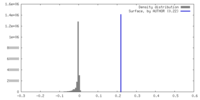
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: Half map
| ファイル | emd_16303_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
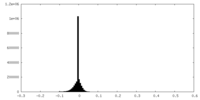
| 密度ヒストグラム |
-ハーフマップ: Half map
| ファイル | emd_16303_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
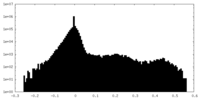
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Cryo-focused ion beam lamellae of human cells (HeLa, U2OS, Sum159...
| 全体 | 名称: Cryo-focused ion beam lamellae of human cells (HeLa, U2OS, Sum159) expressing the engineered genetically encoded multimeric tag GEM2 |
|---|---|
| 要素 |
|
-超分子 #1: Cryo-focused ion beam lamellae of human cells (HeLa, U2OS, Sum159...
| 超分子 | 名称: Cryo-focused ion beam lamellae of human cells (HeLa, U2OS, Sum159) expressing the engineered genetically encoded multimeric tag GEM2 タイプ: cell / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Synechococcus elongatus PCC 7942 = FACHB-805 (バクテリア) Synechococcus elongatus PCC 7942 = FACHB-805 (バクテリア) |
-分子 #1: Cryo-focused ion beam lamellae of human cells (HeLa, U2OS, Sum159...
| 分子 | 名称: Cryo-focused ion beam lamellae of human cells (HeLa, U2OS, Sum159) expressing the engineered genetically encoded multimeric tag GEM2 タイプ: protein_or_peptide / ID: 1 詳細: Fusion of Synechococcus elongatus Srp1 with Halo-tag and the FKBP-rapamycin-binding (FRB) domain of mTOR 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MGSTDNAPQL ALRDVAARQL ANATKTVPQL RTITPRWLVR LLHWTPVEAG IYRVNQVKDA SQITVACSER DESELPETFV DYIDNPREYL LSAVNTVVDV HTRISDLYSN PHDQIREQLR LTIEIMKERQ ESELINSREY GLLNNVAPGQ LVHTRNGAPT PDDLDELLIR ...文字列: MGSTDNAPQL ALRDVAARQL ANATKTVPQL RTITPRWLVR LLHWTPVEAG IYRVNQVKDA SQITVACSER DESELPETFV DYIDNPREYL LSAVNTVVDV HTRISDLYSN PHDQIREQLR LTIEIMKERQ ESELINSREY GLLNNVAPGQ LVHTRNGAPT PDDLDELLIR VWKEPAFFLA HPQAIAAFGR ECTRRGVPPA TVSLFGSSFI TWRGVPLIPS DKVPLENGKT KILLLRVGES RQGVVGLYQP NLPGEQGMGL SVRFMGINRK ALASYLVSLY CSLAVLTDDA LAVLDNVDVT QYHTYRYNGT GGGGSGGGSG GGSAEIGTGF PFDPHYVEVL GERMHYVDVG PRDGTPVLFL HGNPTSSYVW RNIIPHVAPT HRCIAPDLIG MGKSDKPDLG YFFDDHVRFM DAFIEALGLE EVVLVIHDWG SALGFHWAKR NPERVKGIAF MEFIRPIPTW DEWPEFARET FQAFRTTDVG RKLIIDQNVF IEGTLPMGVV RPLTEVEMDH YREPFLNPVD REPLWRFPNE LPIAGEPANI VALVEEYMDW LHQSPVPKLL FWGTPGVLIP PAEAARLAKS LPNCKAVDIG PGLNLLQEDN PDLIGSEIAR WLSTLEISGK LGSAGSAAGS GEGILWHEMW HEGLEEASRL YFGERNVKGM FEVLEPLHAM MERGPQTLKE TSFNQAYGRD LMEAQEWCRK YMKSGNVKDL LQAWDLYYHV FRRISK UniProtKB: Type 2A encapsulin shell protein SrpI |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | サブトモグラム平均法 |
| 試料の集合状態 | cell |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | #0 - Image recording ID: 1 #0 - フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) #0 - 平均電子線量: 2.0 e/Å2 / #1 - Image recording ID: 2 #1 - フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) #1 - 平均電子線量: 2.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 4.0 µm / 最小 デフォーカス(公称値): 1.5 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー





 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)