+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Release state - ES27up (pre-60S) | ||||||||||||
 マップデータ マップデータ | Composite map | ||||||||||||
 試料 試料 |
| ||||||||||||
| 生物種 |  | ||||||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.0 Å | ||||||||||||
 データ登録者 データ登録者 | Thoms M / Mitterer V / Buschauer R / Berninghausen O / Hurt E / Beckmann R | ||||||||||||
| 資金援助 |  ドイツ, European Union, 3件 ドイツ, European Union, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Elife / 年: 2023 ジャーナル: Elife / 年: 2023タイトル: Concurrent remodelling of nucleolar 60S subunit precursors by the Rea1 ATPase and Spb4 RNA helicase. 著者: Valentin Mitterer / Matthias Thoms / Robert Buschauer / Otto Berninghausen / Ed Hurt / Roland Beckmann /  要旨: Biogenesis intermediates of nucleolar ribosomal 60S precursor particles undergo a number of structural maturation steps before they transit to the nucleoplasm and are finally exported into the ...Biogenesis intermediates of nucleolar ribosomal 60S precursor particles undergo a number of structural maturation steps before they transit to the nucleoplasm and are finally exported into the cytoplasm. The AAA-ATPase Rea1 participates in the nucleolar exit by releasing the Ytm1-Erb1 heterodimer from the evolving pre-60S particle. Here, we show that the DEAD-box RNA helicase Spb4 with its interacting partner Rrp17 is further integrated into this maturation event. Spb4 binds to a specific class of late nucleolar pre-60S intermediates, whose cryo-EM structure revealed how its helicase activity facilitates melting and restructuring of 25S rRNA helices H62 and H63/H63a prior to Ytm1-Erb1 release. In vitro maturation of such Spb4-enriched pre-60S particles, incubated with purified Rea1 and its associated pentameric Rix1-complex in the presence of ATP, combined with cryo-EM analysis depicted the details of the Rea1-dependent large-scale pre-ribosomal remodeling. Our structural insights unveil how the Rea1 ATPase and Spb4 helicase remodel late nucleolar pre-60S particles by rRNA restructuring and dismantling of a network of several ribosomal assembly factors. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_16276.map.gz emd_16276.map.gz | 19.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-16276-v30.xml emd-16276-v30.xml emd-16276.xml emd-16276.xml | 34.7 KB 34.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_16276.png emd_16276.png | 87.4 KB | ||
| その他 |  emd_16276_additional_1.map.gz emd_16276_additional_1.map.gz emd_16276_additional_2.map.gz emd_16276_additional_2.map.gz emd_16276_additional_3.map.gz emd_16276_additional_3.map.gz emd_16276_additional_4.map.gz emd_16276_additional_4.map.gz emd_16276_additional_5.map.gz emd_16276_additional_5.map.gz emd_16276_additional_6.map.gz emd_16276_additional_6.map.gz emd_16276_additional_7.map.gz emd_16276_additional_7.map.gz emd_16276_half_map_1.map.gz emd_16276_half_map_1.map.gz emd_16276_half_map_2.map.gz emd_16276_half_map_2.map.gz | 6.6 MB 8.4 MB 7.1 MB 2.5 MB 21.7 MB 239.6 MB 450.2 MB 442 MB 442 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-16276 http://ftp.pdbj.org/pub/emdb/structures/EMD-16276 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-16276 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-16276 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_16276_validation.pdf.gz emd_16276_validation.pdf.gz | 809.5 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_16276_full_validation.pdf.gz emd_16276_full_validation.pdf.gz | 809 KB | 表示 | |
| XML形式データ |  emd_16276_validation.xml.gz emd_16276_validation.xml.gz | 18.6 KB | 表示 | |
| CIF形式データ |  emd_16276_validation.cif.gz emd_16276_validation.cif.gz | 22.1 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16276 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16276 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16276 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16276 | HTTPS FTP |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_16276.map.gz / 形式: CCP4 / 大きさ: 476.8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_16276.map.gz / 形式: CCP4 / 大きさ: 476.8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Composite map | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.059 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: Foot - Local Refinement - Local filtered map
| ファイル | emd_16276_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Foot - Local Refinement - Local filtered map | ||||||||||||
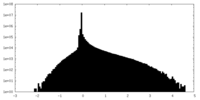
| 投影像・断面図 |
| ||||||||||||
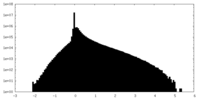
| 密度ヒストグラム |
-追加マップ: ES27 - Local Refinement - Local filtered map
| ファイル | emd_16276_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | ES27 - Local Refinement - Local filtered map | ||||||||||||
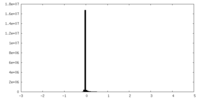
| 投影像・断面図 |
| ||||||||||||
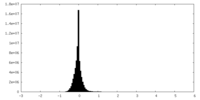
| 密度ヒストグラム |
-追加マップ: Nog1 - Local Refinement - Local filtered map
| ファイル | emd_16276_additional_3.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Nog1 - Local Refinement - Local filtered map | ||||||||||||
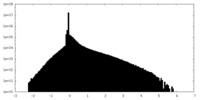
| 投影像・断面図 |
| ||||||||||||
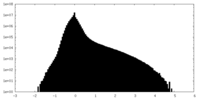
| 密度ヒストグラム |
-追加マップ: Spb1-MTD - Local Refinement - Local filtered map
| ファイル | emd_16276_additional_4.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Spb1-MTD - Local Refinement - Local filtered map | ||||||||||||
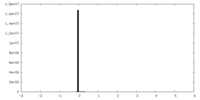
| 投影像・断面図 |
| ||||||||||||
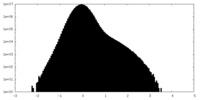
| 密度ヒストグラム |
-追加マップ: Consensus cryo-EM map - Local filtered map
| ファイル | emd_16276_additional_5.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Consensus cryo-EM map - Local filtered map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: Consensus cryo-EM map - Homogeneous Refinement
| ファイル | emd_16276_additional_6.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Consensus cryo-EM map - Homogeneous Refinement | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: Consensus cryo-EM map - Homogeneous Refinement - sharpened map
| ファイル | emd_16276_additional_7.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Consensus cryo-EM map - Homogeneous Refinement - sharpened map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Consensus cryo-EM map - half map A
| ファイル | emd_16276_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Consensus cryo-EM map - half map A | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Consensus cryo-EM map - half map B
| ファイル | emd_16276_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Consensus cryo-EM map - half map B | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Release state - ES27up (pre-60S)
| 全体 | 名称: Release state - ES27up (pre-60S) |
|---|---|
| 要素 |
|
-超分子 #1: Release state - ES27up (pre-60S)
| 超分子 | 名称: Release state - ES27up (pre-60S) / タイプ: complex / ID: 1 / キメラ: Yes / 親要素: 0 / 含まれる分子: #1-#60 |
|---|---|
| 由来(天然) | 生物種:  |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 平均電子線量: 46.8 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: SPOT SCAN / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 3.5 µm / 最小 デフォーカス(公称値): 0.5 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: EMDB MAP EMDB ID: |
|---|---|
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 3.0 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 29118 |
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
 ムービー
ムービー コントローラー
コントローラー




 Z
Z Y
Y X
X