+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of the CODV-IL13-RefAb triple complex | |||||||||
 マップデータ マップデータ | Pre-sharpened, unmasked map of CODV-IL13-RefAb after non-uniform refinement | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Cytosolic / bispecific / Fab / therapeutical / IMMUNE SYSTEM | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報negative regulation of complement-dependent cytotoxicity / cytokine receptor binding / negative regulation of endothelial cell apoptotic process / cytokine activity / immune response / extracellular space 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
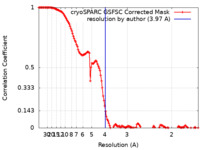
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.97 Å | |||||||||
 データ登録者 データ登録者 | Fernandez-Martinez D / Kandiah E | |||||||||
| 資金援助 |  フランス, 1件 フランス, 1件
| |||||||||
 引用 引用 |  ジャーナル: Sci Rep / 年: 2023 ジャーナル: Sci Rep / 年: 2023タイトル: Structural insights into the bi-specific cross-over dual variable antibody architecture by cryo-EM. 著者: David Fernandez-Martinez / Mark D Tully / Gordon Leonard / Magali Mathieu / Eaazhisai Kandiah /  要旨: Multi-specific antibodies (msAbs) are being developed as next generation antibody-based therapeutics. Knowledge of the three-dimensional structures, in the full antibody context, of their fragment ...Multi-specific antibodies (msAbs) are being developed as next generation antibody-based therapeutics. Knowledge of the three-dimensional structures, in the full antibody context, of their fragment antigen-binding (Fab) moieties with or without bound antigens is key to elucidating their therapeutic efficiency and stability. However, the flexibility of msAbs, a feature essential for their multi specificity, has hindered efforts in this direction. Cross-Over Dual Variable immunoglobulin (CODV) is a promising bispecific antibody format, designed to simultaneously target the interleukins IL4 and IL13. In this work we present the biophysical and structural characterisation of a CODV:IL13 complex in the full antibody context, using cryo-electron microscopy at an overall resolution of 4.2 Å. Unlike the 1:2 stoichiometry previously observed for CODV:IL4, CODV:IL13 shows a 1:1 stoichiometry. As well as providing details of the IL13-CODV binding interface, including the residues involved in the epitope-paratope region, the structure of CODV:IL13 also validates the use of labelling antibody as a new strategy for the single particle cryo-EM study of msAbs in complex with one, or more, antigens. This strategy reduced the inherent flexibility of the IL13 binding domain of CODV without inducing either structural changes at the epitope level or steric hindrance between the IL4 and IL13 binding regions of CODV. The work presented here thus also contributes to the development of methodology for the structural study of msAbs, a promising platform for cancer immunotherapy. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_16113.map.gz emd_16113.map.gz | 120 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-16113-v30.xml emd-16113-v30.xml emd-16113.xml emd-16113.xml | 20.7 KB 20.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_16113_fsc.xml emd_16113_fsc.xml | 15 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_16113.png emd_16113.png | 46 KB | ||
| マスクデータ |  emd_16113_msk_1.map emd_16113_msk_1.map | 244.1 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-16113.cif.gz emd-16113.cif.gz | 6.1 KB | ||
| その他 |  emd_16113_additional_1.map.gz emd_16113_additional_1.map.gz emd_16113_half_map_1.map.gz emd_16113_half_map_1.map.gz emd_16113_half_map_2.map.gz emd_16113_half_map_2.map.gz | 217.1 MB 226.3 MB 226.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-16113 http://ftp.pdbj.org/pub/emdb/structures/EMD-16113 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-16113 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-16113 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_16113_validation.pdf.gz emd_16113_validation.pdf.gz | 791.8 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_16113_full_validation.pdf.gz emd_16113_full_validation.pdf.gz | 791.4 KB | 表示 | |
| XML形式データ |  emd_16113_validation.xml.gz emd_16113_validation.xml.gz | 21.7 KB | 表示 | |
| CIF形式データ |  emd_16113_validation.cif.gz emd_16113_validation.cif.gz | 27.8 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16113 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16113 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16113 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16113 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8blqMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_16113.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_16113.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Pre-sharpened, unmasked map of CODV-IL13-RefAb after non-uniform refinement | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.827 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_16113_msk_1.map emd_16113_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: Final sharpened and masked map of CODV-IL13-RefAb
| ファイル | emd_16113_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Final sharpened and masked map of CODV-IL13-RefAb | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map B of the pre-sharpened, unmasked map...
| ファイル | emd_16113_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map B of the pre-sharpened, unmasked map of CODV-IL13-RefAb during non-uniform refinement | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map A of the pre-sharpened, unmasked map...
| ファイル | emd_16113_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map A of the pre-sharpened, unmasked map of CODV-IL13-RefAb during non-uniform refinement | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Ternary complex of CODV-Fab with IL13 and RefAb
| 全体 | 名称: Ternary complex of CODV-Fab with IL13 and RefAb |
|---|---|
| 要素 |
|
-超分子 #1: Ternary complex of CODV-Fab with IL13 and RefAb
| 超分子 | 名称: Ternary complex of CODV-Fab with IL13 and RefAb / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: CODV-Fab, heavy chain
| 分子 | 名称: CODV-Fab, heavy chain / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 36.907098 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: VQLQQSGPEL VKPGASVKIS CKASGYSFTS YWIHWIKQRP GQGLEWIGMI DPSDGETRLN QRFQGRATLT VDESTSTAYM QLRSPTSED SAVYYCTRLK EYGNYDSFYF DVWGAGTLVT VSSGEVQLKE SGPGLVAPGG SLSITCTVSG FSLTDSSINW V RQPPGKGL ...文字列: VQLQQSGPEL VKPGASVKIS CKASGYSFTS YWIHWIKQRP GQGLEWIGMI DPSDGETRLN QRFQGRATLT VDESTSTAYM QLRSPTSED SAVYYCTRLK EYGNYDSFYF DVWGAGTLVT VSSGEVQLKE SGPGLVAPGG SLSITCTVSG FSLTDSSINW V RQPPGKGL EWLGMIWGDG RIDYADALKS RLSISKDSSK SQVFLEMTSL RTDDTATYYC ARDGYFPYAM DFWGQGTSVT VS SGGASTK GPSVFPLAPS SKSTSGGTAA LGCLVKDYFP EPVTVSWNSG ALTSGVHTFP AVLQSSGLYS LSSVVTVPSS SLG TQTYIC NVNHKPSNTK VDKKVEP |
-分子 #2: CODV-Fab, light chain
| 分子 | 名称: CODV-Fab, light chain / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 35.726516 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: DIVLTQSPAS LAVSLGQRAT ISCRASESVD SYGQSYMHWY QQKAGQPPKL LIYLASNLES GVPARFSGSG SRTDFTLTID PVQAEDAAT YYCQQNAEDS RTFGGGTKLE IKGGGGGGGD IQMTQSPASL SVSVGDTITL TCHASQNIDV WLSWFQQKPG N IPKLLIYK ...文字列: DIVLTQSPAS LAVSLGQRAT ISCRASESVD SYGQSYMHWY QQKAGQPPKL LIYLASNLES GVPARFSGSG SRTDFTLTID PVQAEDAAT YYCQQNAEDS RTFGGGTKLE IKGGGGGGGD IQMTQSPASL SVSVGDTITL TCHASQNIDV WLSWFQQKPG N IPKLLIYK ASNLHTGVPS RFSGSGSGTG FTLTISSLQP EDIATYYCQQ AHSYPFTFGG GTKLEIKGGG GGRTVAAPSV FI FPPSDEQ LKSGTASVVC LLNNFYPREA KVQWKVDNAL QSGNSQESVT EQDSKDSTYS LSSTLTLSKA DYEKHKVYAC EVT HQGLSS PVTKSFNR |
-分子 #3: RefAb, light chain
| 分子 | 名称: RefAb, light chain / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 22.51002 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: YVLTQPPSVS VAPGKTARIT CGGNIIGSKL VHWYQQKPGQ APVLVIYDDG DRPSGIPERF SGSNSGNTAT LTISRVEAGD EADYYCQVW DTGSDPVVFG GGTKLTVLGQ PKAAPSVTLF PPSSEELQAN KATLVCLISD FYPGAVTVAW KADSSPVKAG V ETTTPSKQ ...文字列: YVLTQPPSVS VAPGKTARIT CGGNIIGSKL VHWYQQKPGQ APVLVIYDDG DRPSGIPERF SGSNSGNTAT LTISRVEAGD EADYYCQVW DTGSDPVVFG GGTKLTVLGQ PKAAPSVTLF PPSSEELQAN KATLVCLISD FYPGAVTVAW KADSSPVKAG V ETTTPSKQ SNNKYAASSY LSLTPEQWKS HRSYSCQVTH EGSTVEKTVA PTEC |
-分子 #4: Interleukin-13, human
| 分子 | 名称: Interleukin-13, human / タイプ: protein_or_peptide / ID: 4 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 12.077022 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: PPSTALRELI EELVNITQNQ KAPLCNGSMV WSINLTAGMY CAALESLINV SGCSAIEKTQ RMLSGFCPHK VSAGQFSSLH VRDTKIEVA QFVKDLLLHL KKLFREGQFN UniProtKB: Interleukin-13 |
-分子 #5: RefAb, heavy chain
| 分子 | 名称: RefAb, heavy chain / タイプ: protein_or_peptide / ID: 5 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 23.751457 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: VQLVQSGAEV KKPGASVKVS CKASGYTFTN YGLSWVRQAP GQGLEWMGWI SANNGDTNYG QEFQGRVTMT TDTSTSTAYM ELRSLRSDD TAVYYCARDS SSSWARWFFD LWGRGTLVTV SSASTKGPSV FPLAPSSKST SGGTAALGCL VKDYFPEPVT V SWNSGALT ...文字列: VQLVQSGAEV KKPGASVKVS CKASGYTFTN YGLSWVRQAP GQGLEWMGWI SANNGDTNYG QEFQGRVTMT TDTSTSTAYM ELRSLRSDD TAVYYCARDS SSSWARWFFD LWGRGTLVTV SSASTKGPSV FPLAPSSKST SGGTAALGCL VKDYFPEPVT V SWNSGALT SGVHTFPAVL QSSGLYSLSS VVTVPSSSLG TQTYICNVNH KPSNTKVDKK VEPK |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / 平均電子線量: 46.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 100.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 1.2 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: FLEXIBLE FIT |
|---|---|
| 得られたモデル |  PDB-8blq: |
 ムービー
ムービー コントローラー
コントローラー







 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)