+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Elongating E. coli 70S ribosome containing deacylated tRNA(iMet) in the P-site and m6AAA mRNA codon with cognate dipeptidyl-tRNA(Lys) in the A-site | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | m6A / mRNA modification / tRNA recognition / TRANSLATION | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報negative regulation of cytoplasmic translational initiation / ornithine decarboxylase inhibitor activity / transcription antitermination factor activity, RNA binding / misfolded RNA binding / Group I intron splicing / RNA folding / transcriptional attenuation / endoribonuclease inhibitor activity / RNA-binding transcription regulator activity / positive regulation of ribosome biogenesis ...negative regulation of cytoplasmic translational initiation / ornithine decarboxylase inhibitor activity / transcription antitermination factor activity, RNA binding / misfolded RNA binding / Group I intron splicing / RNA folding / transcriptional attenuation / endoribonuclease inhibitor activity / RNA-binding transcription regulator activity / positive regulation of ribosome biogenesis / negative regulation of cytoplasmic translation / four-way junction DNA binding / DnaA-L2 complex / translation repressor activity / negative regulation of translational initiation / regulation of mRNA stability / negative regulation of DNA-templated DNA replication initiation / mRNA regulatory element binding translation repressor activity / assembly of large subunit precursor of preribosome / positive regulation of RNA splicing / ribosome assembly / regulation of DNA-templated transcription elongation / transcription elongation factor complex / cytosolic ribosome assembly / response to reactive oxygen species / DNA endonuclease activity / transcription antitermination / translational initiation / regulation of cell growth / DNA-templated transcription termination / response to radiation / maintenance of translational fidelity / mRNA 5'-UTR binding / ribosome biogenesis / large ribosomal subunit / regulation of translation / transferase activity / ribosome binding / ribosomal small subunit biogenesis / ribosomal small subunit assembly / small ribosomal subunit / small ribosomal subunit rRNA binding / 5S rRNA binding / ribosomal large subunit assembly / cytosolic small ribosomal subunit / large ribosomal subunit rRNA binding / cytosolic large ribosomal subunit / cytoplasmic translation / tRNA binding / negative regulation of translation / rRNA binding / structural constituent of ribosome / ribosome / translation / response to antibiotic / negative regulation of DNA-templated transcription / mRNA binding / DNA binding / RNA binding / zinc ion binding / membrane / cytoplasm / cytosol 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.85 Å | |||||||||
 データ登録者 データ登録者 | Koziej L / Glatt S | |||||||||
| 資金援助 | European Union, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2023 ジャーナル: Nat Commun / 年: 2023タイトル: Modulation of translational decoding by mA modification of mRNA. 著者: Sakshi Jain / Lukasz Koziej / Panagiotis Poulis / Igor Kaczmarczyk / Monika Gaik / Michal Rawski / Namit Ranjan / Sebastian Glatt / Marina V Rodnina /   要旨: N-methyladenosine (mA) is an abundant, dynamic mRNA modification that regulates key steps of cellular mRNA metabolism. mA in the mRNA coding regions inhibits translation elongation. Here, we show how ...N-methyladenosine (mA) is an abundant, dynamic mRNA modification that regulates key steps of cellular mRNA metabolism. mA in the mRNA coding regions inhibits translation elongation. Here, we show how mA modulates decoding in the bacterial translation system using a combination of rapid kinetics, smFRET and single-particle cryo-EM. We show that, while the modification does not impair the initial binding of aminoacyl-tRNA to the ribosome, in the presence of mA fewer ribosomes complete the decoding process due to the lower stability of the complexes and enhanced tRNA drop-off. The mRNA codon adopts a π-stacked codon conformation that is remodeled upon aminoacyl-tRNA binding. mA does not exclude canonical codon-anticodon geometry, but favors alternative more dynamic conformations that are rejected by the ribosome. These results highlight how modifications outside the Watson-Crick edge can still interfere with codon-anticodon base pairing and complex recognition by the ribosome, thereby modulating the translational efficiency of modified mRNAs. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_16062.map.gz emd_16062.map.gz | 439.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-16062-v30.xml emd-16062-v30.xml emd-16062.xml emd-16062.xml | 91.3 KB 91.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_16062_fsc.xml emd_16062_fsc.xml | 16.9 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_16062.png emd_16062.png | 173.4 KB | ||
| マスクデータ |  emd_16062_msk_1.map emd_16062_msk_1.map | 512 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-16062.cif.gz emd-16062.cif.gz | 15.4 KB | ||
| その他 |  emd_16062_additional_1.map.gz emd_16062_additional_1.map.gz emd_16062_half_map_1.map.gz emd_16062_half_map_1.map.gz emd_16062_half_map_2.map.gz emd_16062_half_map_2.map.gz | 257.1 MB 475.5 MB 475.5 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-16062 http://ftp.pdbj.org/pub/emdb/structures/EMD-16062 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-16062 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-16062 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_16062_validation.pdf.gz emd_16062_validation.pdf.gz | 1.2 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_16062_full_validation.pdf.gz emd_16062_full_validation.pdf.gz | 1.2 MB | 表示 | |
| XML形式データ |  emd_16062_validation.xml.gz emd_16062_validation.xml.gz | 26.5 KB | 表示 | |
| CIF形式データ |  emd_16062_validation.cif.gz emd_16062_validation.cif.gz | 34.9 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16062 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16062 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16062 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16062 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8bhnMC  8bf7C  8bgeC  8bghC  8bh4C  8bhjC  8bhlC  8bhpC  8bilC  8bimC C: 同じ文献を引用 ( M: このマップから作成された原子モデル |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_16062.map.gz / 形式: CCP4 / 大きさ: 512 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_16062.map.gz / 形式: CCP4 / 大きさ: 512 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.86 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_16062_msk_1.map emd_16062_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
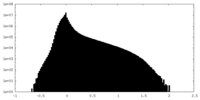
| 密度ヒストグラム |
-追加マップ: Map combined from half maps used for generation...
| ファイル | emd_16062_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Map combined from half maps used for generation of the final high-resolution DeepEM enhanced map. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_16062_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_16062_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
+全体 : Elongating E. coli 70S ribosome with deacylated tRNA(iMet) in the...
+超分子 #1: Elongating E. coli 70S ribosome with deacylated tRNA(iMet) in the...
+超分子 #2: 50S subunit
+超分子 #3: 30S subunit
+超分子 #4: 23S and 5S rRNA
+超分子 #5: 16S rRNA
+超分子 #6: mRNA and tRNA
+分子 #1: 50S ribosomal protein L2
+分子 #2: 50S ribosomal protein L3
+分子 #3: 50S ribosomal protein L4
+分子 #4: 50S ribosomal protein L5
+分子 #5: 50S ribosomal protein L6
+分子 #6: 50S ribosomal protein L9
+分子 #7: 50S ribosomal protein L13
+分子 #8: 50S ribosomal protein L14
+分子 #9: 50S ribosomal protein L15
+分子 #10: 50S ribosomal protein L16
+分子 #11: 50S ribosomal protein L17
+分子 #12: 50S ribosomal protein L18
+分子 #13: 50S ribosomal protein L19
+分子 #14: 50S ribosomal protein L20
+分子 #15: 50S ribosomal protein L21
+分子 #16: 50S ribosomal protein L22
+分子 #17: 50S ribosomal protein L23
+分子 #18: 50S ribosomal protein L24
+分子 #19: 50S ribosomal protein L25
+分子 #20: 50S ribosomal protein L27
+分子 #21: 50S ribosomal protein L28
+分子 #22: 50S ribosomal protein L29
+分子 #23: 50S ribosomal protein L30
+分子 #24: 50S ribosomal protein L31
+分子 #25: 50S ribosomal protein L32
+分子 #26: 50S ribosomal protein L33
+分子 #27: 50S ribosomal protein L34
+分子 #28: 50S ribosomal protein L35
+分子 #29: 50S ribosomal protein L36
+分子 #30: 30S ribosomal protein S2
+分子 #31: 30S ribosomal protein S3
+分子 #32: 30S ribosomal protein S4
+分子 #33: 30S ribosomal protein S5
+分子 #34: 30S ribosomal protein S6
+分子 #35: 30S ribosomal protein S7
+分子 #36: 30S ribosomal protein S8
+分子 #37: 30S ribosomal protein S9
+分子 #38: 30S ribosomal protein S10
+分子 #39: 30S ribosomal protein S11
+分子 #40: 30S ribosomal protein S12
+分子 #41: 30S ribosomal protein S13
+分子 #42: 30S ribosomal protein S14
+分子 #43: 30S ribosomal protein S15
+分子 #44: 30S ribosomal protein S16
+分子 #45: 30S ribosomal protein S17
+分子 #46: 30S ribosomal protein S18
+分子 #47: 30S ribosomal protein S19
+分子 #48: 30S ribosomal protein S20
+分子 #49: 30S ribosomal protein S21
+分子 #56: fMet-Lys dipeptide
+分子 #50: 23S rRNA RRLG-RRNA
+分子 #51: 5S rRNA
+分子 #52: 16S rRNA RRSB-RRNA
+分子 #53: mRNA
+分子 #54: Deacylated P-site tRNA(fMet)
+分子 #55: Dipeptidyl A-site tRNA(Lys)
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 10 mg/mL | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
詳細: 1x TAKM7 buffer | |||||||||||||||
| グリッド | モデル: Quantifoil R2/1 / 材質: COPPER / メッシュ: 200 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 60 sec. / 前処理 - 雰囲気: AIR | |||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV | |||||||||||||||
| 詳細 | Initiation complex containing: 1x TAKM7 buffer 1 mM DTT (dithiothreitol) 1 mM GTP (guanosine 5'-triphosphate) 2.0 uM 70S ribosomes 3.0 uM IF1, IF2, IF3 mix 6.0 uM A-site m6AAA mRNA 3.0 uM fMet-tRNAfMet Mixed 1:1 with a ternary complex containing: 1x TAKM7 buffer 3 mM PEP (phosphoenolpyruvate) 1 mM GTP (guanosine 5'-triphosphate) 1 mM DTT (dithiothreitol) 1% PK (pyruvate kinase) 0.02 uM EF-Ts 1.5 uM EF-Tu 0.3 uM Lys-tRNALys Incubated on ice for 60 seconds Final concentration of the sample: 20 A260 (absorbance at 260 nm, 10 mm path), 10 A280 (absorbance at 280 nm, 10 mm path) |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) デジタル化 - サイズ - 横: 5760 pixel / デジタル化 - サイズ - 縦: 4092 pixel / 撮影したグリッド数: 1 / 実像数: 9366 / 平均露光時間: 1.82 sec. / 平均電子線量: 40.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 倍率(補正後): 105000 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.1 µm / 最小 デフォーカス(公称値): 0.9 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 詳細 | Following rigid body fitting we used online Namdiantor tool to perform initial flexible fitting: https://namdinator.au.dk/namdinator/ |
|---|---|
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT |
| 得られたモデル |  PDB-8bhn: |
-原子モデル構築 2
| 精密化 | 空間: REAL / プロトコル: FLEXIBLE FIT |
|---|---|
| 得られたモデル |  PDB-8bhn: |
 ムービー
ムービー コントローラー
コントローラー

















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)