+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-1600 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Motor mechanism for protein threading through Hsp104 | |||||||||
 マップデータ マップデータ | Yeast Hsp104 N728A 3D density map. Hexamer formed in the presence of ATP. | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Clp/Hsp100 / AAA+ / protein remodeling / disaggregation / molecular machines / heat shock protein / Hsp104 / ClpB / yeast prions | |||||||||
| 生物種 |  | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 11.5 Å | |||||||||
 データ登録者 データ登録者 | Wendler P / Shorter J / Snead D / Plisson C / Clare DK / Lindquist S / Saibil H | |||||||||
 引用 引用 |  ジャーナル: Mol Cell / 年: 2009 ジャーナル: Mol Cell / 年: 2009タイトル: Motor mechanism for protein threading through Hsp104. 著者: Petra Wendler / James Shorter / David Snead / Celia Plisson / Daniel K Clare / Susan Lindquist / Helen R Saibil /  要旨: The protein-remodeling machine Hsp104 dissolves amorphous aggregates as well as ordered amyloid assemblies such as yeast prions. Force generation originates from a tandem AAA+ (ATPases associated ...The protein-remodeling machine Hsp104 dissolves amorphous aggregates as well as ordered amyloid assemblies such as yeast prions. Force generation originates from a tandem AAA+ (ATPases associated with various cellular activities) cassette, but the mechanism and allostery of this action remain to be established. Our cryoelectron microscopy maps of Hsp104 hexamers reveal substantial domain movements upon ATP binding and hydrolysis in the first nucleotide-binding domain (NBD1). Fitting atomic models of Hsp104 domains to the EM density maps plus supporting biochemical measurements show how the domain movements displace sites bearing the substrate-binding tyrosine loops. This provides the structural basis for N- to C-terminal substrate threading through the central cavity, enabling a clockwise handover of substrate in the NBD1 ring and coordinated substrate binding between NBD1 and NBD2. Asymmetric reconstructions of Hsp104 in the presence of ATPgammaS or ATP support sequential rather than concerted ATP hydrolysis in the NBD1 ring. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_1600.map.gz emd_1600.map.gz | 311.5 KB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-1600-v30.xml emd-1600-v30.xml emd-1600.xml emd-1600.xml | 9.6 KB 9.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  1600.png 1600.png | 338.6 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-1600 http://ftp.pdbj.org/pub/emdb/structures/EMD-1600 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1600 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1600 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_1600_validation.pdf.gz emd_1600_validation.pdf.gz | 198.8 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_1600_full_validation.pdf.gz emd_1600_full_validation.pdf.gz | 197.9 KB | 表示 | |
| XML形式データ |  emd_1600_validation.xml.gz emd_1600_validation.xml.gz | 5.5 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1600 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1600 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1600 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1600 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_1600.map.gz / 形式: CCP4 / 大きさ: 7.8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_1600.map.gz / 形式: CCP4 / 大きさ: 7.8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Yeast Hsp104 N728A 3D density map. Hexamer formed in the presence of ATP. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 2.8 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
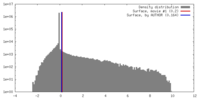
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : S. cerevisiae Hsp104 N728A ATP
| 全体 | 名称: S. cerevisiae Hsp104 N728A ATP |
|---|---|
| 要素 |
|
-超分子 #1000: S. cerevisiae Hsp104 N728A ATP
| 超分子 | 名称: S. cerevisiae Hsp104 N728A ATP / タイプ: sample / ID: 1000 / 集合状態: hexamer / Number unique components: 2 |
|---|---|
| 分子量 | 理論値: 612 KDa |
-分子 #1: Hsp104 N728A
| 分子 | 名称: Hsp104 N728A / タイプ: protein_or_peptide / ID: 1 / Name.synonym: Hsp104 N728A / 詳細: Hexamers were formed in the presence of 5mM ADP / コピー数: 6 / 集合状態: hexamer / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 102 KDa |
| 組換発現 | 生物種:  |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.3 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 詳細: 20 mM HEPES pH 7.5, 20 mM NaCl, 10 mM MgCl2, 1 mM DTT, 2-5mM ATP |
| グリッド | 詳細: 300 mesh copper grid- holey carbon film |
| 凍結 | 凍結剤: ETHANE / 装置: HOMEMADE PLUNGER / 詳細: Vitrification instrument: home made 手法: The grids were blotted for 2-3 sec and immediately plunged into liquid ethane |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI F20 |
|---|---|
| 温度 | 最低: 77 K / 最高: 85 K / 平均: 77 K |
| アライメント法 | Legacy - 非点収差: corrected for at specimen level |
| 日付 | 2004年1月28日 |
| 撮影 | カテゴリ: FILM / フィルム・検出器のモデル: KODAK SO-163 FILM / デジタル化 - スキャナー: ZEISS SCAI / デジタル化 - サンプリング間隔: 7 µm / 実像数: 21 / 平均電子線量: 15 e/Å2 / Od range: 1 / ビット/ピクセル: 8 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 50000 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2 mm / 最大 デフォーカス(公称値): 3.5 µm / 最小 デフォーカス(公称値): 1.5 µm / 倍率(公称値): 50000 |
| 試料ステージ | 試料ホルダー: single tilt cryo / 試料ホルダーモデル: GATAN LIQUID NITROGEN |
| 実験機器 |  モデル: Tecnai F20 / 画像提供: FEI Company |
- 画像解析
画像解析
| CTF補正 | 詳細: phase flipping, each particle |
|---|---|
| 最終 再構成 | 想定した対称性 - 点群: C6 (6回回転対称) / アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 11.5 Å / 解像度の算出法: FSC 0.5 CUT-OFF / ソフトウェア - 名称: IMAGIC SPIDER MRC / 詳細: 6-fold symmetrised reconstruction / 使用した粒子像数: 4046 |
 ムービー
ムービー コントローラー
コントローラー







 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)