+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
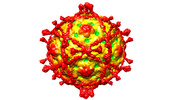
| タイトル | Complex of Echovirus 11 with its attaching receptor decay-accelerating factor (CD55) | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Echovirus / receptor / DAF / CD55 / VIRUS | |||||||||
| 生物種 |   Echovirus E11 (ウイルス) / Echovirus E11 (ウイルス) /  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.1 Å | |||||||||
 データ登録者 データ登録者 | Stuart DI / Ren J / Zhou D / Qin L | |||||||||
| 資金援助 |  英国, 1件 英国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Viruses / 年: 2022 ジャーナル: Viruses / 年: 2022タイトル: Switching of Receptor Binding Poses between Closely Related Enteroviruses. 著者: Daming Zhou / Ling Qin / Helen M E Duyvesteyn / Yuguang Zhao / Tzou-Yien Lin / Elizabeth E Fry / Jingshan Ren / Kuan-Ying A Huang / David I Stuart /   要旨: Echoviruses, for which there are currently no approved vaccines or drugs, are responsible for a range of human diseases, for example echovirus 11 (E11) is a major cause of serious neonatal morbidity ...Echoviruses, for which there are currently no approved vaccines or drugs, are responsible for a range of human diseases, for example echovirus 11 (E11) is a major cause of serious neonatal morbidity and mortality. Decay-accelerating factor (DAF, also known as CD55) is an attachment receptor for E11. Here, we report the structure of the complex of E11 and the full-length ectodomain of DAF (short consensus repeats, SCRs, 1-4) at 3.1 Å determined by cryo-electron microscopy (cryo-EM). SCRs 3 and 4 of DAF interact with E11 at the southern rim of the canyon via the VP2 EF and VP3 BC loops. We also observe an unexpected interaction between the N-linked glycan (residue 95 of DAF) and the VP2 BC loop of E11. DAF is a receptor for at least 20 enteroviruses and we classify its binding patterns from reported DAF/virus complexes into two distinct positions and orientations, named as E6 and E11 poses. Whilst 60 DAF molecules can attach to the virion in the E6 pose, no more than 30 can attach to E11 due to steric restrictions. Analysis of the distinct modes of interaction and structure and sequence-based phylogenies suggests that the two modes evolved independently, with the E6 mode likely found earlier. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_15920.map.gz emd_15920.map.gz | 226.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-15920-v30.xml emd-15920-v30.xml emd-15920.xml emd-15920.xml | 17.3 KB 17.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_15920.png emd_15920.png | 67.7 KB | ||
| Filedesc metadata |  emd-15920.cif.gz emd-15920.cif.gz | 6.1 KB | ||
| その他 |  emd_15920_half_map_1.map.gz emd_15920_half_map_1.map.gz emd_15920_half_map_2.map.gz emd_15920_half_map_2.map.gz | 159.1 MB 159 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-15920 http://ftp.pdbj.org/pub/emdb/structures/EMD-15920 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-15920 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-15920 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_15920_validation.pdf.gz emd_15920_validation.pdf.gz | 956.4 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_15920_full_validation.pdf.gz emd_15920_full_validation.pdf.gz | 955.9 KB | 表示 | |
| XML形式データ |  emd_15920_validation.xml.gz emd_15920_validation.xml.gz | 15.4 KB | 表示 | |
| CIF形式データ |  emd_15920_validation.cif.gz emd_15920_validation.cif.gz | 18.1 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15920 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15920 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15920 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15920 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_15920.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_15920.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.05 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #1
| ファイル | emd_15920_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
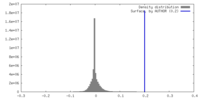
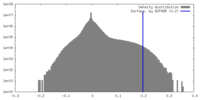
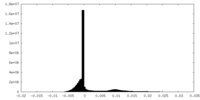
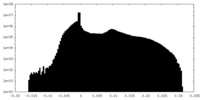
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_15920_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
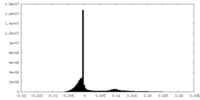
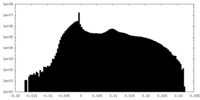
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Complex of Echovirus 11 with its attaching receptor DECAY ACCELER...
| 全体 | 名称: Complex of Echovirus 11 with its attaching receptor DECAY ACCELERATING FACTOR (CD55) |
|---|---|
| 要素 |
|
-超分子 #1: Complex of Echovirus 11 with its attaching receptor DECAY ACCELER...
| 超分子 | 名称: Complex of Echovirus 11 with its attaching receptor DECAY ACCELERATING FACTOR (CD55) タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#5 |
|---|---|
| 由来(天然) | 生物種:   Echovirus E11 (ウイルス) Echovirus E11 (ウイルス) |
-分子 #1: VP1
| 分子 | 名称: VP1 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Echovirus E11 (ウイルス) Echovirus E11 (ウイルス) |
| 分子量 | 理論値: 32.838828 KDa |
| 配列 | 文字列: GDVVEAIEGA VARVADTISS GPTNSQAVPA LTAVETGHTS QVVPGDTMQT RHVKNYHSRS ESTIENFLSR SACVYMGEYY TTNTDETKR FASWTINARR MVQMRRKLEM FTYVRFDVEV TFVITSKQDQ GTQLGQDMPP LTHQIMYIPP GGPIPKSTTD Y AWQTSTNP ...文字列: GDVVEAIEGA VARVADTISS GPTNSQAVPA LTAVETGHTS QVVPGDTMQT RHVKNYHSRS ESTIENFLSR SACVYMGEYY TTNTDETKR FASWTINARR MVQMRRKLEM FTYVRFDVEV TFVITSKQDQ GTQLGQDMPP LTHQIMYIPP GGPIPKSTTD Y AWQTSTNP SIFWTEGNAP PRMSIPFVSI GNAYSNFYDG WSHFSQNGVY GYNTLNNMGQ LYMRHVNGPS PLPMTSIVRV YF KPKHVKA WVPRPPRLCQ YKNASTVNFS STNITDKRDS ITHVPDTVKP DVTTH |
-分子 #2: VP2
| 分子 | 名称: VP2 / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Echovirus E11 (ウイルス) Echovirus E11 (ウイルス) |
| 分子量 | 理論値: 28.886428 KDa |
| 配列 | 文字列: SPSAEECGYS DRVRSITLGN STITTQECAN VVVAYGRWPE YLSDKEATAE DQPTQPDVAT CRFYTLESVT WEKDSPGWWW KFPDALKDM GLFGQNMYYH YLGRAGYTIH VQCNASKFHQ GCLLVVCVPE AEMGCSDVGG TVNEHAISEG EIAKKFSATA T NGAHTVQS ...文字列: SPSAEECGYS DRVRSITLGN STITTQECAN VVVAYGRWPE YLSDKEATAE DQPTQPDVAT CRFYTLESVT WEKDSPGWWW KFPDALKDM GLFGQNMYYH YLGRAGYTIH VQCNASKFHQ GCLLVVCVPE AEMGCSDVGG TVNEHAISEG EIAKKFSATA T NGAHTVQS IVTNAGMGVG VGNLTIYPHQ WVNLRTNNSA TIVMPYINSV PMDNMFRHHN FTLMIIPFVS LDYSSDASTY VP ITVTVAP MCAEYNGLRL ATSLQ |
-分子 #3: VP3
| 分子 | 名称: VP3 / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Echovirus E11 (ウイルス) Echovirus E11 (ウイルス) |
| 分子量 | 理論値: 26.029674 KDa |
| 配列 | 文字列: GLPVMNTPGS NQFLTSDDFQ SPSAMPQFDV TPELDIPGEV KNLMEIAEVD SVVPVNNVVG KLDTMDIFRI PVQSGNHQST QVFGFQVQP GLDSVFKHTL LGEILNYYAH WSGSVKLTFV FCGSAMATGK FLLAYSPPGA NAPKTRKDAM LGTHVIWDVG L QSSCVLCI ...文字列: GLPVMNTPGS NQFLTSDDFQ SPSAMPQFDV TPELDIPGEV KNLMEIAEVD SVVPVNNVVG KLDTMDIFRI PVQSGNHQST QVFGFQVQP GLDSVFKHTL LGEILNYYAH WSGSVKLTFV FCGSAMATGK FLLAYSPPGA NAPKTRKDAM LGTHVIWDVG L QSSCVLCI PWISQTHYRL VHQDEYTSAG NVTCWYQTGI VVPAGTPTLC SIMCFVSACN DFSVRLLKDT PFIEQSALLQ |
-分子 #4: VP4
| 分子 | 名称: VP4 / タイプ: protein_or_peptide / ID: 4 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Echovirus E11 (ウイルス) Echovirus E11 (ウイルス) |
| 分子量 | 理論値: 7.498273 KDa |
| 配列 | 文字列: MGAQVSTQKT GAHETGLNAS GNSIIHYTNI NYYKDAASNS ANRQDFTQDP GKFTEPVKDI MIKSMPALN |
-分子 #5: DECAY ACCELERATING FACTOR (CD55)
| 分子 | 名称: DECAY ACCELERATING FACTOR (CD55) / タイプ: protein_or_peptide / ID: 5 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 29.771059 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: (MSE)GCVAETGDC GLPPDVPNAQ PALEGRTSFP EDTVITYKCE ESFVKIPGEK DSVICLKGSQ WSDIEEFCNR SCEVPT RLN SASLKQPYIT QNYFPVGTVV EYECRPGYRR EPSLSPKLTC LQNLKWSTAV EFCKKKSCPN PGEIRNGQID VPGGILF GA ...文字列: (MSE)GCVAETGDC GLPPDVPNAQ PALEGRTSFP EDTVITYKCE ESFVKIPGEK DSVICLKGSQ WSDIEEFCNR SCEVPT RLN SASLKQPYIT QNYFPVGTVV EYECRPGYRR EPSLSPKLTC LQNLKWSTAV EFCKKKSCPN PGEIRNGQID VPGGILF GA TISFSCNTGY KLFGSTSSFC LISGSSVQWS DPLPECREIY CPAPPQIDNG IIQGERDHYG YRQSVTYACN KGFT (MSE)IGEH SIYCTVNNDE GEWSGPPPEC RGGTKHHHHH H |
-分子 #6: SPHINGOSINE
| 分子 | 名称: SPHINGOSINE / タイプ: ligand / ID: 6 / コピー数: 1 / 式: SPH |
|---|---|
| 分子量 | 理論値: 299.492 Da |
| Chemical component information |  ChemComp-SPH: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1 mg/mL |
|---|---|
| 緩衝液 | pH: 7.4 |
| 凍結 | 凍結剤: ETHANE-PROPANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON IV (4k x 4k) 平均電子線量: 41.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: SPOT SCAN / 撮影モード: DARK FIELD / 最大 デフォーカス(公称値): 1.0 µm / 最小 デフォーカス(公称値): 3.0 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: PDB ENTRY PDBモデル - PDB ID: |
|---|---|
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 3.1 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 6394 |
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
 ムービー
ムービー コントローラー
コントローラー






 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)