[日本語] English
 万見
万見- EMDB-15651: Structure of the giant inhibitor of apoptosis, BIRC6 (multibody map 3) -
+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of the giant inhibitor of apoptosis, BIRC6 (multibody map 3) | |||||||||
 マップデータ マップデータ | Multibody map 3 | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | E2/E3 ubiquitin ligase / APOPTOSIS | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報spongiotrophoblast layer development / labyrinthine layer development / ALK mutants bind TKIs / Flemming body / microtubule organizing center / cysteine-type endopeptidase inhibitor activity / ubiquitin conjugating enzyme activity / regulation of cytokinesis / negative regulation of extrinsic apoptotic signaling pathway / RING-type E3 ubiquitin transferase ...spongiotrophoblast layer development / labyrinthine layer development / ALK mutants bind TKIs / Flemming body / microtubule organizing center / cysteine-type endopeptidase inhibitor activity / ubiquitin conjugating enzyme activity / regulation of cytokinesis / negative regulation of extrinsic apoptotic signaling pathway / RING-type E3 ubiquitin transferase / trans-Golgi network / spindle pole / ubiquitin-protein transferase activity / Signaling by ALK fusions and activated point mutants / regulation of cell population proliferation / midbody / cell population proliferation / protein ubiquitination / endosome / cell cycle / cell division / protein phosphorylation / centrosome / apoptotic process / positive regulation of cell population proliferation / negative regulation of apoptotic process / membrane / nucleus / cytosol 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.1 Å | |||||||||
 データ登録者 データ登録者 | Dietz L / Elliott PR | |||||||||
| 資金援助 |  英国, 1件 英国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Science / 年: 2023 ジャーナル: Science / 年: 2023タイトル: Structural basis for SMAC-mediated antagonism of caspase inhibition by the giant ubiquitin ligase BIRC6. 著者: Larissa Dietz / Cara J Ellison / Carlos Riechmann / C Keith Cassidy / F Daniel Felfoldi / Adán Pinto-Fernández / Benedikt M Kessler / Paul R Elliott /  要旨: Certain inhibitor of apoptosis (IAP) family members are sentinel proteins that prevent untimely cell death by inhibiting caspases. Antagonists, including second mitochondria-derived activator of ...Certain inhibitor of apoptosis (IAP) family members are sentinel proteins that prevent untimely cell death by inhibiting caspases. Antagonists, including second mitochondria-derived activator of caspases (SMAC), regulate IAPs and drive cell death. Baculoviral IAP repeat-containing protein 6 (BIRC6), a giant IAP with dual E2 and E3 ubiquitin ligase activity, regulates programmed cell death through unknown mechanisms. We show that BIRC6 directly restricts executioner caspase-3 and -7 and ubiquitinates caspase-3, -7, and -9, working exclusively with noncanonical E1, UBA6. Notably, we show that SMAC suppresses both mechanisms. Cryo-electron microscopy structures of BIRC6 alone and in complex with SMAC reveal that BIRC6 is an antiparallel dimer juxtaposing the substrate-binding module against the catalytic domain. Furthermore, we discover that SMAC multisite binding to BIRC6 results in a subnanomolar affinity interaction, enabling SMAC to competitively displace caspases, thus antagonizing BIRC6 anticaspase function. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_15651.map.gz emd_15651.map.gz | 96.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-15651-v30.xml emd-15651-v30.xml emd-15651.xml emd-15651.xml | 20.1 KB 20.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_15651.png emd_15651.png | 18.9 KB | ||
| マスクデータ |  emd_15651_msk_1.map emd_15651_msk_1.map | 103 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-15651.cif.gz emd-15651.cif.gz | 7.9 KB | ||
| その他 |  emd_15651_half_map_1.map.gz emd_15651_half_map_1.map.gz emd_15651_half_map_2.map.gz emd_15651_half_map_2.map.gz | 56.7 MB 56.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-15651 http://ftp.pdbj.org/pub/emdb/structures/EMD-15651 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-15651 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-15651 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_15651_validation.pdf.gz emd_15651_validation.pdf.gz | 983.3 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_15651_full_validation.pdf.gz emd_15651_full_validation.pdf.gz | 982.9 KB | 表示 | |
| XML形式データ |  emd_15651_validation.xml.gz emd_15651_validation.xml.gz | 13 KB | 表示 | |
| CIF形式データ |  emd_15651_validation.cif.gz emd_15651_validation.cif.gz | 15.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15651 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15651 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15651 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15651 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_15651.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_15651.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Multibody map 3 | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.86 Å | ||||||||||||||||||||||||||||||||||||
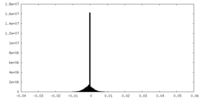
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
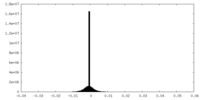
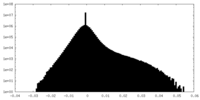
-マスク #1
| ファイル |  emd_15651_msk_1.map emd_15651_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
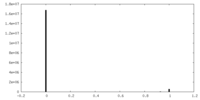
| 密度ヒストグラム |
-ハーフマップ: 2nd half map multibody map 3
| ファイル | emd_15651_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | 2nd half map multibody map 3 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
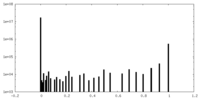
| 密度ヒストグラム |
-ハーフマップ: 1st half map multibody map 3
| ファイル | emd_15651_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | 1st half map multibody map 3 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
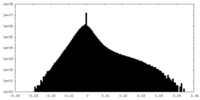
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Anti-parallel homodimer
| 全体 | 名称: Anti-parallel homodimer |
|---|---|
| 要素 |
|
-超分子 #1: Anti-parallel homodimer
| 超分子 | 名称: Anti-parallel homodimer / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 1 MDa |
-分子 #1: BIRC6
| 分子 | 名称: BIRC6 / タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO / EC番号: RING-type E3 ubiquitin transferase |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: GPMVTGGGAA PPGTVTEPLP SVIVLSAGRK MAAAAAAASG PGCSSAAGAG AAGVSEWLVL RDGCMHCDAD GLHSLSYHPA LNAILAVTSR GTIKVIDGTS GATLQASALS AKPGGQVKCQ YISAVDKVIF VDDYAVGCRK DLNGILLLDT ALQTPVSKQD DVVQLELPVT ...文字列: GPMVTGGGAA PPGTVTEPLP SVIVLSAGRK MAAAAAAASG PGCSSAAGAG AAGVSEWLVL RDGCMHCDAD GLHSLSYHPA LNAILAVTSR GTIKVIDGTS GATLQASALS AKPGGQVKCQ YISAVDKVIF VDDYAVGCRK DLNGILLLDT ALQTPVSKQD DVVQLELPVT EAQQLLSACL EKVDISSTEG YDLFITQLKD GLKNTSHETA ANHKVAKWAT VTFHLPHHVL KSIASAIVNE LKKINQNVAA LPVASSVMDR LSYLLPSARP ELGVGPGRSV DRSLMYSEAN RRETFTSWPH VGYRWAQPDP MAQAGFYHQP ASSGDDRAMC FTCSVCLVCW EPTDEPWSEH ERHSPNCPFV KGEHTQNVPL SVTLATSPAQ FPCTDGTDRI SCFGSGSCPH FLAAATKRGK ICIWDVSKLM KVHLKFEINA YDPAIVQQLI LSGDPSSGVD SRRPTLAWLE DSSSCSDIPK LEGDSDDLLE DSDSEEHSRS DSVTGHTSQK EAMEVSLDIT ALSILQQPEK LQWEIVANVL EDTVKDLEEL GANPCLTNSK SEKTKEKHQE QHNIPFPCLL AGGLLTYKSP ATSPISSNSH RSLDGLSRTQ GESISEQGST DNESCTNSEL NSPLVRRTLP VLLLYSIKES DEKAGKIFSQ MNNIMSKSLH DDGFTVPQII EMELDSQEQL LLQDPPVTYI QQFADAAANL TSPDSEKWNS VFPKPGTLVQ CLRLPKFAEE ENLCIDSITP CADGIHLLVG LRTCPVESLS AINQVEALNN LNKLNSALCN RRKGELESNL AVVNGANISV IQHESPADVQ TPLIIQPEQR NVSGGYLVLY KMNYATRIVT LEEEPIKIQH IKDPQDTITS LILLPPDILD NREDDCEEPI EDMQLTSKNG FEREKTSDIS TLGHLVITTQ GGYVKILDLS NFEILAKVEP PKKEGTEEQD TFVSVIYCSG TDRLCACTKG GELHFLQIGG TCDDIDEADI LVDGSLSKGI EPSSEGSKPL SNPSSPGISG VDLLVDQPFT LEILTSLVEL TRFETLTPRF SATVPPCWVE VQQEQQQRRH PQHLHQQHHG DAAQHTRTWK LQTDSNSWDE HVFELVLPKA CMVGHVDFKF VLNSNITNIP QIQVTLLKNK APGLGKVNAL NIEVEQNGKP SLVDLNEEMQ HMDVEESQCL RLCPFLEDHK EDILCGPVWL ASGLDLSGHA GMLTLTSPKL VKGMAGGKYR SFLIHVKAVN ERGTEEICNG GMRPVVRLPS LKHQSNKGYS LASLLAKVAA GKEKSSNVKN ENTSGTRKSE NLRGCDLLQE VSVTIRRFKK TSISKERVQR CAMLQFSEFH EKLLNTLCRK TDDGQITEHA QSLVLDTLCW LAGVHSNGPG SSKEGNENLL SKTRKFLSDI VRVCFFEAGR SIAHKCARFL ALCISNGKCD PCQPAFGPVL LKALLDNMSF LPAATTGGSV YWYFVLLNYV KDEDLAGCST ACASLLTAVS RQLQDRLTPM EALLQTRYGL YSSPFDPVLF DLEMSGSSCK NVYNSSIGVQ SDEIDLSDVL SGNGKVSSCT AAEGSFTSLT GLLEVEPLHF TCVSTSDGTR IERDDAMSSF GVTPAVGGLS SGTVGEASTA LSSAAQVALQ SLSHAMASAE QQLQVLQEKQ QQLLKLQQQK AKLEAKLHQT TAAAAAAASA VGPVHNSVPS NPVAAPGFFI HPSDVIPPTP KTTPLFMTPP LTPPNEAVSV VINAELAQLF PGSVIDPPAV NLAAHNKNSN KSRMNPLGSG LALAISHASH FLQPPPHQSI IIERMHSGAR RFVTLDFGRP ILLTDVLIPT CGDLASLSID IWTLGEEVDG RRLVVATDIS THSLILHDLI PPPVCRFMKI TVIGRYGSTN ARAKIPLGFY YGHTYILPWE SELKLMHDPL KGEGESANQP EIDQHLAMMV ALQEDIQCRY NLACHRLETL LQSIDLPPLN SANNAQYFLR KPDKAVEEDS RVFSAYQDCI QLQLQLNLAH NAVQRLKVAL GASRKMLSET SNPEDLIQTS STEQLRTIIR YLLDTLLSLL HASNGHSVPA VLQSTFHAQA CEELFKHLCI SGTPKIRLHT GLLLVQLCGG ERWWGQFLSN VLQELYNSEQ LLIFPQDRVF MLLSCIGQRS LSNSGVLESL LNLLDNLLSP LQPQLPMHRR TEGVLDIPMI SWVVMLVSRL LDYVATVEDE AAAAKKPLNG NQWSFINNNL HTQSLNRSSK GSSSLDRLYS RKIRKQLVHH KQQLNLLKAK QKALVEQMEK EKIQSNKGSS YKLLVEQAKL KQATSKHFKD LIRLRRTAEW SRSNLDTEVT TAKESPEIEP LPFTLAHERC ISVVQKLVLF LLSMDFTCHA DLLLFVCKVL ARIANATRPT IHLCEIVNEP QLERLLLLLV GTDFNRGDIS WGGAWAQYSL TCMLQDILAG ELLAPVAAEA MEEGTVGDDV GATAGDSDDS LQQSSVQLLE TIDEPLTHDI TGAPPLSSLE KDKEIDLELL QDLMEVDIDP LDIDLEKDPL AAKVFKPISS TWYDYWGADY GTYNYNPYIG GLGIPVAKPP ANTEKNGSQT VSVSVSQALD ARLEVGLEQQ AELMLKMMST LEADSILQAL TNTSPTLSQS PTGTDDSLLG GLQAANQTSQ LIIQLSSVPM LNVCFNKLFS MLQVHHVQLE SLLQLWLTLS LNSSSTGNKE NGADIFLYNA NRIPVISLNQ ASITSFLTVL AWYPNTLLRT WCLVLHSLTL MTNMQLNSGS SSAIGTQEST AHLLVSDPNL IHVLVKFLSG TSPHGTNQHS PQVGPTATQA MQEFLTRLQV HLSSTCPQIF SEFLLKLIHI LSTERGAFQT GQGPLDAQVK LLEFTLEQNF EVVSVSTISA VIESVTFLVH HYITCSDKVM SRSGSDSSVG ARACFGGLFA NLIRPGDAKA VCGEMTRDQL MFDLLKLVNI LVQLPLSGNR EYSARVSVTT NTTDSVSDEE KVSGGKDGNG SSTSVQGSPA YVADLVLANQ QIMSQILSAL GLCNSSAMAM IIGASGLHLT KHENFHGGLD AISVGDGLFT ILTTLSKKAS TVHMMLQPIL TYMACGYMGR QGSLATCQLS EPLLWFILRV LDTSDALKAF HDMGGVQLIC NNMVTSTRAI VNTARSMVST IMKFLDSGPN KAVDSTLKTR ILASEPDNAE GIHNFAPLGT ITSSSPTAQP AEVLLQATPP HRRARSAAWS YIFLPEEAWC DLTIHLPAAV LLKEIHIQPH LASLATCPSS VSVEVSADGV NMLPLSTPVV TSGLTYIKIQ LVKAEVASAV CLRLHRPRDA STLGLSQIKL LGLTAFGTTS SATVNNPFLP SEDQVSKTSI GWLRLLHHCL THISDLEGMM ASAAAPTANL LQTCAALLMS PYCGMHSPNI EVVLVKIGLQ STRIGLKLID ILLRNCAASG SDPTDLNSPL LFGRLNGLSS DSTIDILYQL GTTQDPGTKD RIQALLKWVS DSARVAAMKR SGRMNYMCPN SSTVEYGLLM PSPSHLHCVA AILWHSYELL VEYDLPALLD QELFELLFNW SMSLPCNMVL KKAVDSLLCS MCHVHPNYFS LLMGWMGITP PPVQCHHRLS MTDDSKKQDL SSSLTDDSKN AQAPLALTES HLATLASSSQ SPEAIKQLLD SGLPSLLVRS LASFCFSHIS SSESIAQSID ISQDKLRRHH VPQQCNKMPI TADLVAPILR FLTEVGNSHI MKDWLGGSEV NPLWTALLFL LCHSGSTSGS HNLGAQQTSA RSASLSSAAT TGLTTQQRTA IENATVAFFL QCISCHPNNQ KLMAQVLCEL FQTSPQRGNL PTSGNISGFI RRLFLQLMLE DEKVTMFLQS PCPLYKGRIN ATSHVIQHPM YGAGHKFRTL HLPVSTTLSD VLDRVSDTPS ITAKLISEQK DDKEKKNHEE KEKVKAENGF QDNYSVVVAS GLKSQSKRAV SATPPRPPSR RGRTIPDKIG STSGAEAANK IITVPVFHLF HKLLAGQPLP AEMTLAQLLT LLYDRKLPQG YRSIDLTVKL GSRVITDPSL SKTDSYKRLH PEKDHGDLLA SCPEDEALTP GDECMDGILD ESLLETCPIQ SPLQVFAGMG GLALIAERLP MLYPEVIQQV SAPVVTSTTQ EKPKDSDQFE WVTIEQSGEL VYEAPETVAA EPPPIKSAVQ TMSPIPAHSL AAFGLFLRLP GYAEVLLKER KHAQCLLRLV LGVTDDGEGS HILQSPSANV LPTLPFHVLR SLFSTTPLTT DDGVLLRRMA LEIGALHLIL VCLSALSHHS PRVPNSSVNQ TEPQVSSSHN PTSTEEQQLY WAKGTGFGTG STASGWDVEQ ALTKQRLEEE HVTCLLQVLA SYINPVSSAV NGEAQSSHET RGQNSNALPS VLLELLSQSC LIPAMSSYLR NDSVLDMARH VPLYRALLEL LRAIASCAAM VPLLLPLSTE NGEEEEEQSE CQTSVGTLLA KMKTCVDTYT NRLRSKRENV KTGVKPDASD QEPEGLTLLV PDIQKTAEIV YAATTSLRQA NQEKKLGEYS KKAAMKPKPL SVLKSLEEKY VAVMKKLQFD TFEMVSEDED GKLGFKVNYH YMSQVKNAND ANSAARARRL AQEAVTLSTS LPLSSSSSVF VRCDEERLDI MKVLITGPAD TPYANGCFEF DVYFPQDYPS SPPLVNLETT GGHSVRFNPN LYNDGKVCLS ILNTWHGRPE EKWNPQTSSF LQVLVSVQSL ILVAEPYFNE PGYERSRGTP SGTQSSREYD GNIRQATVKW AMLEQIRNPS PCFKEVIHKH FYLKRVEIMA QCEEWIADIQ QYSSDKRVGR TMSHHAAALK RHTAQLREEL LKLPCPEGLD PDTDDAPEVC RATTGAEETL MHDQVKPSSS KELPSDFQL UniProtKB: Baculoviral IAP repeat-containing protein 6 |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 4 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK III |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均露光時間: 3.5 sec. / 平均電子線量: 49.9 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 最大 デフォーカス(補正後): 2.5 µm / 最小 デフォーカス(補正後): 1.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 105000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: OTHER |
|---|
 ムービー
ムービー コントローラー
コントローラー













 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)