[日本語] English
 万見
万見- EMDB-15603: Cryo-EM structure of human CtBP1/RAI2(303-362) delta(331-341) filament -
+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of human CtBP1/RAI2(303-362) delta(331-341) filament | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Complex / Tumor suppressor / oncogenic / filament assembly / ANTITUMOR PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報Signaling by TCF7L2 mutants / Repression of WNT target genes / animal organ development / synaptic vesicle clustering / presynaptic active zone cytoplasmic component / embryo development ending in birth or egg hatching / 酸化還元酵素; CH-OHの結合に対し酸化酵素として働く; NAD又はNADPを用いる / oxidoreductase activity, acting on the CH-OH group of donors, NAD or NADP as acceptor / lncRNA binding / white fat cell differentiation ...Signaling by TCF7L2 mutants / Repression of WNT target genes / animal organ development / synaptic vesicle clustering / presynaptic active zone cytoplasmic component / embryo development ending in birth or egg hatching / 酸化還元酵素; CH-OHの結合に対し酸化酵素として働く; NAD又はNADPを用いる / oxidoreductase activity, acting on the CH-OH group of donors, NAD or NADP as acceptor / lncRNA binding / white fat cell differentiation / synaptic vesicle endocytosis / Notch signaling pathway / transcription repressor complex / SUMOylation of transcription cofactors / Deactivation of the beta-catenin transactivating complex / viral genome replication / transcription coregulator binding / transcription corepressor binding / GABA-ergic synapse / NAD binding / transcription corepressor activity / DNA-binding transcription factor binding / RNA polymerase II-specific DNA-binding transcription factor binding / transcription coactivator activity / protein phosphorylation / regulation of cell cycle / protein domain specific binding / negative regulation of cell population proliferation / negative regulation of DNA-templated transcription / chromatin binding / regulation of transcription by RNA polymerase II / glutamatergic synapse / negative regulation of transcription by RNA polymerase II / nucleoplasm / identical protein binding / nucleus 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
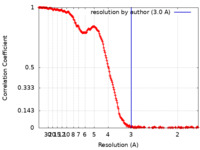
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.0 Å | |||||||||
 データ登録者 データ登録者 | Goradia N / Mullapudi E / Wilmanns M | |||||||||
| 資金援助 |  ドイツ, 1件 ドイツ, 1件
| |||||||||
 引用 引用 |  ジャーナル: To Be Published ジャーナル: To Be Publishedタイトル: Cryo-EM structure of human CtBP1/RAI2(303-362) delta(331-341) filament 著者: Goradia N / Mullapudi E / Wilmanns M | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_15603.map.gz emd_15603.map.gz | 267.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-15603-v30.xml emd-15603-v30.xml emd-15603.xml emd-15603.xml | 16.8 KB 16.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_15603_fsc.xml emd_15603_fsc.xml | 23.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_15603.png emd_15603.png | 89.8 KB | ||
| その他 |  emd_15603_half_map_1.map.gz emd_15603_half_map_1.map.gz emd_15603_half_map_2.map.gz emd_15603_half_map_2.map.gz | 496.9 MB 496.9 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-15603 http://ftp.pdbj.org/pub/emdb/structures/EMD-15603 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-15603 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-15603 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_15603_validation.pdf.gz emd_15603_validation.pdf.gz | 945.2 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_15603_full_validation.pdf.gz emd_15603_full_validation.pdf.gz | 944.8 KB | 表示 | |
| XML形式データ |  emd_15603_validation.xml.gz emd_15603_validation.xml.gz | 26.4 KB | 表示 | |
| CIF形式データ |  emd_15603_validation.cif.gz emd_15603_validation.cif.gz | 35.3 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15603 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15603 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15603 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15603 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8ariMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_15603.map.gz / 形式: CCP4 / 大きさ: 536.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_15603.map.gz / 形式: CCP4 / 大きさ: 536.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.87 Å | ||||||||||||||||||||||||||||||||||||
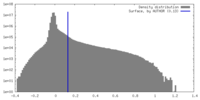
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #1
| ファイル | emd_15603_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
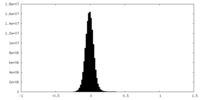
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_15603_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
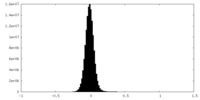
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Complex of hCtBP1 with hRAI2
| 全体 | 名称: Complex of hCtBP1 with hRAI2 |
|---|---|
| 要素 |
|
-超分子 #1: Complex of hCtBP1 with hRAI2
| 超分子 | 名称: Complex of hCtBP1 with hRAI2 / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#2 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 1.0 MDa |
-分子 #1: C-terminal-binding protein 1
| 分子 | 名称: C-terminal-binding protein 1 / タイプ: protein_or_peptide / ID: 1 / コピー数: 24 / 光学異性体: LEVO EC番号: 酸化還元酵素; CH-OHの結合に対し酸化酵素として働く; NAD又はNADPを用いる |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 49.525348 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: HHHHHHSAGL EVLFQGPMGS SHLLNKGLPL GVRPPIMNGP LHPRPLVALL DGRDCTVEMP ILKDVATVAF CDAQSTQEIH EKVLNEAVG ALMYHTITLT REDLEKFKAL RIIVRIGSGF DNIDIKSAGD LGIAVCNVPA ASVEETADST LCHILNLYRR A TWLHQALR ...文字列: HHHHHHSAGL EVLFQGPMGS SHLLNKGLPL GVRPPIMNGP LHPRPLVALL DGRDCTVEMP ILKDVATVAF CDAQSTQEIH EKVLNEAVG ALMYHTITLT REDLEKFKAL RIIVRIGSGF DNIDIKSAGD LGIAVCNVPA ASVEETADST LCHILNLYRR A TWLHQALR EGTRVQSVEQ IREVASGAAR IRGETLGIIG LGRVGQAVAL RAKAFGFNVL FYDPYLSDGV ERALGLQRVS TL QDLLFHS DCVTLHCGLN EHNHHLINDF TVKQMRQGAF LVNTARGGLV DEKALAQALK EGRIRGAALD VHESEPFSFS QGP LKDAPN LICTPHAAWY SEQASIEMRE EAAREIRRAI TGRIPDSLKN CVNKDHLTAA THWASMDPAV VHPELNGAAY RYPP GVVGV APTGIPAAVE GIVPSAMSLS HGLPPVAHPP HAPSPGQTVK PEADRDHASD QL UniProtKB: C-terminal-binding protein 1 |
-分子 #2: Retinoic acid-induced protein 2
| 分子 | 名称: Retinoic acid-induced protein 2 / タイプ: protein_or_peptide / ID: 2 / コピー数: 10 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 14.252756 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: HHHHHHPMKQ YKLILNGKTL KGETTTEAVD AATAEKVFKQ YANDNGVDGE WTYDDATKTF TVTEGSGSGS ENLYFQGAMD SRHTVIKMG SENEALDLSM KSVPWLKAGA LDLSVAAHRK SEPPPETLYD UniProtKB: Retinoic acid-induced protein 2 |
-分子 #3: NICOTINAMIDE-ADENINE-DINUCLEOTIDE
| 分子 | 名称: NICOTINAMIDE-ADENINE-DINUCLEOTIDE / タイプ: ligand / ID: 3 / コピー数: 24 / 式: NAD |
|---|---|
| 分子量 | 理論値: 663.425 Da |
| Chemical component information |  ChemComp-NAD: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | filament |
- 試料調製
試料調製
| 緩衝液 | pH: 7.2 |
|---|---|
| グリッド | モデル: Quantifoil R2/2 / 材質: COPPER / メッシュ: 200 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 60 sec. |
| 凍結 | 凍結剤: ETHANE-PROPANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 42.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.75 µm / 最小 デフォーカス(公称値): 0.75 µm / 倍率(公称値): 105000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー



 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)