+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Symmetric hexamer of vaccinia virus DNA helicase D5 residues 323-785 | ||||||||||||||||||
 マップデータ マップデータ | Flipped to correct hand, masked map from Relion PostRefine | ||||||||||||||||||
 試料 試料 |
| ||||||||||||||||||
 キーワード キーワード | DNA helicase / D5_N domain / DUF5906 domain / Pox_D5 domain / SF3 helicase / viral protein | ||||||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報helicase activity / 加水分解酵素; 酸無水物に作用; 酸無水物に作用・細胞または細胞小器官の運動に関与 / host cell cytoplasm / hydrolase activity / ATP binding 類似検索 - 分子機能 | ||||||||||||||||||
| 生物種 |  Vaccinia virus Copenhagen (ウイルス) Vaccinia virus Copenhagen (ウイルス) | ||||||||||||||||||
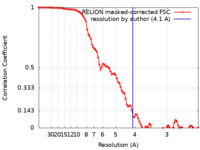
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.1 Å | ||||||||||||||||||
 データ登録者 データ登録者 | Burmeister WP / Hutin S / Ling WL / Grimm C / Schoehn G | ||||||||||||||||||
| 資金援助 |  フランス, 5件 フランス, 5件
| ||||||||||||||||||
 引用 引用 |  ジャーナル: Viruses / 年: 2022 ジャーナル: Viruses / 年: 2022タイトル: The Vaccinia Virus DNA Helicase Structure from Combined Single-Particle Cryo-Electron Microscopy and AlphaFold2 Prediction. 著者: Stephanie Hutin / Wai Li Ling / Nicolas Tarbouriech / Guy Schoehn / Clemens Grimm / Utz Fischer / Wim P Burmeister /   要旨: Poxviruses are large DNA viruses with a linear double-stranded DNA genome circularized at the extremities. The helicase-primase D5, composed of six identical 90 kDa subunits, is required for DNA ...Poxviruses are large DNA viruses with a linear double-stranded DNA genome circularized at the extremities. The helicase-primase D5, composed of six identical 90 kDa subunits, is required for DNA replication. D5 consists of a primase fragment flexibly attached to the hexameric C-terminal polypeptide (res. 323-785) with confirmed nucleotide hydrolase and DNA-binding activity but an elusive helicase activity. We determined its structure by single-particle cryo-electron microscopy. It displays an AAA+ helicase core flanked by N- and C-terminal domains. Model building was greatly helped by the predicted structure of D5 using AlphaFold2. The 3.9 Å structure of the N-terminal domain forms a well-defined tight ring while the resolution decreases towards the C-terminus, still allowing the fit of the predicted structure. The N-terminal domain is partially present in papillomavirus E1 and polyomavirus LTA helicases, as well as in a bacteriophage NrS-1 helicase domain, which is also closely related to the AAA+ helicase domain of D5. Using the Pfam domain database, a D5_N domain followed by DUF5906 and Pox_D5 domains could be assigned to the cryo-EM structure, providing the first 3D structures for D5_N and Pox_D5 domains. The same domain organization has been identified in a family of putative helicases from large DNA viruses, bacteriophages, and selfish DNA elements. | ||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_15574.map.gz emd_15574.map.gz | 5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-15574-v30.xml emd-15574-v30.xml emd-15574.xml emd-15574.xml | 19.8 KB 19.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_15574_fsc.xml emd_15574_fsc.xml | 9.1 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_15574.png emd_15574.png | 238.8 KB | ||
| Filedesc metadata |  emd-15574.cif.gz emd-15574.cif.gz | 6.3 KB | ||
| その他 |  emd_15574_half_map_1.map.gz emd_15574_half_map_1.map.gz emd_15574_half_map_2.map.gz emd_15574_half_map_2.map.gz | 48.7 MB 48.6 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-15574 http://ftp.pdbj.org/pub/emdb/structures/EMD-15574 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-15574 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-15574 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_15574_validation.pdf.gz emd_15574_validation.pdf.gz | 725 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_15574_full_validation.pdf.gz emd_15574_full_validation.pdf.gz | 724.6 KB | 表示 | |
| XML形式データ |  emd_15574_validation.xml.gz emd_15574_validation.xml.gz | 16.2 KB | 表示 | |
| CIF形式データ |  emd_15574_validation.cif.gz emd_15574_validation.cif.gz | 21.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15574 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15574 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15574 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15574 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8aplMC  8apmC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_15574.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_15574.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Flipped to correct hand, masked map from Relion PostRefine | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.21 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: Flipped to correct hand, half map from Relion...
| ファイル | emd_15574_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Flipped to correct hand, half map from Relion Refine3D after polishing. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Flipped to correct hand, half map from Relion Refine3D after polishin
| ファイル | emd_15574_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Flipped to correct hand, half map from Relion Refine3D after polishin | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : D5 C-terminal fragment res. 323 - res. 785
| 全体 | 名称: D5 C-terminal fragment res. 323 - res. 785 |
|---|---|
| 要素 |
|
-超分子 #1: D5 C-terminal fragment res. 323 - res. 785
| 超分子 | 名称: D5 C-terminal fragment res. 323 - res. 785 / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all / 詳細: construct |
|---|---|
| 由来(天然) | 生物種:  Vaccinia virus Copenhagen (ウイルス) Vaccinia virus Copenhagen (ウイルス) |
| 分子量 | 理論値: 321 KDa |
-分子 #1: Primase D5
| 分子 | 名称: Primase D5 / タイプ: protein_or_peptide / ID: 1 / コピー数: 6 / 光学異性体: LEVO EC番号: 加水分解酵素; 酸無水物に作用; 酸無水物に作用・細胞または細胞小器官の運動に関与 |
|---|---|
| 由来(天然) | 生物種:  Vaccinia virus Copenhagen (ウイルス) / 株: Copenhagen Vaccinia virus Copenhagen (ウイルス) / 株: Copenhagen |
| 分子量 | 理論値: 53.495285 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: AMGNKLFNIA QRILDTNSVL LTERGDYIVW INNSWKFNSE EPLITKLILS IRHQLPKEYS SELLCPRKRK TVEANIRDML VDSVETDTY PDKLPFKNGV LDLVDGMFYS GDDAKKYTCT VSTGFKFDDT KFVEDSPEME ELMNIINDIQ PLTDENKKNR E LYEKTLSS ...文字列: AMGNKLFNIA QRILDTNSVL LTERGDYIVW INNSWKFNSE EPLITKLILS IRHQLPKEYS SELLCPRKRK TVEANIRDML VDSVETDTY PDKLPFKNGV LDLVDGMFYS GDDAKKYTCT VSTGFKFDDT KFVEDSPEME ELMNIINDIQ PLTDENKKNR E LYEKTLSS CLCGATKGCL TFFFGETATG KSTTKRLLKS AIGDLFVETG QTILTDVLDK GPNPFIANMH LKRSVFCSEL PD FACSGSK KIRSDNIKKL TEPCVIGRPC FSNKINNRNH ATIIIDTNYK PVFDRIDNAL MRRIAVVRFR THFSQPSGRE AAE NNDAYD KVKLLDEGLD GKIQNNRYRF AFLYLLVKWY KKYHVPIMKL YPTPEEIPDF AFYLKIGTLL VSSSVKHIPL MTDL SKKGY ILYDNVVTLP LTTFQQKISK YFNSRLFGHD IESFINRHKK FANVSDEYLQ YIFIEDISSP UniProtKB: Uncoating factor OPG117 |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.24 mg/mL | |||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7 構成要素:
| |||||||||||||||||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 雰囲気: AIR | |||||||||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 293 K / 装置: FEI VITROBOT MARK IV | |||||||||||||||||||||
| 詳細 | The sample contained also a dsDNA oligomer in a close to stoechiometric concentration not visible in this structure. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI POLARA 300 |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / デジタル化 - 画像ごとのフレーム数: 1-40 / 撮影したグリッド数: 1 / 実像数: 830 / 平均電子線量: 1.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 最大 デフォーカス(補正後): 3.0 µm / 最小 デフォーカス(補正後): 1.5 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 1.5 µm |
| 試料ステージ | ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Tecnai Polara / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: OTHER / 温度因子: 152 |
|---|---|
| 得られたモデル |  PDB-8apl: |
 ムービー
ムービー コントローラー
コントローラー







 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)