+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of human tankyrase 2 SAM-PARP filament (G1032W mutant) | |||||||||||||||
 マップデータ マップデータ | ||||||||||||||||
 試料 試料 |
| |||||||||||||||
 キーワード キーワード | Poly-ADP-ribosyltransferase / Enzyme / Polymer / SIGNALING PROTEIN | |||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報XAV939 stabilizes AXIN / positive regulation of telomere capping / NAD+ ADP-ribosyltransferase / negative regulation of telomere maintenance via telomere lengthening / protein auto-ADP-ribosylation / protein localization to chromosome, telomeric region / NAD+-protein-aspartate ADP-ribosyltransferase activity / protein poly-ADP-ribosylation / NAD+-protein-glutamate ADP-ribosyltransferase activity / NAD+-protein mono-ADP-ribosyltransferase activity ...XAV939 stabilizes AXIN / positive regulation of telomere capping / NAD+ ADP-ribosyltransferase / negative regulation of telomere maintenance via telomere lengthening / protein auto-ADP-ribosylation / protein localization to chromosome, telomeric region / NAD+-protein-aspartate ADP-ribosyltransferase activity / protein poly-ADP-ribosylation / NAD+-protein-glutamate ADP-ribosyltransferase activity / NAD+-protein mono-ADP-ribosyltransferase activity / pericentriolar material / 転移酵素; グリコシル基を移すもの; 五炭糖残基を移すもの / NAD+ poly-ADP-ribosyltransferase activity / positive regulation of telomere maintenance via telomerase / nucleotidyltransferase activity / TCF dependent signaling in response to WNT / Degradation of AXIN / Regulation of PTEN stability and activity / Wnt signaling pathway / protein polyubiquitination / positive regulation of canonical Wnt signaling pathway / nuclear envelope / chromosome, telomeric region / Ub-specific processing proteases / Golgi membrane / perinuclear region of cytoplasm / enzyme binding / metal ion binding / nucleus / cytoplasm / cytosol 類似検索 - 分子機能 | |||||||||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||||||||
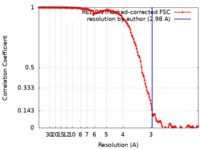
| 手法 | らせん対称体再構成法 / クライオ電子顕微鏡法 / 解像度: 2.98 Å | |||||||||||||||
 データ登録者 データ登録者 | Mariotti L / Inian O / Desfosses A / Beuron F / Morris EP / Guettler S | |||||||||||||||
| 資金援助 |  英国, 4件 英国, 4件
| |||||||||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2022 ジャーナル: Nature / 年: 2022タイトル: Structural basis of tankyrase activation by polymerization. 著者: Nisha Pillay / Laura Mariotti / Mariola Zaleska / Oviya Inian / Matthew Jessop / Sam Hibbs / Ambroise Desfosses / Paul C R Hopkins / Catherine M Templeton / Fabienne Beuron / Edward P Morris ...著者: Nisha Pillay / Laura Mariotti / Mariola Zaleska / Oviya Inian / Matthew Jessop / Sam Hibbs / Ambroise Desfosses / Paul C R Hopkins / Catherine M Templeton / Fabienne Beuron / Edward P Morris / Sebastian Guettler /   要旨: The poly-ADP-ribosyltransferase tankyrase (TNKS, TNKS2) controls a wide range of disease-relevant cellular processes, including WNT-β-catenin signalling, telomere length maintenance, Hippo ...The poly-ADP-ribosyltransferase tankyrase (TNKS, TNKS2) controls a wide range of disease-relevant cellular processes, including WNT-β-catenin signalling, telomere length maintenance, Hippo signalling, DNA damage repair and glucose homeostasis. This has incentivized the development of tankyrase inhibitors. Notwithstanding, our knowledge of the mechanisms that control tankyrase activity has remained limited. Both catalytic and non-catalytic functions of tankyrase depend on its filamentous polymerization. Here we report the cryo-electron microscopy reconstruction of a filament formed by a minimal active unit of tankyrase, comprising the polymerizing sterile alpha motif (SAM) domain and its adjacent catalytic domain. The SAM domain forms a novel antiparallel double helix, positioning the protruding catalytic domains for recurring head-to-head and tail-to-tail interactions. The head interactions are highly conserved among tankyrases and induce an allosteric switch in the active site within the catalytic domain to promote catalysis. Although the tail interactions have a limited effect on catalysis, they are essential to tankyrase function in WNT-β-catenin signalling. This work reveals a novel SAM domain polymerization mode, illustrates how supramolecular assembly controls catalytic and non-catalytic functions, provides important structural insights into the regulation of a non-DNA-dependent poly-ADP-ribosyltransferase and will guide future efforts to modulate tankyrase and decipher its contribution to disease mechanisms. | |||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_15520.map.gz emd_15520.map.gz | 105 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-15520-v30.xml emd-15520-v30.xml emd-15520.xml emd-15520.xml | 15.3 KB 15.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_15520_fsc.xml emd_15520_fsc.xml | 14.1 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_15520.png emd_15520.png | 193.3 KB | ||
| Filedesc metadata |  emd-15520.cif.gz emd-15520.cif.gz | 6.3 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-15520 http://ftp.pdbj.org/pub/emdb/structures/EMD-15520 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-15520 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-15520 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_15520_validation.pdf.gz emd_15520_validation.pdf.gz | 423.5 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_15520_full_validation.pdf.gz emd_15520_full_validation.pdf.gz | 423 KB | 表示 | |
| XML形式データ |  emd_15520_validation.xml.gz emd_15520_validation.xml.gz | 14.5 KB | 表示 | |
| CIF形式データ |  emd_15520_validation.cif.gz emd_15520_validation.cif.gz | 19.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15520 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15520 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15520 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15520 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8alyMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_15520.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_15520.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.06 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : TNKS2 SAM-PARP (867-1162) filament
| 全体 | 名称: TNKS2 SAM-PARP (867-1162) filament |
|---|---|
| 要素 |
|
-超分子 #1: TNKS2 SAM-PARP (867-1162) filament
| 超分子 | 名称: TNKS2 SAM-PARP (867-1162) filament / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 詳細: double-helical filament of human TNKS2 SAM-PARP G1032W, residues 867-1162, N-terminal vector-derived SNA tripeptide, 20 protomers in refined structure |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 34 kDa/nm |
-分子 #1: Poly [ADP-ribose] polymerase tankyrase-2
| 分子 | 名称: Poly [ADP-ribose] polymerase tankyrase-2 / タイプ: protein_or_peptide / ID: 1 / コピー数: 20 / 光学異性体: LEVO / EC番号: NAD+ ADP-ribosyltransferase |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 34.077668 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: SNAEKKEVPG VDFSITQFVR NLGLEHLMDI FEREQITLDV LVEMGHKELK EIGINAYGHR HKLIKGVERL ISGQQGLNPY LTLNTSGSG TILIDLSPDD KEFQSVEEEM QSTVREHRDG GHAGGIFNRY NILKIQKVCN KKLWERYTHR RKEVSEENHN H ANERMLFH ...文字列: SNAEKKEVPG VDFSITQFVR NLGLEHLMDI FEREQITLDV LVEMGHKELK EIGINAYGHR HKLIKGVERL ISGQQGLNPY LTLNTSGSG TILIDLSPDD KEFQSVEEEM QSTVREHRDG GHAGGIFNRY NILKIQKVCN KKLWERYTHR RKEVSEENHN H ANERMLFH WSPFVNAIIH KGFDERHAYI GGMFGAGIYF AENSSKSNQY VYGIGGGTGC PVHKDRSCYI CHRQLLFCRV TL GKSFLQF SAMKMAHSPP GHHSVTGRPS VNGLALAEYV IYRGEQAYPE YLITYQIMRP EG UniProtKB: Poly [ADP-ribose] polymerase tankyrase-2 |
-分子 #2: ZINC ION
| 分子 | 名称: ZINC ION / タイプ: ligand / ID: 2 / コピー数: 20 / 式: ZN |
|---|---|
| 分子量 | 理論値: 65.409 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | らせん対称体再構成法 |
| 試料の集合状態 | filament |
- 試料調製
試料調製
| 濃度 | 0.86 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
詳細: After brief incubation on the grid, the sample was washed 10 times with water to gradually lower the salt concentration and improve sample contrast. | ||||||||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 400 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: CONTINUOUS / 前処理 - タイプ: GLOW DISCHARGE | ||||||||||||
| 凍結 | 凍結剤: ETHANE / 装置: FEI VITROBOT MARK IV | ||||||||||||
| 詳細 | The PARP domain is inactivated by a G1032W mutation. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS KRIOS |
|---|---|
| 温度 | 最低: 80.0 K / 最高: 80.0 K |
| 撮影 | #0 - Image recording ID: 1 #0 - フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) #0 - 検出モード: INTEGRATING / #0 - 平均電子線量: 40.0 e/Å2 / #1 - Image recording ID: 2 #1 - フィルム・検出器のモデル: GATAN K3 (6k x 4k) #1 - 平均電子線量: 40.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.5 µm / 最小 デフォーカス(公称値): 1.2 µm / 倍率(公称値): 81000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル |
| ||||||
|---|---|---|---|---|---|---|---|
| 精密化 | 空間: REAL / プロトコル: FLEXIBLE FIT / 温度因子: 7.41 / 当てはまり具合の基準: FSC | ||||||
| 得られたモデル |  PDB-8aly: |
 ムービー
ムービー コントローラー
コントローラー








 X (Sec.)
X (Sec.) Y (Row.)
Y (Row.) Z (Col.)
Z (Col.)