+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-1542 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Three-dimensional structure of Pyrococcus furiosus A1Ao ATP synthase | |||||||||
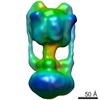
 マップデータ マップデータ | volume of Pyrococcus furiosus A-type ATP synthase | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | ATP synthase / A1Ao ATP synthase | |||||||||
| 生物種 |   Pyrococcus furiosus (古細菌) Pyrococcus furiosus (古細菌) | |||||||||
| 手法 | 単粒子再構成法 / ネガティブ染色法 / 解像度: 23.0 Å | |||||||||
 データ登録者 データ登録者 | Vonck J / Pisa KY / Morgner N / Brutschy B / Mueller V | |||||||||
 引用 引用 |  ジャーナル: J Biol Chem / 年: 2009 ジャーナル: J Biol Chem / 年: 2009タイトル: Three-dimensional structure of A1A0 ATP synthase from the hyperthermophilic archaeon Pyrococcus furiosus by electron microscopy. 著者: Janet Vonck / Kim Y Pisa / Nina Morgner / Bernhard Brutschy / Volker Müller /  要旨: The archaeal ATP synthase is a multisubunit complex that consists of a catalytic A(1) part and a transmembrane, ion translocation domain A(0). The A(1)A(0) complex from the hyperthermophile ...The archaeal ATP synthase is a multisubunit complex that consists of a catalytic A(1) part and a transmembrane, ion translocation domain A(0). The A(1)A(0) complex from the hyperthermophile Pyrococcus furiosus was isolated. Mass analysis of the complex by laser-induced liquid bead ion desorption (LILBID) indicated a size of 730 +/- 10 kDa. A three-dimensional map was generated by electron microscopy from negatively stained images. The map at a resolution of 2.3 nm shows the A(1) and A(0) domain, connected by a central stalk and two peripheral stalks, one of which is connected to A(0), and both connected to A(1) via prominent knobs. X-ray structures of subunits from related proteins were fitted to the map. On the basis of the fitting and the LILBID analysis, a structural model is presented with the stoichiometry A(3)B(3)CDE(2)FH(2)ac(10). | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_1542.map.gz emd_1542.map.gz | 516.8 KB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-1542-v30.xml emd-1542-v30.xml emd-1542.xml emd-1542.xml | 9.6 KB 9.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  1542.gif 1542.gif | 19.3 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-1542 http://ftp.pdbj.org/pub/emdb/structures/EMD-1542 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1542 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1542 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_1542_validation.pdf.gz emd_1542_validation.pdf.gz | 198.5 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_1542_full_validation.pdf.gz emd_1542_full_validation.pdf.gz | 197.6 KB | 表示 | |
| XML形式データ |  emd_1542_validation.xml.gz emd_1542_validation.xml.gz | 5.3 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1542 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1542 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1542 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1542 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_1542.map.gz / 形式: CCP4 / 大きさ: 1.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_1542.map.gz / 形式: CCP4 / 大きさ: 1.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | volume of Pyrococcus furiosus A-type ATP synthase | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 4.77 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : A1Ao ATP synthase from Pyrococcus furiosus
| 全体 | 名称: A1Ao ATP synthase from Pyrococcus furiosus |
|---|---|
| 要素 |
|
-超分子 #1000: A1Ao ATP synthase from Pyrococcus furiosus
| 超分子 | 名称: A1Ao ATP synthase from Pyrococcus furiosus / タイプ: sample / ID: 1000 / 集合状態: nine different subunits / Number unique components: 9 |
|---|---|
| 分子量 | 実験値: 738 KDa / 理論値: 738 KDa / 手法: LILBID mass spectroscopy |
-分子 #1: A1Ao ATP synthase
| 分子 | 名称: A1Ao ATP synthase / タイプ: protein_or_peptide / ID: 1 / Name.synonym: ATP synthase / 集合状態: heteromultimer / 組換発現: No |
|---|---|
| 由来(天然) | 生物種:   Pyrococcus furiosus (古細菌) / 細胞: archaeon / 細胞中の位置: plasma membrane Pyrococcus furiosus (古細菌) / 細胞: archaeon / 細胞中の位置: plasma membrane |
| 分子量 | 実験値: 738 KDa / 理論値: 738 KDa |
-分子 #2: A1Ao ATP synthase
| 分子 | 名称: A1Ao ATP synthase / タイプ: protein_or_peptide / ID: 2 / Name.synonym: ATP synthase / 組換発現: No |
|---|---|
| 由来(天然) | 生物種:   Pyrococcus furiosus (古細菌) Pyrococcus furiosus (古細菌) |
| 分子量 | 実験値: 738 KDa / 理論値: 738 KDa |
-実験情報
-構造解析
| 手法 | ネガティブ染色法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 詳細: 50 mM this/HCl, 5 mM MgCl2, 10% glycerol, 50 mM KCl, 0.1% Triton-X100, 0.1 mM PMSF |
| 染色 | タイプ: NEGATIVE 詳細: a drop of 1% (w/v) uranyl acetate was added to a carbon-coated grid with absorbed protein and blotted after 30 s. |
| グリッド | 詳細: 400 mesh copper grid |
| 凍結 | 凍結剤: NONE / 装置: OTHER |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI/PHILIPS CM120T |
|---|---|
| アライメント法 | Legacy - 非点収差: objective lens astigmatism was corrected at 160,000 times magnification |
| 撮影 | カテゴリ: FILM / フィルム・検出器のモデル: KODAK SO-163 FILM / デジタル化 - スキャナー: ZEISS SCAI / デジタル化 - サンプリング間隔: 7 µm / 実像数: 51 / 平均電子線量: 20 e/Å2 / ビット/ピクセル: 8 |
| 電子線 | 加速電圧: 120 kV / 電子線源: LAB6 |
| 電子光学系 | 倍率(補正後): 44000 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2 mm / 最大 デフォーカス(公称値): 0.4 µm / 最小 デフォーカス(公称値): 0.4 µm / 倍率(公称値): 45000 |
| 試料ステージ | 試料ホルダー: eucentric / 試料ホルダーモデル: OTHER |
- 画像解析
画像解析
| 最終 再構成 | 想定した対称性 - 点群: C1 (非対称) / 解像度のタイプ: BY AUTHOR / 解像度: 23.0 Å / 解像度の算出法: FSC 0.5 CUT-OFF / ソフトウェア - 名称: EMAN / 使用した粒子像数: 12912 |
|---|---|
| 最終 2次元分類 | クラス数: 392 |
 ムービー
ムービー コントローラー
コントローラー



 UCSF Chimera
UCSF Chimera







 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)





















