+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Architecture of the ESCPE-1 membrane coat | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | sorting nexins / PX domain / BAR domain / endosomes / retrograde transport / endocytic recycling / cargo recognition / protein coat / membrane recruitment / membrane deformation / membrane tubules. / TRANSPORT PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報retromer, tubulation complex / lamellipodium morphogenesis / epidermal growth factor catabolic process / pinocytosis / cytoplasmic side of early endosome membrane / leptin receptor binding / tubular endosome / macropinocytic cup / clathrin coat / early endosome to Golgi transport ...retromer, tubulation complex / lamellipodium morphogenesis / epidermal growth factor catabolic process / pinocytosis / cytoplasmic side of early endosome membrane / leptin receptor binding / tubular endosome / macropinocytic cup / clathrin coat / early endosome to Golgi transport / Retrograde transport at the Trans-Golgi-Network / response to tetrachloromethane / insulin-like growth factor receptor activity / retromer complex binding / insulin-like growth factor binding / phosphatidylinositol-5-phosphate binding / transferrin receptor binding / retromer complex / insulin-like growth factor II binding / trans-Golgi network transport vesicle / host-mediated activation of viral process / retinoic acid binding / phosphatidylinositol-4-phosphate binding / retrograde transport, endosome to Golgi / phosphatidylinositol-3,5-bisphosphate binding / epidermal growth factor receptor binding / lysosomal transport / Golgi Associated Vesicle Biogenesis / nuclear envelope lumen / D-mannose binding / brush border / dynactin binding / endocytic vesicle / G-protein alpha-subunit binding / animal organ regeneration / regulation of macroautophagy / positive regulation of insulin receptor signaling pathway / response to retinoic acid / D1 dopamine receptor binding / phagocytic cup / transport vesicle / negative regulation of blood pressure / ruffle / phosphatidylinositol binding / receptor-mediated endocytosis / secretory granule membrane / trans-Golgi network membrane / post-embryonic development / insulin receptor binding / intracellular protein transport / phosphoprotein binding / trans-Golgi network / clathrin-coated endocytic vesicle membrane / liver development / receptor internalization / cytoplasmic side of plasma membrane / positive regulation of protein catabolic process / late endosome / Cargo recognition for clathrin-mediated endocytosis / signaling receptor activity / lamellipodium / Clathrin-mediated endocytosis / early endosome membrane / spermatogenesis / vesicle / early endosome / lysosome / endosome / endosome membrane / positive regulation of apoptotic process / cadherin binding / G protein-coupled receptor signaling pathway / protein heterodimerization activity / Golgi membrane / focal adhesion / intracellular membrane-bounded organelle / Neutrophil degranulation / positive regulation of DNA-templated transcription / perinuclear region of cytoplasm / enzyme binding / cell surface / Golgi apparatus / signal transduction / protein homodimerization activity / protein-containing complex / extracellular exosome / identical protein binding / membrane / plasma membrane / cytoplasm / cytosol 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
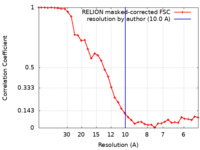
| 手法 | サブトモグラム平均法 / クライオ電子顕微鏡法 / 解像度: 10.0 Å | |||||||||
 データ登録者 データ登録者 | Lopez-Robles C / Scaramuzza S / Astorga-Simon E / Ishida M / Williamsom CD / Banos-Mateos S / Gil-Carton D / Romero M / Vidaurrazaga A / Fernandez-Recio J ...Lopez-Robles C / Scaramuzza S / Astorga-Simon E / Ishida M / Williamsom CD / Banos-Mateos S / Gil-Carton D / Romero M / Vidaurrazaga A / Fernandez-Recio J / Rojas AL / Bonifacino JS / Castano-Diez D / Hierro A | |||||||||
| 資金援助 |  スペイン, 1件 スペイン, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Struct Mol Biol / 年: 2023 ジャーナル: Nat Struct Mol Biol / 年: 2023タイトル: Architecture of the ESCPE-1 membrane coat. 著者: Carlos Lopez-Robles / Stefano Scaramuzza / Elsa N Astorga-Simon / Morié Ishida / Chad D Williamson / Soledad Baños-Mateos / David Gil-Carton / Miguel Romero-Durana / Ander Vidaurrazaga / ...著者: Carlos Lopez-Robles / Stefano Scaramuzza / Elsa N Astorga-Simon / Morié Ishida / Chad D Williamson / Soledad Baños-Mateos / David Gil-Carton / Miguel Romero-Durana / Ander Vidaurrazaga / Juan Fernandez-Recio / Adriana L Rojas / Juan S Bonifacino / Daniel Castaño-Díez / Aitor Hierro /    要旨: Recycling of membrane proteins enables the reuse of receptors, ion channels and transporters. A key component of the recycling machinery is the endosomal sorting complex for promoting exit 1 (ESCPE-1) ...Recycling of membrane proteins enables the reuse of receptors, ion channels and transporters. A key component of the recycling machinery is the endosomal sorting complex for promoting exit 1 (ESCPE-1), which rescues transmembrane proteins from the endolysosomal pathway for transport to the trans-Golgi network and the plasma membrane. This rescue entails the formation of recycling tubules through ESCPE-1 recruitment, cargo capture, coat assembly and membrane sculpting by mechanisms that remain largely unknown. Herein, we show that ESCPE-1 has a single-layer coat organization and suggest how synergistic interactions between ESCPE-1 protomers, phosphoinositides and cargo molecules result in a global arrangement of amphipathic helices to drive tubule formation. Our results thus define a key process of tubule-based endosomal sorting. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_15413.map.gz emd_15413.map.gz | 3.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-15413-v30.xml emd-15413-v30.xml emd-15413.xml emd-15413.xml | 18.2 KB 18.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_15413_fsc.xml emd_15413_fsc.xml | 3.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_15413.png emd_15413.png | 161.3 KB | ||
| Filedesc metadata |  emd-15413.cif.gz emd-15413.cif.gz | 6.5 KB | ||
| その他 |  emd_15413_half_map_1.map.gz emd_15413_half_map_1.map.gz emd_15413_half_map_2.map.gz emd_15413_half_map_2.map.gz | 3.1 MB 3.1 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-15413 http://ftp.pdbj.org/pub/emdb/structures/EMD-15413 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-15413 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-15413 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_15413_validation.pdf.gz emd_15413_validation.pdf.gz | 764.1 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_15413_full_validation.pdf.gz emd_15413_full_validation.pdf.gz | 763.6 KB | 表示 | |
| XML形式データ |  emd_15413_validation.xml.gz emd_15413_validation.xml.gz | 8.9 KB | 表示 | |
| CIF形式データ |  emd_15413_validation.cif.gz emd_15413_validation.cif.gz | 11.7 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15413 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15413 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15413 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15413 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8afzMC  8a1gC  8abqC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_15413.map.gz / 形式: CCP4 / 大きさ: 3.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_15413.map.gz / 形式: CCP4 / 大きさ: 3.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 2.73 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #2
| ファイル | emd_15413_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_15413_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Endosomal Sorting Complex for Promoting Exit 1 (ESCPE-1)
| 全体 | 名称: Endosomal Sorting Complex for Promoting Exit 1 (ESCPE-1) |
|---|---|
| 要素 |
|
-超分子 #1: Endosomal Sorting Complex for Promoting Exit 1 (ESCPE-1)
| 超分子 | 名称: Endosomal Sorting Complex for Promoting Exit 1 (ESCPE-1) タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 110 KDa |
-分子 #1: Sorting nexin-1
| 分子 | 名称: Sorting nexin-1 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 59.14434 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MASGGGGCSA SERLPPPFPG LEPESEGAAG GSEPEAGDSD TEGEDIFTGA AVVSKHQSPK ITTSLLPINN GSKENGIHEE QDQEPQDLF ADATVELSLD STQNNQKKVL AKTLISLPPQ EATNSSKPQP TYEELEEEEQ EDQFDLTVGI TDPEKIGDGM N AYVAYKVT ...文字列: MASGGGGCSA SERLPPPFPG LEPESEGAAG GSEPEAGDSD TEGEDIFTGA AVVSKHQSPK ITTSLLPINN GSKENGIHEE QDQEPQDLF ADATVELSLD STQNNQKKVL AKTLISLPPQ EATNSSKPQP TYEELEEEEQ EDQFDLTVGI TDPEKIGDGM N AYVAYKVT TQTSLPLFRS KQFAVKRRFS DFLGLYEKLS EKHSQNGFIV PPPPEKSLIG MTKVKVGKED SSSAEFLEKR RA ALERYLQ RIVNHPTMLQ DPDVREFLEK EELPRAVGTQ TLSGAGLLKM FNKATDAVSK MTIKMNESDI WFEEKLQEVE CEE QRLRKL HAVVETLVNH RKELALNTAQ FAKSLAMLGS SEDNTALSRA LSQLAEVEEK IEQLHQEQAN NDFFLLAELL SDYI RLLAI VRAAFDQRMK TWQRWQDAQA TLQKKREAEA RLLWANKPDK LQQAKDEILE WESRVTQYER DFERISTVVR KEVIR FEKE KSKDFKNHVI KYLETLLYSQ QQLAKYWEAF LPEAKAIS UniProtKB: Sorting nexin-1 |
-分子 #2: Sorting nexin-5
| 分子 | 名称: Sorting nexin-5 / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 46.891504 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MAAVPELLQQ QEEDRSKLRS VSVDLNVDPS LQIDIPDALS ERDKVKFTVH TKTTLPTFQS PEFSVTRQHE DFVWLHDTLI ETTDYAGLI IPPAPTKPDF DGPREKMQKL GEGEGSMTKE EFAKMKQELE AEYLAVFKKT VSSHEVFLQR LSSHPVLSKD R NFHVFLEY ...文字列: MAAVPELLQQ QEEDRSKLRS VSVDLNVDPS LQIDIPDALS ERDKVKFTVH TKTTLPTFQS PEFSVTRQHE DFVWLHDTLI ETTDYAGLI IPPAPTKPDF DGPREKMQKL GEGEGSMTKE EFAKMKQELE AEYLAVFKKT VSSHEVFLQR LSSHPVLSKD R NFHVFLEY DQDLSVRRKN TKEMFGGFFK SVVKSADEVL FTGVKEVDDF FEQEKNFLIN YYNRIKDSCV KADKMTRSHK NV ADDYIHT AACLHSLALE EPTVIKKYLL KVAELFEKLR KVEGRVSSDE DLKLTELLRY YMLNIEAAKD LLYRRTKALI DYE NSNKAL DKARLKSKDV KLAEAHQQEC CQKFEQLSES AKEELINFKR KRVAAFRKNL IEMSELEIKH ARNNVSLLQS CIDL FKNN UniProtKB: Sorting nexin-5 |
-分子 #3: Cation-independent mannose-6-phosphate receptor
| 分子 | 名称: Cation-independent mannose-6-phosphate receptor / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 3.766033 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: SNVSYKYSKV NKEEETDENE TEWLMEEIQL P UniProtKB: Cation-independent mannose-6-phosphate receptor |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | サブトモグラム平均法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 3.0 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 90 % / チャンバー内温度: 282 K / 装置: FEI VITROBOT MARK II / 詳細: Incubation time 30s Blotting time 2s. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / 撮影したグリッド数: 1 / 平均電子線量: 2.8 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 5.0 µm / 最小 デフォーカス(公称値): 2.0 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー








 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)