+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | African cichlid nackednavirus capsid at pH 7.5 | ||||||||||||
 マップデータ マップデータ | ACNDV capsid at pH 7.5 | ||||||||||||
 試料 試料 |
| ||||||||||||
 キーワード キーワード | T=3 / Capsid protein / VIRAL PROTEIN | ||||||||||||
| 機能・相同性 | C protein 機能・相同性情報 機能・相同性情報 | ||||||||||||
| 生物種 |  African cichlid nackednavirus (ウイルス) African cichlid nackednavirus (ウイルス) | ||||||||||||
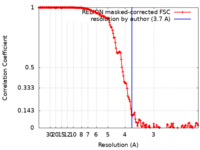
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.7 Å | ||||||||||||
 データ登録者 データ登録者 | Pfister S / Rabl J / Boehringer D / Meier BH | ||||||||||||
| 資金援助 | European Union,  スイス, 3件 スイス, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2023 ジャーナル: Nat Commun / 年: 2023タイトル: Structural conservation of HBV-like capsid proteins over hundreds of millions of years despite the shift from non-enveloped to enveloped life-style. 著者: Sara Pfister / Julius Rabl / Thomas Wiegand / Simone Mattei / Alexander A Malär / Lauriane Lecoq / Stefan Seitz / Ralf Bartenschlager / Anja Böckmann / Michael Nassal / Daniel Boehringer / Beat H Meier /    要旨: The discovery of nackednaviruses provided new insight into the evolutionary history of the hepatitis B virus (HBV): The common ancestor of HBV and nackednaviruses was non-enveloped and while HBV ...The discovery of nackednaviruses provided new insight into the evolutionary history of the hepatitis B virus (HBV): The common ancestor of HBV and nackednaviruses was non-enveloped and while HBV acquired an envelope during evolution, nackednaviruses remained non-enveloped. We report the capsid structure of the African cichlid nackednavirus (ACNDV), determined by cryo-EM at 3.7 Å resolution. This enables direct comparison with the known capsid structures of HBV and duck HBV, prototypic representatives of the mammalian and avian lineages of the enveloped Hepadnaviridae, respectively. The sequence identity with HBV is 24% and both the ACNDV capsid protein fold and the capsid architecture are very similar to those of the Hepadnaviridae and HBV in particular. Acquisition of the hepadnaviral envelope was thus not accompanied by a major change in capsid structure. Dynamic residues at the spike tip are tentatively assigned by solid-state NMR, while the C-terminal domain is invisible due to dynamics. Solid-state NMR characterization of the capsid structure reveals few conformational differences between the quasi-equivalent subunits of the ACNDV capsid and an overall higher capsid structural disorder compared to HBV. Despite these differences, the capsids of ACNDV and HBV are structurally highly similar despite the 400 million years since their separation. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_15295.map.gz emd_15295.map.gz | 45.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-15295-v30.xml emd-15295-v30.xml emd-15295.xml emd-15295.xml | 24.3 KB 24.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_15295_fsc.xml emd_15295_fsc.xml | 15.3 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_15295.png emd_15295.png | 245.8 KB | ||
| Filedesc metadata |  emd-15295.cif.gz emd-15295.cif.gz | 6.7 KB | ||
| その他 |  emd_15295_additional_1.map.gz emd_15295_additional_1.map.gz emd_15295_additional_2.map.gz emd_15295_additional_2.map.gz emd_15295_half_map_1.map.gz emd_15295_half_map_1.map.gz emd_15295_half_map_2.map.gz emd_15295_half_map_2.map.gz | 2.2 MB 753.7 KB 242.1 MB 242.1 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-15295 http://ftp.pdbj.org/pub/emdb/structures/EMD-15295 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-15295 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-15295 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_15295_validation.pdf.gz emd_15295_validation.pdf.gz | 860.7 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_15295_full_validation.pdf.gz emd_15295_full_validation.pdf.gz | 860.2 KB | 表示 | |
| XML形式データ |  emd_15295_validation.xml.gz emd_15295_validation.xml.gz | 23.5 KB | 表示 | |
| CIF形式データ |  emd_15295_validation.cif.gz emd_15295_validation.cif.gz | 30.6 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15295 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15295 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15295 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15295 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8aacMC  8c0oC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_15295.map.gz / 形式: CCP4 / 大きさ: 307.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_15295.map.gz / 形式: CCP4 / 大きさ: 307.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | ACNDV capsid at pH 7.5 | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.07 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: Map obtained by symmetry expansion
| ファイル | emd_15295_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Map obtained by symmetry expansion | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: Map obtained by symmetry expansion showing only the...
| ファイル | emd_15295_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Map obtained by symmetry expansion showing only the three chains of the asymmetric unit | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: ACNDV capsid at pH 7.5, unfiltered half map 2
| ファイル | emd_15295_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | ACNDV capsid at pH 7.5, unfiltered half map 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: ACNDV capsid at pH 7.5, unfiltered half map 1
| ファイル | emd_15295_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | ACNDV capsid at pH 7.5, unfiltered half map 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : African cichlid nackednavirus
| 全体 | 名称:  African cichlid nackednavirus (ウイルス) African cichlid nackednavirus (ウイルス) |
|---|---|
| 要素 |
|
-超分子 #1: African cichlid nackednavirus
| 超分子 | 名称: African cichlid nackednavirus / タイプ: virus / ID: 1 / 親要素: 0 / 含まれる分子: all / NCBI-ID: 2497433 / 生物種: African cichlid nackednavirus / ウイルスタイプ: VIRUS-LIKE PARTICLE / ウイルス・単離状態: OTHER / ウイルス・エンベロープ: No / ウイルス・中空状態: No |
|---|---|
| 宿主 | 生物種:  Ophthalmotilapia ventralis (魚類) Ophthalmotilapia ventralis (魚類) |
| 分子量 | 理論値: 3.57 MDa |
| ウイルス殻 | Shell ID: 1 / 名称: capsid / 直径: 230.0 Å / T番号(三角分割数): 3 |
-分子 #1: C protein
| 分子 | 名称: C protein / タイプ: protein_or_peptide / ID: 1 / コピー数: 180 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  African cichlid nackednavirus (ウイルス) African cichlid nackednavirus (ウイルス) |
| 分子量 | 理論値: 19.851234 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MGTFIELVKN MKGYKELLLP MEMVPLPAVV LKHVKLILTS QKEHQPWMTE MALKADQCLI HKATLDLAGK ATSNEAKPLI EAMQQIILA MTRELWGQIQ RHHYGIVQVE HYVKQITLWQ DTPQAFRGDQ PKPPSFRSDG PTRGQGSFRP FFRGRGRGRG R GRGSQSPA RKGPLPK UniProtKB: C protein |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 構成要素:
| |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| グリッド | モデル: Quantifoil / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: CONTINUOUS / 支持フィルム - Film thickness: 1 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 15 sec. / 詳細: 15 mA | |||||||||||||||
| 凍結 | 凍結剤: ETHANE-PROPANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV | |||||||||||||||
| 詳細 | The protein concentration is approx. 0.1-0.5 mg/mL |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K2 QUANTUM (4k x 4k) 検出モード: COUNTING / デジタル化 - 画像ごとのフレーム数: 1-40 / 実像数: 8999 / 平均電子線量: 76.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 100.0 µm / 最大 デフォーカス(補正後): 2.0 µm / 最小 デフォーカス(補正後): 0.5 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 0.5 µm / 倍率(公称値): 130000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 詳細 | One protein chain was manually built in Coot. The chain was multiplied to give 180 chains, which were arranged as an icosahedral capsid. The capsid was refined with phenix and Isolde. |
|---|---|
| 精密化 | 空間: REAL / プロトコル: AB INITIO MODEL |
| 得られたモデル |  PDB-8aac: |
 ムービー
ムービー コントローラー
コントローラー





 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)