+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
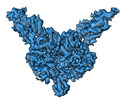
| タイトル | Cryo-EM density map of Tn4430 TnpA hyperactive mutant (TnpA3X) in complex with IR48 substrate. | |||||||||
 マップデータ マップデータ | final auto-sharpened map from Non-Uniform refinement in cryoSPARC v3.3.2 220518 | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Tranposition / complex / hyperactive mutant / paired-end complex / DNA BINDING PROTEIN | |||||||||
| 生物種 |  | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 6.0 Å | |||||||||
 データ登録者 データ登録者 | Shkumatov AV / Liu Y / Efremov RG | |||||||||
| 資金援助 |  ベルギー, 1件 ベルギー, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nucleic Acids Res / 年: 2023 ジャーナル: Nucleic Acids Res / 年: 2023タイトル: AFM-based force spectroscopy unravels stepwise formation of the DNA transposition complex in the widespread Tn3 family mobile genetic elements. 著者: Maricruz Fernandez / Alexander V Shkumatov / Yun Liu / Claire Stulemeijer / Sylvie Derclaye / Rouslan G Efremov / Bernard Hallet / David Alsteens /  要旨: Transposon Tn4430 belongs to a widespread family of bacterial transposons, the Tn3 family, which plays a prevalent role in the dissemination of antibiotic resistance among pathogens. Despite recent ...Transposon Tn4430 belongs to a widespread family of bacterial transposons, the Tn3 family, which plays a prevalent role in the dissemination of antibiotic resistance among pathogens. Despite recent data on the structural architecture of the transposition complex, the molecular mechanisms underlying the replicative transposition of these elements are still poorly understood. Here, we use force-distance curve-based atomic force microscopy to probe the binding of the TnpA transposase of Tn4430 to DNA molecules containing one or two transposon ends and to extract the thermodynamic and kinetic parameters of transposition complex assembly. Comparing wild-type TnpA with previously isolated deregulated TnpA mutants supports a stepwise pathway for transposition complex formation and activation during which TnpA first binds as a dimer to a single transposon end and then undergoes a structural transition that enables it to bind the second end cooperatively and to become activated for transposition catalysis, the latter step occurring at a much faster rate for the TnpA mutants. Our study thus provides an unprecedented approach to probe the dynamic of a complex DNA processing machinery at the single-particle level. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_15213.map.gz emd_15213.map.gz | 56.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-15213-v30.xml emd-15213-v30.xml emd-15213.xml emd-15213.xml | 20 KB 20 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
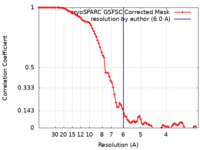
| FSC (解像度算出) |  emd_15213_fsc.xml emd_15213_fsc.xml | 8.3 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_15213.png emd_15213.png | 116.7 KB | ||
| マスクデータ |  emd_15213_msk_1.map emd_15213_msk_1.map | 59.6 MB |  マスクマップ マスクマップ | |
| その他 |  emd_15213_half_map_1.map.gz emd_15213_half_map_1.map.gz emd_15213_half_map_2.map.gz emd_15213_half_map_2.map.gz | 55.2 MB 55.2 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-15213 http://ftp.pdbj.org/pub/emdb/structures/EMD-15213 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-15213 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-15213 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_15213.map.gz / 形式: CCP4 / 大きさ: 59.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_15213.map.gz / 形式: CCP4 / 大きさ: 59.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | final auto-sharpened map from Non-Uniform refinement in cryoSPARC v3.3.2 220518 | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.568 Å | ||||||||||||||||||||||||||||||||||||
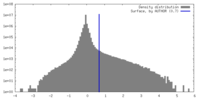
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_15213_msk_1.map emd_15213_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
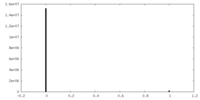
| 密度ヒストグラム |
-ハーフマップ: half map A
| ファイル | emd_15213_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map A | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
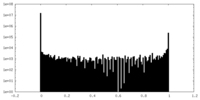
| 密度ヒストグラム |
-ハーフマップ: half map B
| ファイル | emd_15213_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map B | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
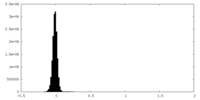
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : TnpA3X-IR48 complex
| 全体 | 名称: TnpA3X-IR48 complex |
|---|---|
| 要素 |
|
-超分子 #1: TnpA3X-IR48 complex
| 超分子 | 名称: TnpA3X-IR48 complex / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all 詳細: hyperactive TnpA mutant (W24R, A174V, E740G) in complex with 48 bp DNA containing transposon recognition sequence. |
|---|
-超分子 #2: TnpA 3X mutant
| 超分子 | 名称: TnpA 3X mutant / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1 / 詳細: hyperactive TnpA mutant (W24R, A174V, E740G) |
|---|---|
| 由来(天然) | 生物種:  |
-超分子 #3: IR48 substrate
| 超分子 | 名称: IR48 substrate / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #2-#3 / 詳細: DNA substrate |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: Transposase for transposon Tn4430
| 分子 | 名称: Transposase for transposon Tn4430 / タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MGVKQLLSEA QRNELMDLSR LTERDLVTFH TFSKHDLHLI LKHRRGYNRL GFALQLVLIR YPGWSLTEYK DIPQYVVAYV ASQLQIPPE EFLVYAKRGN TLWEHLGEIR TEYGYQNFSS EYKETLLQFL VQQAMDNNNT LYLIEITIST LRKMKVILPA M YVIEDIVW ...文字列: MGVKQLLSEA QRNELMDLSR LTERDLVTFH TFSKHDLHLI LKHRRGYNRL GFALQLVLIR YPGWSLTEYK DIPQYVVAYV ASQLQIPPE EFLVYAKRGN TLWEHLGEIR TEYGYQNFSS EYKETLLQFL VQQAMDNNNT LYLIEITIST LRKMKVILPA M YVIEDIVW EAKQQVDQKV YSILHDGLVQ EQKDQLDALL LPTINGKSPL AWLKDVPAQP SPESFLKVID RLQFVQKIGL TI DTTKINT NRLRQLARLG SKYEPYAFRR FNEVKRYSML VSFLLEITQD LIDYAIEIHD RLMMNLQTKG KKEQDEIQQA NGK KLNEKI LQFITVCGTL IEAKETGKDA FAALDEVMSW NEMVESVEEA KQLSRPLNYD YLDLLNTRYS YVRRYAPTLL RSLH FRATK SGEPVLQALD TIHELNETGK RKVPHGAPLH FVSNRWQKHV YDDDGNINRH YYELAALTEL RNHIRSGDIF VSGSR HHKA FDDYLIPYDE WNEVSNIPNG LTAPLKAEDY ITDRINRLNE HLEWLSKNSE KLEGVDISQG KLHVERLDRG TPEEAK AFS KLLHSMLPRI KLTDLLIEVA SWTGFHDQFI HASTNQSPDQ EEQNIVLATL MAMGTNIGLT KMAEATPGIS YRQMANA SQ WRMYDDAMVR AQSILVNFQK EQKLSSYWGD GTTSSSDGMR LSIAVRSLHA DSNPHYGTGK GGTIYRFVSD QLSAYHVK V ITTNARDALH VLDGLLHHGT DLKIEEHYTD TAGYTDQVFA LTHLLGFRFA PRIRDLADTK LFSIPGGEEY ENVQALLKG KINVKLIKEN YEDIRRLAYS VQTGKVSSAL IMGKLGSYAR QNKLATALGE MGRIEKTLFT LDYISNKAVR RRVQKGLNKG EAINALARI IFFGQRGEFR ERALQDQLQR ASALNIIINA ISVWNTVYME KAVEELKARG EFREDLMPYA WPLGWEHINF L GEYKFEGL HDTGQMNLRP LRIKEPFYSP IRSFLEQKLI SEEDLNSAVD HHHHHH |
-分子 #2: IR48 DNA substrate, non transferred strand
| 分子 | 名称: IR48 DNA substrate, non transferred strand / タイプ: dna / ID: 2 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種:  |
| 配列 | 文字列: (DC)(DC)(DA)(DT)(DG)(DG)(DG)(DG)(DG)(DT) (DA)(DC)(DC)(DG)(DC)(DC)(DA)(DG)(DC)(DA) (DT)(DT)(DT)(DC)(DG)(DG)(DA)(DA)(DA) (DA)(DA)(DA)(DA)(DC)(DC)(DA)(DC)(DG)(DC) (DT) (DA)(DA)(DG)(DA)(DT)(DC)(DC)(DT) |
-分子 #3: IR48 transferred strand
| 分子 | 名称: IR48 transferred strand / タイプ: dna / ID: 3 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種:  |
| 配列 | 文字列: (DA)(DG)(DG)(DA)(DT)(DC)(DT)(DT)(DA)(DG) (DC)(DG)(DT)(DG)(DG)(DT)(DT)(DT)(DT)(DT) (DT)(DT)(DC)(DC)(DG)(DA)(DA)(DA)(DT) (DG)(DC)(DT)(DG)(DG)(DC)(DG)(DG)(DT)(DA) (DC) (DC)(DC)(DC)(DC)(DA)(DT)(DG)(DG) |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.7 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 / 詳細: 50 mM HEPES (pH 7.5), 100 mM NaCl, 30 mM L-Arg HCL |
| グリッド | モデル: UltrAuFoil R2/2 / 材質: GOLD / メッシュ: 300 / 支持フィルム - 材質: GOLD / 支持フィルム - トポロジー: HOLEY ARRAY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 60 sec. / 前処理 - 雰囲気: OTHER / 前処理 - 気圧: 0.035 kPa |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 90 % / 装置: GATAN CRYOPLUNGE 3 |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | JEOL CRYO ARM 300 |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: In-column Omega Filter エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 撮影したグリッド数: 1 / 実像数: 6734 / 平均露光時間: 3.0 sec. / 平均電子線量: 64.6 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 4.0 µm / 最小 デフォーカス(公称値): 0.8 µm |
| 試料ステージ | 試料ホルダーモデル: JEOL CRYOSPECPORTER / ホルダー冷却材: NITROGEN |
 ムービー
ムービー コントローラー
コントローラー




 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)