+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Oligomeric structure of SynDLP | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | BDLP / cyanobacteria / membrane remodeling / LIPID BINDING PROTEIN | |||||||||
| 機能・相同性 | Mitofusin family / Dynamin, N-terminal / Dynamin family / GTPase activity / GTP binding / P-loop containing nucleoside triphosphate hydrolase / membrane / Slr0869 protein 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  | |||||||||
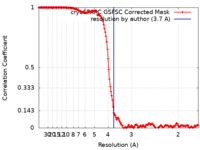
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.7 Å | |||||||||
 データ登録者 データ登録者 | Gewehr L / Junglas B / Jilly R / Franz J / Wenyu EZ / Weidner T / Bonn M / Sachse C / Schneider D | |||||||||
| 資金援助 | European Union, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2023 ジャーナル: Nat Commun / 年: 2023タイトル: SynDLP is a dynamin-like protein of Synechocystis sp. PCC 6803 with eukaryotic features. 著者: Lucas Gewehr / Benedikt Junglas / Ruven Jilly / Johannes Franz / Wenyu Eva Zhu / Tobias Weidner / Mischa Bonn / Carsten Sachse / Dirk Schneider /   要旨: Dynamin-like proteins are membrane remodeling GTPases with well-understood functions in eukaryotic cells. However, bacterial dynamin-like proteins are still poorly investigated. SynDLP, the dynamin- ...Dynamin-like proteins are membrane remodeling GTPases with well-understood functions in eukaryotic cells. However, bacterial dynamin-like proteins are still poorly investigated. SynDLP, the dynamin-like protein of the cyanobacterium Synechocystis sp. PCC 6803, forms ordered oligomers in solution. The 3.7 Å resolution cryo-EM structure of SynDLP oligomers reveals the presence of oligomeric stalk interfaces typical for eukaryotic dynamin-like proteins. The bundle signaling element domain shows distinct features, such as an intramolecular disulfide bridge that affects the GTPase activity, or an expanded intermolecular interface with the GTPase domain. In addition to typical GD-GD contacts, such atypical GTPase domain interfaces might be a GTPase activity regulating tool in oligomerized SynDLP. Furthermore, we show that SynDLP interacts with and intercalates into membranes containing negatively charged thylakoid membrane lipids independent of nucleotides. The structural characteristics of SynDLP oligomers suggest it to be the closest known bacterial ancestor of eukaryotic dynamin. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_14993.map.gz emd_14993.map.gz | 171.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-14993-v30.xml emd-14993-v30.xml emd-14993.xml emd-14993.xml | 17.3 KB 17.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_14993_fsc.xml emd_14993_fsc.xml | 14.9 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_14993.png emd_14993.png | 45.7 KB | ||
| マスクデータ |  emd_14993_msk_1.map emd_14993_msk_1.map | 347.6 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-14993.cif.gz emd-14993.cif.gz | 6.3 KB | ||
| その他 |  emd_14993_half_map_1.map.gz emd_14993_half_map_1.map.gz emd_14993_half_map_2.map.gz emd_14993_half_map_2.map.gz | 317.7 MB 317.8 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-14993 http://ftp.pdbj.org/pub/emdb/structures/EMD-14993 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-14993 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-14993 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_14993_validation.pdf.gz emd_14993_validation.pdf.gz | 743.1 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_14993_full_validation.pdf.gz emd_14993_full_validation.pdf.gz | 742.7 KB | 表示 | |
| XML形式データ |  emd_14993_validation.xml.gz emd_14993_validation.xml.gz | 24.1 KB | 表示 | |
| CIF形式データ |  emd_14993_validation.cif.gz emd_14993_validation.cif.gz | 31.5 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14993 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14993 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14993 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14993 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7zw6MC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_14993.map.gz / 形式: CCP4 / 大きさ: 347.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_14993.map.gz / 形式: CCP4 / 大きさ: 347.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.869 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_14993_msk_1.map emd_14993_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_14993_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_14993_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : filamentous homo-oligomer of SynDLP
| 全体 | 名称: filamentous homo-oligomer of SynDLP |
|---|---|
| 要素 |
|
-超分子 #1: filamentous homo-oligomer of SynDLP
| 超分子 | 名称: filamentous homo-oligomer of SynDLP / タイプ: cell / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: Slr0869 protein
| 分子 | 名称: Slr0869 protein / タイプ: protein_or_peptide / ID: 1 / コピー数: 8 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 93.705398 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MSKIAPQCQN LREQVNQLIE LLRQEPTLRS QQDTSIVETA LGKALSPRFE IVFAGAFSAG KSMLINALLE RELLYSAEGH ATGTECHIE YANANEERVV LTFLSEAEIR QQALILAKYL NVNVGDLNIN QPEAVKVVSQ YCQKIIAEEG GENKSERAKQ A NALHLLLI ...文字列: MSKIAPQCQN LREQVNQLIE LLRQEPTLRS QQDTSIVETA LGKALSPRFE IVFAGAFSAG KSMLINALLE RELLYSAEGH ATGTECHIE YANANEERVV LTFLSEAEIR QQALILAKYL NVNVGDLNIN QPEAVKVVSQ YCQKIIAEEG GENKSERAKQ A NALHLLLI GFEQNRERIN TVQNSTYSMD QLNFSSLAEA AGYARRGANS AVLKRLDYFC NHSLLKDGNV LVDLPGIDAP VK EDAERAY RKIESPDTSA VICVLKPAAA GDMSAEETQL LERISKNHGI RDRVFYVFNR IDDTWYNTQL RQRLEGLIQS QFR DNSRVY KTSGLLGFYG SQVKQTNSST RFGLDSIFAT TIKGFDGEEE TPQFVSEFNN YCANSGKLLS TAFRVSVNGY ETSN ENYVR ILSEWGIPLV DQLIHDSGIE SFRSGIGLYL AEEKYPELFA TLANDLQPLC IALRQFYLEN YRQLDSQPRE IAAMK AQEL TLLNQEMQNL GIEFKKYMSA QINDVVIGND REFDQDFTKL KARMVARLDE LLKTFSVMNA YKRATESHPR NSTAPF IAV LVEALYYLAN ELEDAFIEAI HELVKNFFQR LGDRLRKVDC YHQVYRLVGN DGGIEQLLRR AEEDITKALV NEARTEC DR YVRESPRFYD EGTFSIYQFR QTLQQTSQGY DAQAIVEAEP AIKELLKLDF EPKVFNTVRK NFRQTVNNTL KTHLLPMA E EQAQIILEQY DVARKYREQT LEQDAEEKIA RNSRLQSEIK QKIDLYQTSI VSINECLKAM QIFEQLPVIT ESDITKQAE IVADADFVEI VELEHHHHHH UniProtKB: Slr0869 protein |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 3.0 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
| ||||||||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 200 / 前処理 - タイプ: GLOW DISCHARGE | ||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 90 % / チャンバー内温度: 293 K / 装置: FEI VITROBOT MARK IV / 詳細: Blotting force -10 Blotting time 3 s. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TALOS ARCTICA |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 撮影したグリッド数: 1 / 実像数: 8322 / 平均露光時間: 2.0 sec. / 平均電子線量: 26.5 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 4.0 µm / 最小 デフォーカス(公称値): 2.0 µm / 倍率(公称値): 49000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Talos Arctica / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: AB INITIO MODEL |
|---|---|
| 得られたモデル |  PDB-7zw6: |
 ムービー
ムービー コントローラー
コントローラー




 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)